INTRODUCCIÓN
La interpretación típica de un estudio ecocardiográfico completo consiste en la revisión de múltiples grupos y tipos de imágenes, sea cual sea el orden en que hayan sido adquiridas. El ecocardiografista debe clasificar e integrar la información contenida en todas estas imágenes, e interpretarlas para reportar características de la anatomía y/o la función cardiovascular. Por lo tanto, no resulta infrecuente tener que realizar múltiples revisiones de un gran número de imágenes, lo cual puede tornarse un proceso largo y tedioso para el profesional. Además, una vez que las imágenes relevantes son identificadas, deben realizarse un número significativo de mediciones ecocardiográficas siguiendo las recomendaciones de las principales guías de sociedades internacionales. Es importante considerar que dichas mediciones varían significativamente entre distintos lectores. 1), (2), (3), (4), (5), (6), (7), (8 Aunque es difícil determinar con precisión el tiempo exacto que consume este proceso de selección, categorización y cuantificación durante un estudio ecocardiográfico, la posibilidad de acortar la duración de dichas etapas resulta una propuesta sumamente atractiva para optimizar todo el proceso y flujo de trabajo involucrado.
Estudios recientes han demostrado que tanto la clasificación de imágenes como las mediciones ecocardiográficas pueden ser automatizadas mediante la utilización de técnicas de machine learning (ML). 9), (10), (11 Nuestra hipótesis es que mediante estas nuevas herramientas podría optimizarse todo el proceso de interpretación clínica de los estudios ecocardiográficos y completarlo en una forma más rápida y reproducible que las técnicas manuales convencionales, contribuyendo finalmente a una mayor eficiencia y mejoría en el flujo de trabajo del servicio médico en cuestión. Este nuevo abordaje implicaría la incorporación de: (a) identificación automatizada del tipo y vista de la imagen ecocardiográfica, (b) clasificación y agrupación de las imágenes en categorías o grupos (stacks) definidos por estructuras anatómicas predeterminadas, (c) mediciones completamente automatizadas de parámetros estándar de cada estructura, y (d) corrección manual de dichas mediciones automatizadas (en caso de ser considerado necesario) para mejorar su precisión.
Nuestro objetivo fue analizar la factibilidad de este nuevo paradigma de trabajo a través de la evaluación de sus efectos en la eficiencia y reproducibilidad en las interpretaciones humanas.
MATERIAL Y MÉTODOS
Imágenes y población
Utilizamos diferentes estudios ecocardiográficos con el objetivo de intentar representar un espectro amplio de distintos grados de zoom, profundidad, ancho de zona de interés, foco, ganancia y calidad de imagen. Dichos estudios fueron realizados en 2000 individuos sanos, previamente incluidos en el estudio WASE (World Alliance Societies of Echocardiography Normal Values Study). El objetivo de dicho estudio fue determinar valores normales de múltiples parámetros ecocardiográficos cuantificables. 12 Los individuos incluidos, provenientes de 19 centros en 15 países, comprendieron un amplio grupo de adultos de ambos sexos, incluyendo diversas razas, etnias y grupos etarios. Dichos estudios fueron obtenidos utilizando distintos sistemas ecocardiográficos comercialmente disponibles y siguiendo las recientes recomendaciones de la Sociedad Americana de Ecocardiografía (American Society of Echocardiography, ASE). 13 Las imágenes fueron categorizadas por un experto según el tipo de imagen (2D, Doppler pulsado, etc.) y vista ecocardiográfica (apical 4 cámaras, paraesternal en eje largo, etc.). Mediciones habituales fueron realizadas por un experto en ecocardiografía en forma centralizada (core laboratory) siguiendo en forma estricta las más recientes recomendaciones. 14 Para nuestro estudio, utilizamos imágenes pertenecientes a 1800 individuos para el desarrollo y entrenamiento de los algoritmos de ML (grupo entrenamiento), mientras que los 200 restantes fueron utilizados para la evaluación de dichos algoritmos (grupo prueba).
Diseño del estudio
El estudio se dividió en 3 protocolos. El Protocolo 1 fue diseñado con el objeto de desarrollar los algoritmos de ML y evaluar su precisión en la identificación automatizada del tipo de imagen y vista ecocardiográfica, similar al abordaje utilizado en estudios recientes, 9), (10), (11 así como también en la clasificación de dichas imágenes en stacks de acuerdo con ciertas estructuras anatómicas o “eventos fisiológicos” de referencia (p.ej. válvula mitral, función diastólica), que servirán eventualmente como guía en la revisión e interpretación de las imágenes. El Protocolo 2 fue diseñado para desarrollar y evaluar la precisión de ML con el objeto de medir en forma automática parámetros ecocardiográficos convencionales, similar a los utilizados por Zhang et al. 10 Finalmente, el Protocolo 3 fue diseñado para determinar la factibilidad y efectividad resultante de la combinación e integración de dichos protocolos en la lectura de ecocardiogramas, con evaluación de parámetros de eficiencia (ahorro de tiempo) y reproducibilidad (variabilidad de las mediciones), y la comparación de dichos resultados con los métodos actuales de interpretación y medición.
La evaluación del Protocolo 1 consistió en el desarrollo y entrenamiento de una red neuronal convolucional (RNC) a partir de las mediciones provistas por un lector independiente (TM) no familiarizado con el desarrollo del algoritmo de ML. La evaluación del Protocolo 2 fue realizada mediante la comparación de las mediciones obtenidas por el algoritmo de ML con aquellas provistas por un segundo lector (KA) perteneciente a una institución diferente a la del primer lector. Finalmente, en el Protocolo 3 comparamos la performance de 2 lectores (KA y KK) que no participaron en la fase de desarrollo y entrenamiento del algoritmo de ML.
El desarrollo del software de ML, así como el diseño del estudio fueron producto de la colaboración entre los investigadores a cargo (MedStar y Universidad de Chicago) y TOMTEC Imaging Systems (Unterschleissheim, Germany). Los investigadores principales (RML, FMA) tuvieron acceso irrestricto a los datos y llevaron a cabo el análisis de estos en forma independiente de TOMTEC, asumiendo plena responsabilidad de los hallazgos aquí presentados.
Algoritmo de la red neuronal convolucional
El entrenamiento en la detección de cada parámetro de interés, se llevó a cabo a través de una estrategia en cascada que involucró 3 etapas de subdetectores. Esta configuración inicial fue acompañada por un mecanismo de preprocesamiento de las imágenes, con el objetivo de estandarizar la información obtenida de las mismas. Esto incluyó la detección del sector correspondiente al haz de ultrasonido, la eliminación de información adicional insertada en las imágenes ecocardiográficas y la adaptación de la intensidad y color de los archivos DICOM.
Inicialmente, para estimar aproximadamente la posición, tamaño, y orientación espacial de la estructura de interés, cada subdetector de la RNC fue alimentado en forma regresiva con una submuestra de imágenes con una resolución fija de menos del 20 % de número de pixeles de la imagen original. En una segunda etapa, a cada detector le fue presentada la región y estructura de interés de cada imagen con el objeto de reducir los cambios en escala y posición. La resolución de imagen para esta segunda etapa fue estandarizada a la resolución promedio (mm/píxel) de todas las imágenes, compensando la potencial variación de resolución de imágenes individuales. En esta instancia, la identificación de las estructuras de interés fue realizada con una RNC regresiva, al igual que en la primera etapa del proceso. Finalmente, en una tercera etapa utilizamos un grupo de imágenes más refinadas con mejor localización de las estructuras de interés (en comparación con la etapa anterior) a fin de mejorar su localización. Nuevamente, la implementación fue posible mediante una RNC regresiva.
El entrenamiento de cada etapa de subdetectores fue realizada utilizando un subgrupo de mediciones como la verdad absoluta (ground truth). Durante dicho entrenamiento, cada uno de estos detectores pudo analizar el input de información según estas mediciones asumidas como verdad absoluta, junto con 32 versiones aumentadas de dicho input. Dicho aumento fue posible mediante pequeñas rotaciones y cambios en escala de la región de interés para lograr un reconocimiento efectivo de un amplio número de variantes y estructuras.
Protocolo 1: Identificación y agrupamiento (stacking) automatizados de imágenes
La RNC fue entrenada para reconocer 18 vistas ecocardiográficas estándar: 6 correspondientes a ecocardiografía bidimensional (2D) y 12 correspondientes a Doppler tisular (DTI), Doppler pulsado (DP) y Doppler continuo (DC). Las imágenes 2D incluyeron las vistas apicales en 2, 3 y 4 cámaras (A2C, A3C y A4C respectivamente) como así las paraesternales en eje largo y eje corto (ELPE y ECPE respectivamente) y la vista subcostal (SC). Las imágenes de DTI incluyeron muestreos septal y lateral del anillo mitral y de la porción lateral del anillo tricúspideo. Las imágenes correspondientes a DP fueron obtenidas en el tracto de salida del ventrículo izquierdo (TSVI) en una vista apical, el tracto de entrada mitral en 4 cámaras y en tracto de salida del ventrículo derecho (TSVD) en una vista ECPE. Las adquisiciones de DC incluyeron la evaluación del flujo aórtico en una vista apical, como así también del flujo de regurgitación mitral en una vista apical de 4 cámaras, el de insuficiencia tricúspidea en un apical de 4 cámaras y ECPE, y el flujo pulmonar en una vista ECPE. Todas estas imágenes fueron utilizadas como input de la RNC para construir nuestro banco de tipos de imágenes y vistas ecocardiográficas.
Luego, la RNC fue entrenada para identificar las vistas y tipos de imagen en un grupo independiente de imágenes de prueba, para consiguientemente asignar o clasificar cada una de estas imágenes dentro de 8 stacks según la identificación de los siguientes parámetros o estructuras: función sistólica y tamaño ventricular izquierdo (VI), función diastólica del VI, función y tamaño del ventrículo derecho (VD) y aurícula derecha (AD), válvulas (mitral, aortica, tricúspide y pulmonar) y pericardio. La Tabla 1 muestra una lista de las imágenes necesarias para completar cada stack. Finalmente, los resultados de la identificación y clasificación automática de las imágenes fueron comparados con la “verdad absoluta” previamente determinados por el lector experto.
Protocolo 2: Precisión y reproducibilidad del análisis automatizado
Luego que cada imagen fuese automáticamente clasificada según tipo y vista de imagen y que la fase del ciclo cardíaco para realizar las mediciones fuera seleccionada, un algoritmo supervisado de ML fue entrenado para medir 16 parámetros estándar habitualmente cuantificados en la práctica clínica. Estos parámetros incluyeron: grosor del septum interventricular en fin de diástole, diámetro interno del VI en fin de diástole y fin de sístole, grosor de la pared posterior del VI en fin de diástole, diámetro mesosistólico del TSVI, integral velocidad-tiempo (VTI) del TSVI, máximo volumen de la aurícula izquierda (AI) en A2C y A4C, volúmenes del VI en fin de diástole y en fin de sístole, onda E y onda A del flujo de entrada mitral y las velocidades tisulares del anillo lateral y septal mitral. Todos los volúmenes de las cavidades fueron medidos mediante el método de discos de Simpson biplano.
La precisión de dichas mediciones automáticas fue comparada con la de las mediciones manuales obtenidas por un lector experto en el grupo de prueba de 200 sujetos. Además, en un subgrupo de 30 estudios seleccionados aleatoriamente, las diferencias entre las mediciones automáticas del ML y las proporcionadas por el lector experto de referencia, fueron comparadas con la variabilidad humana inter e intraobservador, a través de reiteradas mediciones a ciegas. Para evaluar la variabilidad interobservador, estas mediciones fueron realizadas independientemente por dos cardiólogos expertos en ecocardiografía, con el empleo de métodos convencionales de interpretación y medición. Finalmente, para determinar la variabilidad intraobservador, uno de estos expertos volvió a leer los mismos estudios ecocardiográficos al menos dos semanas después del primer análisis, nuevamente, completamente ciego a las mediciones previas.
Protocolo 3: Eficiencia y reproducibilidad del enfoque con asistencia del ML
En este protocolo, en forma independiente, ambos expertos repitieron la interpretación de estos 30 estudios al menos 2 semanas después, esta vez utilizando la asistencia de ML. En este caso, los expertos podían utilizar los stacks y mediciones determinadas automáticamente por los algoritmos, modificándolas por medio de los ajustes que ellos consideraran necesarios para mejorar la precisión en la identificación y medición de las estructuras o fenómenos de interés. Para ambas sesiones, el tiempo necesario para completar el análisis del estudio ecocardiográfico fue determinado por la estación de trabajo utilizada, y la variabilidad intra e interobservador fue evaluada para todos los parámetros previamente descriptos. Finalmente, los tiempos de análisis con y sin asistencia de ML fueron comparados entre sí.
Análisis estadístico
La precisión del método automático en la identificación del tipo de imagen y vista ecocardiografía y su comparación con los expertos fue cuantificada como el porcentaje de clasificaciones correctas en función del número total de secuencias de imágenes analizadas. Para cada parámetro, la concordancia entre las mediciones del ML y los valores de referencia obtenidos por los expertos fue evaluada mediante métodos de regresión linear y análisis de sesgos y límites de concordancia con el método de Bland-Altman. Para realizar comparaciones entre distintos parámetros, los sesgos fueron expresados como porcentajes del valor promedio de cada parámetro (%Sesgo). La variabilidad intra e interobservador para cada parámetro fue cuantificada utilizando coeficientes de variación, definidos como la diferencia absoluta entre mediciones repetidas y expresadas como porcentajes de la media. La significancia estadística de las diferencias en el tiempo de análisis con y sin asistencia del ML fue determinada utilizando la prueba t de Student utilizando mediciones pareadas a dos colas. Valores de p < 0,05 fueron considerados significativos.
RESULTADOS
Protocolo 1
La clasificación automática de las vistas ecocardiográficas por parte de la RNC se realizó en un promedio de 0,87 segundos por estudio. La RNC fue capaz de identificar los tipos de imagen y vistas ecocardiográficas con una precisión del 90 % para imágenes 2D y 94 % para imágenes Doppler. La Figura 1 muestra los datos correspondientes a la concordancia entre la clasificación obtenida por ML y por la “verdad absoluta” del lector experto, para cada vista ecocardiográfica. Si bien dicha concordancia fue excelente en la gran mayoría de los casos, el mayor número de errores ocurrió en la clasificación de vistas no estándar, subóptimas, erróneamente identificadas como A3C (anotadas como “Otras”). La RNC fue capaz de separar y agrupar las imágenes en stacks con una precisión del 91 %. Al contar el número de vistas requeridas para completar cada stack, ignorando aquellas consideradas innecesarias, la composición final de cada stack de imágenes tuvo una concordancia del 99 % entre ML y los lectores expertos (Tabla 1).
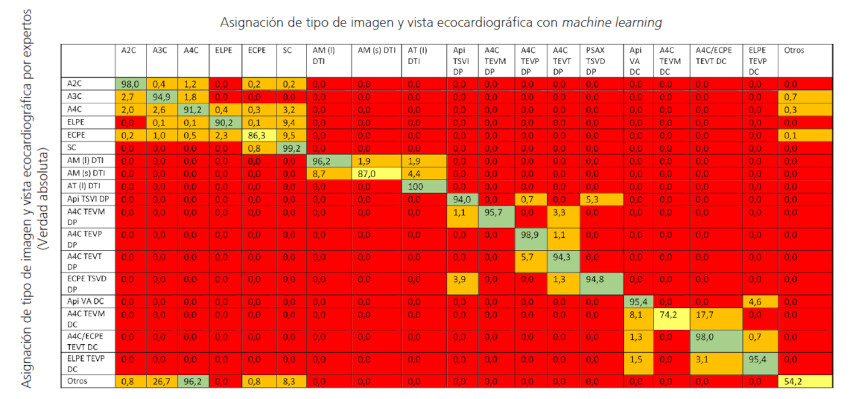
Fig. 1. Identificación del tipo y vista de imágenes ecocardiográficas: concordancia entre mediciones automatizadas con ML y mediciones manuales por expertos. Cada celda representa el porcentaje de concordancia para cada tipo de imagen y vista evaluados. Las celdas tienen distintos colores según el porcentaje de acuerdo: rojo ( 0%), naranja (0,1%-50 %), amarillo (50,1%-90%) y verde (90,1%-100%) Abreviaturas: A2C, A3C y A4C = apical 2, 3 y 4 cámaras, respectivamente; ELPE, ECPE, SC: paraesternal en eje largo, eje corto y subcostal, respectivamente; AM (l), AM (s), AT (l) = anillo mitral lateral, anillo mitral septal y anillo tricúspideo lateral, respectivamente; Api TSVI = vista apical del tracto de salida del ventrículo izquierdo; Api VA = vista apical de la válvula aortica; TEVM, TEVT, TEVP = tractos de entrada mitral, tricúspideo y pulmonar respectivamente; DC: Doppler continuo; DP: Doppler pulsado
Tabla 1 Composición de los grupos (stacks) de imágenes ecocardiográficas según el tópico o estructura de interés y porcentaje de clasificaciones automáticas completas mediante machine learning.
| Stack o grupo de interés | Tipo y vista de imagen ecocardiográfíca | %Stacks completos | |
|---|---|---|---|
| Vistas 2D | Doppler | ||
| Función sistólica y dimensiones del ventrículo izquierdo |
|
99,5 % | |
| *Función diastólica del ventrículo izquierdo |
|
|
100 % |
| Función y dimensiones del ventrículo y aurícula derechos |
|
99 % | |
| Válvula mitral |
|
|
99,8 % |
| Válvula aórtica |
|
|
96 % |
| Válvula tricúspide |
|
|
99,4 % |
| Válvula pulmonar |
|
|
98,4 % |
| Pericardio |
|
100 % | |
Protocolo 2
Las mediciones automáticas realizadas mediante ML mostraron una concordancia excelente respecto a los valores de referencia obtenidos por los expertos, demostrados por altos valores de índices de correlación, sesgos pequeños y límites de concordancia estrechos para la mayoría de los parámetros analizados (Tabla 2). Los sesgos relativos más grandes fueron para los volúmenes auriculares izquierdos tanto en A4C como A2C (18 % y 25 % de los valores de referencia), seguido por los volúmenes ventriculares izquierdos (6,5 % y 7,8 % de los valores de referencia). Para el resto de los parámetros evaluados, los sesgos relativos fueron mínimos, con valores menores al 3 %. El análisis del subgrupo de 30 estudios demostró que las diferencias entre las mediciones realizadas con ML y las obtenidas por los expertos fueron similares a las diferencias entre mediciones manuales realizadas por dos operadores humanos (Tabla 3).
Tabla 2 Evaluación del algoritmo de machine learning (ML)
| Regresión linear | Análisis de Bland Altman | ||||
|---|---|---|---|---|---|
| Coeficiente r de Pearson | Sesgo ± DE | % Sesgo | Límite de concordancia inferior | Límite de concordancia superior | |
| Grosor SIVd (mm) | 0,65 | -0,11 ± 1,3 | -1,5 | -2,7 | 2,4 |
| Grosor PPd (mm) | 0,64 | -0,04 ± 1,1 | -0,5 | -2,2 | 2,1 |
| DIVIs (mm) | 0,78 | 0,85 ± 2,5 | 3 | -4,2 | 5,9 |
| DIVId (mm) | 0,82 | 0,65 ± 3,1 | 1,5 | -5,6 | 6,9 |
| TSVI diam (mm) | 0,82 | 0,85 ± 1,5 | 4,1 | -2,1 | 3,8 |
| Vol VId (A2C) (ml) | 0,91 | 6,6 ± 12,5 | 6,5 | -18,4 | 31,6 |
| Vol VId (A4C) (ml) | 0,94 | 7,4 ± 9,5 | 7,8 | -11,5 | 26,3 |
| Vol VIs (A2C) (ml) | 0,87 | -0,5 ± 6,2 | -1,3 | -12,9 | 11,9 |
| Vol VIs (A4C) (ml) | 0,89 | 0,6 ± 5,6 | 1,6 | -10,7 | 11,8 |
| Vol AI (A2C) (ml) | 0,87 | 11,8 ± 10,3 | 25 | -8,9 | 32,5 |
| Vol AI (A4C) (ml) | 0,89 | 8,6 ± 8,5 | 18 | -8,4 | 25,6 |
| TSVI TVI (cm) | 0,91 | 0,46 ± 1,7 | 2,2 | -2,8 | 3,8 |
| E Vel (cm/s) | 0,96 | -0,01 ± 0,05 | -1,1 | -0,11 | 0,09 |
| A Vel (cm/s) | 0,95 | -0,01 ± 0,05 | -1,1 | -0,11 | 0,1 |
| E' vel (l) (cm/s) | 0,96 | -0,03 ± 1,3 | -0,2 | -2,62 | 2,56 |
| E' vel (s) (cm/s) | 0,96 | -0,03 ± 1,21 | -0,3 | -2,45 | 2,39 |
Concordancia entre mediciones y valores de referencia obtenidos en forma automática con ML y en forma manual por expertos en 200 estudios. Se incluye el coeficiente de regresión linear de Pearson (r) y el análisis Bland-Altman de los sesgos y límites de concordancia. Sesgos positivos representan sobreestimación por parte del ML, mientras que sesgos negativos representan subestimación
Abreviaturas: DE= desviación estándar; SIVd = septum interventricular en fin de diástole; PPd = pared posterior en fin de diástole; DIVIs= diámetro interno del ventrículo izquierdo en fin de sístole; DIVId= diámetro interno del ventrículo izquierdo en fin de diástole; TSVI diam= diámetro del tracto de salida del ventrículo izquierdo en mesosístole; Vol VId A2C y A4C= volúmenes de fin de diástole del ventrículo izquierdo en vista apical de 2 y 4 cámaras respectivamente; Vol VIs A2C y A4C= volúmenes de fin de sístole del ventrículo izquierdo en vista apical de 2 y 4 cámaras respectivamente; Vol AI A2C y A4C= volumen máximo de la aurícula izquierda en vista apical de 2 y 4 cámaras respectivamente; TSVI TVI= velocidad intergral tiempo en el tracto de salida del ventrículo izquierdo; E Vel y A Vel= velocidad de ondas temprana (E) y tardía (A) del flujo de entrada mitral, E' vel (l) y E' vel (s)= velocidad tisular a nivel anillo mitral lateral y septal, respectivamente
Tabla 3 Comparación entre valores de referencia obtenidas automáticamente con ML y medidas obtenidas por un lector experto utilizando métodos convencionales de medición, incluyendo variabilidad intra e interobservador de cada uno de estos métodos
| Interpretación convencional manual | Interpretación automática solo con ML vs Manual | Interpretación con ML + correcciones vs Manual | ||
|---|---|---|---|---|
| Variabilidad intraobservador | Variabilidad interobservador | |||
| Grosor SIVd (mm) | 7 ± 5 | 11 ± 8 | 14 ± 10 | 0 ± 1 * |
| Grosor PPd (mm) | 8 ± 7 | 15 ± 13 | 17 ± 15 | 1 ± 3 * |
| DIVIs (mm) | 3 ± 2 | 8 ± 6 | 10 ± 10 | 3 ± 5 * |
| DIVId (mm) | 2 ± 2 | 4 ± 4 | 6 ± 5 | 0 ± 1 * |
| TSVI diam (mm) | 2 ± 3 | 4 ± 3 | 5 ± 4 | 6 ± 14 |
| Vol VId (A2C) (ml) | 10 ± 9 | 20 ± 13 | 14 ± 10 | 6 ± 8 * |
| Vol VId (A4C) (ml) | 7 ± 5 | 22 ± 7 | 16 ± 8 | 4 ± 5 * |
| Vol VIs (A2C) (ml) | 11 ± 9 | 23 ± 14 | 27 ± 19 | 3 ± 4 * |
| Vol VIs (A4C) (ml) | 9 ± 7 | 32 ± 13 | 35 ± 16 | 4 ± 5 * |
| Vol AI (A2C) (ml) | 14 ± 9 | 17 ± 22 | 14 ± 0 | 9 ± 9 |
| Vol AI (A4C) (ml) | 13 ± 13 | 18 ± 13 | 16 ± 8 | 9 ± 8 * |
| TSVI TVI (cm) | 5 ± 4 | 7 ± 5 | 8 ± 7 | 1 ± 4 * |
| E Vel (cm/s) | 4 ± 4 | 8 ± 7 | 6 ± 5 | 3 ± 16 |
| A Vel (cm/s) | 3 ± 3 | 14 ± 11 | 14 ± 11 | 3 ± 16 * |
| E' vel (l) (cm/s) | 7 ± 9 | 10 ± 0 | 11 ± 7 | 2 ± 8 |
| E' vel (s) (cm/s) | 4 ± 4 | 6 ± 8 | 8 ± 7 | 0 ± 0 * |
Los valores representan coeficientes de variación (ver texto para más detalles). Abreviaturas similares a las presentadas en Tabla 2.
*p < 0,05 para interpretación automatizada con ML.
Protocolo 3
La interpretación convencional de un estudio ecocardiográfico completo requirió un promedio de 11 minutos y 33 segundos (9:29 para el lector #1 y 13:36 para el lector #2). En cambio, al utilizar la asistencia de ML mediante la agrupación en stacks y mediciones automatizadas, el tiempo promedio en completar la evaluación de un estudio fue de 6 minutos y 48 segundos (4:50 para el lector #1 y 8:45 para el lector #2). Es decir, que con el uso de ML es posible reducir en promedio un 40 % el tiempo necesario para interpretar un estudio ecocardiográfico utilizando los métodos convencionales actuales (49 % para el lector #1 y 36 % para el lector #2). Cabe destacar que, en comparación con el método manual, la utilización de ML redujo la variabilidad interobservador en 15 de las 16 mediciones realizadas (Tabla 3), a excepción del diámetro del TSVI, cuya medición, de todas maneras, fue altamente reproducible utilizando ambos métodos.
DISCUSIÓN
En la última década, la utilización de inteligencia artificial en el mundo de las imágenes médicas ha crecido notablemente gracias a su capacidad de asistir en diversas instancias el proceso de interpretación y diagnóstico. 15), (16 Si bien la utilidad de la inteligencia artificial ha sido evaluada principalmente a partir de imágenes estáticas pertenecientes al campo de la radiología, la aplicación de técnicas como el ML en la ecocardiografía ha permanecido relativamente rezagada. Esto podría explicarse, al menos parcialmente, por los desafíos técnicos propios de la ecocardiografía, como ser su menor resolución espacial o calidad de imagen en comparación con otras modalidades como la tomografía computada o la resonancia magnética nuclear, como así también por el aspecto dinámico de las imágenes ecocardiográficas (uso de videos), lo cual exige un análisis de grandes volúmenes de datos e información en un tiempo extremadamente corto. Sin embargo, estudios recientes han superado muchos de estos obstáculos mediante la utilización de nuevas técnicas de ML especialmente diseñadas para la identificación de tipos y vistas ecocardiográficas, 9), (10), (11 o para la medición automatizada de parámetros ecocardiográficos de uso habitual en la práctica clínica. 10), (17), (18 Así, por ejemplo, el uso de inteligencia artificial como guía para la adquisición de imágenes ecocardiográficas ha permitido a usuarios con mínimo entrenamiento en ultrasonido, obtener imágenes de calidad cercana a las de un operario experto. 19 A nuestro entender, nuestro estudio es el primero que ha logrado combinar estas nuevas técnicas de ML para la identificación y clasificación de imágenes de acuerdo con características comunes preestablecidas. Incluso, es el primero en demostrar el potencial impacto de la incorporación de esta nueva herramienta en términos de reducción de los tiempos necesarios para completar el análisis de un ecocardiograma, con la consecuente mejora en la eficiencia del flujo de trabajo dentro del laboratorio de ecocardiografía.
Es cierto que los cardiólogos en general y especialistas en ecocardiografía en particular, se encuentran habituados a identificar, clasificar y analizar grandes cantidades de imágenes, sin necesidad de un proceso previo de agrupación o stacking. Sin embargo, el número creciente de estudios y la necesidad de cumplir estrictamente las recomendaciones de las guías de cuantificación ecocardiográfica han dejado en evidencia una nueva realidad, donde la incorporación de mecanismos de automatización de estas tareas repetitivas resulta cada vez más necesaria. La tecnología basada en procesos de ML resulta especialmente adecuada para resolver este tipo de problemas, gracias a su habilidad única de identificar y clasificar imágenes, 9), (10), (11 e incluso identificar componentes o estructuras específicas dentro de dichas imágenes que requieran ser segmentados y/o medidos. 10 En este estudio, propusimos que la combinación de todas estas capacidades del ML puede contribuir a facilitar la interpretación diagnostica en múltiples instancias del proceso, desde su clasificación hasta la propia realización de las mediciones.
La hipótesis que motivó inicialmente la realización de este estudio fue que implementar una variedad de técnicas de ML podría significar un cambio disruptivo en la manera en que los estudios ecocardiográficos son actualmente leídos e interpretados. En última instancia, nuestro propósito consiste en optimizar el complejo y extenso proceso de interpretación de un estudio ecocardiográfico mediante un aumento en la eficiencia, precisión y reproducibilidad de las lecturas que las herramientas de ML pueden generar.
Al igual que en publicaciones previas, 9), (10), (11 el uso de RNC en nuestro estudio le permitió a una computadora identificar en forma rápida y precisa la gran mayoría de tipos y vistas posibles para una imagen ecocardiográfica. Según esta clasificación, las imágenes fueron organizadas y presentadas a los lectores expertos en stacks temáticos, útiles y prácticos a la hora de responder una determinada pregunta clínica. Es decir, esta herramienta permitió prácticamente eliminar la necesidad de identificar, clasificar y agrupar visualmente, diversas estructuras anatómicas, como así también prescindir (al menos virtualmente) de las mediciones humanas de parámetros ecocardiográficos de uso habitual. De hecho, nuestros resultados demostraron que las diferencias entre las mediciones obtenidas en forma automática y sin ajustes manuales y las obtenidas por un experto no fueron mayores que aquellas diferencias observadas entre dos lectores de igual experiencia y entrenamiento. Así, la asistencia de ML en la interpretación de las imágenes ecocardiográficas permitió reducir a menos de 10 % la variabilidad de las mediciones en la gran mayoría de los parámetros evaluados, lo cual es considerado óptimo para la valoración cualitativa de la función cardiovascular. Además, la combinación de las habilidades de identificación, stacking y medición automática (con corrección manual de ser necesario) de los algoritmos de ML, permitió a los lectores por un lado reducir notablemente su tiempo habitual de análisis, y por otro reducir la variabilidad interobservador en la cuantificación de la mayoría de los parámetros evaluados.
Una ventaja importante del software desarrollado y evaluado en este estudio es su teórica independencia del fabricante (vendor) de sistemas ecocardiográficos, ya que fue diseñado para interpretar imágenes en formato DICOM provenientes de cualquiera de estos, lo cual permitiría su utilización en cualquier laboratorio, independientemente del sistema ecocardiográfico que cada laboratorio o servicio utilicen.
Dentro de las limitaciones de nuestro estudio es menester remarcar que solamente se incluyeron individuos sanos, previamente analizados en el estudio WASE. Sin embargo, otros softwares de ML han sido previamente validados por diversos autores, y en pacientes con un amplio espectro de patologías cardiovasculares. 9), (10), (11), (20), (21 Por lo tanto, realizar nuevamente la validación de dicho software, no era parte de los objetivos de nuestro estudio, el cual estuvo exclusivamente dirigido a evaluar un circuito alternativo en la interpretación de imágenes ecocardiográficas. Así, resulta poco probable que la inclusión de pacientes con patología pueda modificar nuestros hallazgos.
En conclusión, las técnicas de ML tienen el potencial de mejorar significativamente la eficiencia y reproducibilidad de la interpretación de imágenes ecocardiográficas a través del uso de stacks temáticos según estructuras en común y a través de mediciones automatizadas potencialmente corregibles. La implementación de este tipo de tecnologías permitirá ahorrar tiempo y costos, fundamentalmente a partir de una mejora en la eficiencia de los procesos, una mayor satisfacción del equipo médico y un aumento del rendimiento diagnóstico.











 texto en
texto en