Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista veterinaria
On-line version ISSN 1669-6840
Rev. vet. vol.25 no.1 Corrientes June 2014
TRABAJOS DE INVESTIGACIÓN
Detección de leptospiras patógenas en tejido renal de murciélagos de Corrientes, Argentina
Ramirez, N.N.1; Alegre, E.A.1; Ruiz, R.M.1; De Biasio, M.B.2; Bastiani, C.E.
1Cátedra Salud Pública, 2Servicio de Biología Molecular, Facultad de Ciencias Veterinarias, Universidad Nacional del Nordeste (UNNE), Sargento Cabral 2139, Corrientes (3400), Argentina. Tel/fax 03794-425753. E-mail: natyramirez13@hotmail.com
Resumen
Existen muchos animales que participan en la transmisión de diferentes serovares de leptospiras. Los murciélagos han acrecentado su presencia en zonas urbanas; este cambio de hábitat se atribuye a razones de origen antropogénico y ha elevado las probabilidades de contacto entre quirópteros, seres humanos y animales domésticos. El objetivo del trabajo fue identificar, mediante una reacción en cadena de la polimerasa (PCR sencilla), la infección natural de especies de leptospiras patógenas en quirópteros colectados durante los años 2010 a 2012 en la ciudad de Corrientes. Para la captura se utilizaron redes de mano y trampas balde. La identificación de los quirópteros se efectuó en base a su características fenotípicas y morfométricas, determinándose la presencia de ejemplares de tres de las cuatro familias existentes en Argentina. Los animales fueron sacrificados bajo anestesia de hidrato de cloral 20%, obteniéndose muestras de tejido renal. Se realizó el procedimiento de extracción de ácido desoxirribonucleico; se amplificaron 2 genes en PCR sencillas separadas, un gen mitocondrial, mt DNA 12S/16S como control de especie y luego, en las mismas muestras, se amplificó parte de los genes LipL32. Se detectó presencia de ADN de leptospiras en el 20% de los quirópteros, siendo éste el primer hallazgo por PCR publicado para murciélagos del país. Revelar la presencia de ADN de leptospiras resulta de gran importancia, debido a su utilidad en los casos en que la búsqueda molecular del patógeno ha sido no detectable.
Palabras clave: Murciélago; Leptospira; Tejido renal; PCR; Epidemiología; Argentina.
Pathogenic leptospires in renal tissue of bats from Corrientes, Argentina.
Abstract
Many animal species participate in the transmission of different leptospire serovars, such as bats. A large number of bats in urban areas has been associated with habitat changes due to the effects of humans on the environment. This fact increases the probability of contact between human beings and/or pets. The search of pathogenic leptospire species is a fundamental step for understanding the disease for a considered region. Aim of this work was to identify natural infection with pathogenic leptospires in bats from Corrientes, Argentina, by means of polymerase chain reaction (simple PCR) during the period 2010-2012. Animals were collected using nets and traps. Identification of bats was performed taking into account phenotypic characteristics and morphological measures. Animals were euthanized with 20% chloral hydrate and kidney samples were collected for DNA extraction. Two genes were amplifed using simple PCR: mt DNA 12S/16S mitochondrial gene (as a control of species) and part of LipL32 genes. Analysis of bats´ morphology revealed the presence of three out of the four families present in Argentina. Presence of leptospire DNA was detected in 20% of bats, being this the first report for Argentina. The determination of leptospire DNA is a useful and accurate method in those cases where the molecular search of the pathogen was not detected.
Key words: Bat; Leptospire; Renal tissue; PCR; Epidemiology; Argentina.
Recibido: 17 marzo 2014
Aceptado: 25 abril 2014
INTRODUCCIÓN
La leptospirosis es una enfermedad zoonótica de distribución mundial 3, 13, presente con mayor frecuencia en países tropicales, causada por espiroquetas del género Leptospira, clasificadas en L. interrogans (patógenas) y L. biflexa (no patógenas) 4. La transmisión de diferentes serovares se realiza a través de roedores, zorros, lobos, mapaches y mofetas 16, alpacas y vicuñas 26, bovinos, ovinos, caprinos, porcinos y caninos 3, 17. Estos animales pueden participar en la cadena epidemiológica como reservorios, portadores u hospedadores, resultando importante dilucidar el papel de cada especie en la enfermedad 23.
En la Amazonia y el sudeste de Asia se han estudiado roedores, murciélagos y marsupiales que pueden ser infectados por leptospiras e intervenir en el ciclo de transmisión como portadores 3, 5, 17, 29. Otros estudios estiman que el 60% de los patógenos emergentes que afectan al hombre son zoonóticos y que de éstos, más del 70% tienen origen en la fauna silvestre 22.
Los primeros estudios para identificación de leptospiras se realizaron con técnicas directas, como la observación del microorganismo en campo oscuro. Posteriormente apareció la prueba de aglutinación microscópica (MAT) 15, hoy considerada como "prueba de oro" y utilizada únicamente para diagnóstico e investigación en centros de referencia. Las pruebas basadas en el diagnóstico molecular presentan mayor especificidad y sensibilidad en comparación con las pruebas serológicas 24.
La aplicación del diagnóstico molecular es muy útil cuando el material se encuentra contaminado, el aislamiento es difícil o las leptospiras no se encuentran viables; también es ventajosa en el caso de no disponer de muestras pareadas 6. La reacción en cadena de la polimerasa (PCR) es la más sensible y la que se aplica con mayor frecuencia en microbiología clínica, en particular en el caso de patógenos difíciles de cultivar in vitro o aquéllos de lento crecimiento 15. La lipoproteína de membrana externa más estudiada es la LipL32, altamente inmunogénica y más conservada entre las especies patógenas de leptospiras 10, 11, 14.
Es conocida la participación de los murciélagos en numerosas enfermedades. Su aumento en zonas urbanas se ha explicado por alteraciones antropogénicas de su hábitat, como cambios que facilitan la migración, o que alteran dormideros y alimentación. Asociados a la ausencia de depredadores, estos cambios han contribuido a que algunas especies de quirópteros vivan en diversos tipos de construcciones artificiales, entre ellas la vivienda del hombre, lo que produjo un aumento en la probabilidad de contacto de los murciélagos con seres humanos y animales domésticos 28.
Investigaciones desarrolladas en la Amazonia peruana, estimaron que un tercio de los mamíferos silvestres analizados por técnicas moleculares, pertenecientes a los órdenes Rodentia, Marsupialia, Chiroptera y Carnívora, podrían estar excretando leptospiras patógenas en la orina, convirtiéndose en una fuente de infección humana, debido a su cercanía al área urbana 7.
La búsqueda e identificación de especies patógenas es un paso básico para la comprensión de la epidemiología de la enfermedad en cualquier región geográfica. Dada la ausencia de datos en Argentina, el presente trabajo tuvo como objetivo identificar mediante PCR la infección por leptospiras patógenas de quirópteros colectados en la Ciudad de Corrientes (Argentina) durante los años 2010 a 2012.
MATERIAL Y MÉTODOS
Área de estudio y captura. El área seleccionada fue la jurisdicción de la Municipalidad de la Ciudad de Corrientes. Durante 2010 a 2012 se visitaron sitios considerados como posibles refugios. Para la colecta se utilizaron redes de mano (captura al vuelo), trampas balde y captura manual, según ubicación, tipo de refugios y horario. Las coordenadas de cada sitio visitado fueron establecidas mediante un localizador satelital GPS (sistema de posicionamiento global) Garmin eTrex LegendR.
Una vez capturados, los quirópteros fueron transportados en bolsas de paño al laboratorio. Para ser identificados se tuvieron en cuenta las características fenotípicas y morfométricas utilizando una balanza digital y un calibre digital Black Jack D056, siguiendo las claves de identificación tradicionales 1, 2. La edad (juvenil y adulto) se estimó considerando longitud y diámetro de los huesos largos y transparencia de los cartílagos 12. Atento a las normas de bienestar animal, los murciélagos fueron sacrificados con hidrato de cloral al 20%. Post mortem se tomaron muestras de tejido renal, conservándolas en freezer a -20°C hasta su utilización.
Extracción de ADN. Se realizó el procedimiento de extracción de ácido desoxirribonucleico (ADN) efectuándose digestión con detergente CTAB (bromuro de cetil trimetilamonio) y purifcación con cloroformo: alcohol isoamílico (microcentrífuga Spectrafuge 24D Labnet Internacional y baño termostático Faeta). La calidad y cantidad del ADN extraído se evaluó efectuando un screening por electroforesis en geles de agarosa teñidos con bromuro de etidio, buffer TBE 1X y transiluminación (transiluminador UV DyNA dual Intensity Po Box 841 Labnet Internacional).
PCR para control de especie. Se utilizó un par de partidores (primers) complementarios a una secuencia de un gen mitocondrial, mt DNA 12S/16S Fw Bat 5´- TGG GAT TAG ATA CCC CAC TAT – 3´ y 12S/16S Rew Bat 5´- TGA TTA TGC TAC CTT TGC ACG GT-3´ anteriormente descripto 19. Se amplificó en un volumen de 25 µl, cuyo producto es de 1500 pb. Se ensayaron reacciones conteniendo las siguientes concentraciones finales de reactivos: 1X de buffer de PCR (Promega), 2,5 mM MgCl2 (Biodynamics), 0,5mM de cada dNTP (Promega), 0,2 mM de cada primer y 1,0U de Taq DNA polimerasa (Go-Taq, Promega). El programa de ciclado (Termociclador Minigen TC 9600-G / TC 9600-G-230 V /TC 6000-G / TC 6000-G-230 V Lab net Internacional) consistió en desnaturalización inicial a 95ºC durante 5 minutos, seguida de 30 ciclos de amplificación (desnaturalización a 94ºC por 50 segundos, hibridación a 55ºC por 35 segundos y extensión a 72ºC por 2 minutos) y una extensión final a 72ºC por 10 minutos e incubación a 4ºC. Se utilizó agua destilada como control negativo. Los productos de PCR se separaron por electroforesis horizontal (cuba de electroforesis (DYCP-31 DNBA BioAmerica) y fuente de poder para electroforesis (modelo DYY-6 CBA BioAmerica) en geles de agarosa 1% en buffer TBE1X, teñidos con bromuro de etidio, utilizando un marcador de peso molecular (pGem) para la comparación de tamaños de los fragmentos amplificados y visualización por transiluminación UV.
PCR para identificación de leptospiras. Se amplificó parte de los genes LipL32, presente en las especies patógenas aplicando una técnica anteriormente descrita 18, con modificaciones realizadas por nuestro grupo de trabajo en la concentración de primers (0.2 mM).
RESULTADOS
La técnica de PCR sencilla fue aplicada a 70 de las 104 muestras obtenidas. Los análisis morfológicos y morfométricos de los murciélagos indicaron que los ejemplares pertenecían a 3 familias de las 4 existentes en nuestro país: Vespertilionidae, Molossidae y Phyllostomidae.
Los sitios de recolección fueron muy variados, aunque todos ellos estaban en estrecha contigüidad con seres humanos. Se capturaron murciélagos en hogares, centros de salud, escuelas, edificios y espacios libres, algunos de ellos próximos al Río Paraná, y otros en zonas urbanizadas.
Debido a que la extracción de ADN es un punto crítico de la técnica PCR 18, 25, para verificar la presencia de material genético de quirópteros se realizó la PCR control, que arrojó como resultado bandas de amplificación de 1500 pb y ausencia de la misma en el control negativo de PCR en todas las electroforesis realizadas, lo que indicó presencia de material genético.
En el diagnóstico de leptospiras, fue posible constatar presencia de ADN detectable en el 20% (14/70 casos) de las muestras analizadas (Figura 1), con una banda de amplificación de 474 pb, sin amplificación en los controles negativos, no observándose bandas inespecíficas. En tres familias diferentes de quirópteros de hábitos insectívoros, fueron detectadas leptospiras patógenas (Tabla 1).

Figura 1. Electroforesis en gel de agarosa 2% para PCR para leptospiras. Las calles 3, 5, 6, 9 y 15 correspondes a muestras de murciélagos detectables a leptospira. Calle 8 ("M"): marcador de peso molecular. Calle 16: control positivo. Calle 18 control negativo.
Tabla 1. Especies de murciélagos positivos a leptospiras patógenas por PCR.
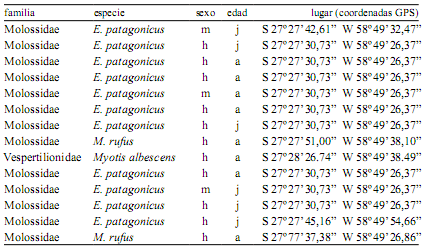
E: Eumops, M: Molossus, m: macho, h: hembra, j: juvenil, a: adulto, GPS: sistema de posicionamiento global.
DISCUSIÓN
En los estudios epidemiológicos, las modernas herramientas moleculares son indispensables como sistemas de tipificación. Por otra parte, aún no está completamente esclarecido el rol que desempeñan los mamíferos voladores en la cadena epidemiológica, conocimiento de vital importancia en salud pública.
Los resultados de este trabajo son los primeros publicados en Argentina con referencia al rol de los murciélagos en la leptospirosis. Desde el punto de vista diagnóstico es primordial establecer una técnica control de la presencia de ADN por su utilidad en los casos en que la búsqueda molecular del patógeno hubiere resultado negativa. En tales casos, al aplicar previamente la técnica de control de especie se confirma la presencia de ADN en la muestra y se descartan posibles errores en la extracción de ADN.
Con relación a los hábitos alimenticios de los murciélagos estudiados, la ausencia de ejemplares "pescadores" (Noctilionidae) se debería atribuir a las dificultades que involucra su captura. Si bien la muestra se integró con gran superioridad de especies insectívoras, esto no implica que otras especies no puedan ser portadoras. Llamativamente los ejemplares de murciélagos hematófagos capturados resultaron negativos a la prueba. Si la recolección hubiera ocurrido en zonas rurales probablemente el numero de especies hematófagas hubiera sido mayor.
Se asevera que los ejemplares del "vampiro" Desmodus rotundus, por su hábito de orinar durante la alimentación, eliminan orina que puede contaminar el ambiente y/o sus víctimas (animales o personas) con leptospiras. Las heridas causadas por la mordedura constituyen una puerta de entrada para dichos gérmenes. Murciélagos hematófagos (n = 204) capturados en tuberías de drenaje y túneles en Botucatú (Brasil) revelaron una prevalencia de de leptospiras del 7,84% 29.
En Perú se aislaron leptospiras al partir de riñones de microquirópteros 7, identificándose L. interrogans serovar icterohaemorrhagie, variedad que se mantiene entre las ratas, lo cual sugiere un ciclo de infección entre estas dos especies 20. A partir del riñón de una comadreja se aisló Leptospira interrogans canicola, atestiguando que para la transmisión solo basta el contacto directo con orina que contenga leptospiras 7.
En Australia se detectaron anticuerpos circulantes anti leptospiras en 173 murciélagos frugívoros Pteropus spp., aislándose Leptospira sp. en el 11% de las muestras renales 9, 27, señalando que los murciélagos que se alimentan con frutas pueden ser portadores de este patógeno. El hallazgo de uno de cada diez animales con ADN de leptospira sugiere que en esa población de murciélagos tales gérmenes son más frecuentes de lo esperado para una especie que no es portadora 29.
El gremio trófico capturado en mayor medida perteneció a los insectívoros, debido a que algunas especies están adaptadas a la ciudad y a las viviendas. La mayor concentración de mosquitos atraídos por el alumbrado de las calles constituye una gran fuente de alimentación. Trabajos realizados en Brasil evidenciaron un 2,8% de leptospirosis en las especies Molossus molossus y Molossus ater, demostrando el alto grado de adaptación de estas especies al entorno urbano, así como el peligro de la potencial exposición de las personas con su orina infectante. Otras especies de murciélagos adaptados a los entornos humanos son Glossophaga soricina (nectarívoro) y Prochilodus lineatus (frugívoro), este ultimo arrojó 1% de positividad a leptospirosis. Además de su potencialidad directa para infectar a las personas, estas especies son peligrosas por su posibilidad de interactuar con otros reservorios de leptospiras como las ratas 28.
Mediante técnicas de biología molecular, en la Amazonia peruana se demostró que los géneros Promops (insectívoro), Desmodus (hematófago) y Myotis (insectívoro) revelaron positividad a leptospiras mediante prueba PCR con tasas de 100%, 50% y 33% respectivamente, sobre un total de 204 murciélagos capturados. Cabe destacar que al aplicar la técnica MAT en estos mismos animales, los resultados fueron negativos, revelando que a pesar de ser la "técnica de oro", su aplicación no siempre es adecuada pues depende del tipo de estudio-clínico o epidemiológico- y no es aplicable a los animales silvestres 20.
La técnica MAT se emplea para detectar anticuerpos en el suero. Si bien es altamente especifica, posee limitada sensibilidad en la fase aguda de la enfermedad, debido a que los anticuerpos son detectables alrededor de los 7 a 10 días de la aparición de los síntomas. En general, requiere una segunda muestra, lo cual representa un inconveniente al trabajar con animales silvestres ya que su obtención se tornaría dificultosa o imposible en algunos casos, como el de los murciélagos. Por otra parte, tratándose de animales silvestres, deberían confrontarse con toda una batería antigénica existente en la región, sin olvidar que requiere más tiempo pues este grupo de bacterias crece lentamente 8.
En un estudio cuyo objetivo fue comparar diferentes técnicas de diagnóstico de leptospirosis en ganado ovino, ningún animal resultó positivo por cultivo, aunque 6 de ellos fueron positivos por PCR convencional en muestras de riñón y 11 fueron positivos por PCR cuantitativa, lo cual demostró la eficacia de la técnica PCR para detectar Leptospira sp. en muestras de tejido renal 13. La efectividad de la PCR con el uso de primers LipL32 expresando especificidad solamente a leptospiras patógenas, fue confirmada en Colombia, cuando un grupo de investigación realizó análisis con diferentes concentraciones de ADN hallando que el gen LipL32, utilizado para identificación en nuestro trabajo, es aplicable para la identificación molecular de aislamientos patógenos de Leptospira sp. de diversas fuentes 21.
Resulta fundamental generar propuestas de investigación que puedan ampliar el conocimiento de la fisiopatología de la leptospirosis, así como incorporar técnicas que identifiquen la presencia del agente etiológico. La PCR aplicada a la detección de leptospiras es sustancial para identificar cuál o cuáles son los serogrupos circulantes en una región, y así interpretar la epidemiología de la enfermedad. Aunque es indiscutible la importancia de los murciélagos como dispersores de semillas y controladores de plagas, ellos también pueden generar problemas de salud pública, por lo cual es necesario profundizar los estudios epidemiológicos de la enfermedad, así como ejecutar campañas de prevención encauzadas al mejoramiento de la salud humana y animal.
1. Barquez RM, Giannini NP, Mares MA. 1993. Guide to the bats of Argentina, Publ. Oklahoma Museum of Natural History, University of Oklahoma (USA), p. 119. [ Links ]
2. Barquez RM, Díaz MM. 2009. Los murciélagos de Argentina: clave de identificación, Ed. Magna, Tucumán (Argentina), p. 69. [ Links ]
3. Bharti A, Nally J, Ricaldi J, Matthias M, Diaz M, Lovett M, Levett P, Gilman R, Willig M, Gotuzzo E, Vinetz J. 2003. Leptospirosis: a zoonotic disease of global importance. Lancet Infect Dis 3: 757-771. [ Links ]
4. Bezerra J, Carvalho E, Hartskeerl RA, Ho PL. 2011. Evaluation of use of selective PCR amplification of LPS biosynthesis genes for molecular typing of leptospira at the serovar level. Curr Microbiol 62: 518-524. [ Links ]
5. Brihuega B, Paván M, Cairó F, Venzano A, Auteri C, Funes D, Romero G, Samartino L. 2007. Leptospira patógena en riñón de Didelphys albiventris (comadreja). Rev Arg Microbiol 39: 19. [ Links ]
6. Brihuega BF. 2008. Diagnóstico y tipificación de leptospiras. Temas de Zoonosis 4: 221-227. [ Links ]
7. Bunnell JE, Hice CL, Watts DM, Montruel V, Tesh RB, Vinetz JM. 2000. Detection of pathogenic Leptospira spp. infections among mammals captured in the Peruvian Amazon Basin region. Am J Trop Med Hyg 63: 255-258. [ Links ]
8. Cardona MN, Moros VR, López LE, Pérez CJ, Hernández RC. 2008. Diagnóstico de leptospirosis mediante PCR en pacientes con síndrome febril icterohemorrágico. Rev Soc Venez Microbiol 28: 24-30. [ Links ]
9. Cox TE, Smythe LD, Leung LK. 2005. Flying foxes as carriers of pathogenic Leptospira species. J Wildl Dis 41: 753-757. [ Links ]
10. Cullen PA, Cordwell SJ, Bulach DM, Haake DA, Adler B. 2002. Global analysis of outer membrane proteins from Leptospira interrogans serovar Lai. Infect Immun 70: 2311-2318. [ Links ]
11. Cullen PA, Xu X, Matsunaga J, Sanchez Y, Ko AI, Haake DA, Adler B. 2005. Surfaceome of Leptospira spp. Infect Immun 73: 4853-4863. [ Links ]
12. Dietz C, Von Helversen O. 2005. Claves de identificación ilustradas de los murciélagos de Europa. On line: www.federmaus-dietz.de/publications/Dietz & Helversen 2004 ID-key Spanish.pdf [ Links ]
13. Fornazari F, da Silva RC, Richini VB, Beserra HE, Luvizotto MC, Langoni H. 2012. Comparison of conventional PCR, quantitative PCR, bacteriological culture and the Warthin Starry technique to detect Leptospira spp. in kidney and liver samples from naturally infected sheep from Brazil. J Microbiol Meth 90: 321-326. [ Links ]
14. Haake DA, Suchard MA, Kelley MM, Dundoo M, Alt DP. 2004. Molecular evolution and mosaicism of leptospiral outer membrane proteins involves horizontal DNA transfer. J Bacteriol 186: 2818-2828. [ Links ]
15. Hernández P, Gómez AP. 2011. Leptospirosis: una zoonosis que afecta a la salud pública y la producción pecuaria Rev Cienc Anim 4: 15-23. [ Links ]
16. Levett P. 2004. Leptospirosis: A forgotten zoonosis? Clin Appl Immunol Rev 4: 435-448. [ Links ]
17. Lilenbaum W, Monteiro RV, Albuquerque CE, Ristow P, Fraguas S, Cardoso VS, Fedullo LP. 2004. Leptospiral antibodies in wild felines from Rio de Janeiro Zoo, Brazil. Vet J 168: 191-193. [ Links ]
18. Magalhaes J, Teruszkin I, Sutter F, Días A, Hillen L, Pereira MM. 2010. Multiplex PCR-based detection of Leptospira in environmental water samples obtained from a slum settlement. Mem Inst Oswaldo Cruz 105: 353-355. [ Links ]
19. Marchesin SR, Beguelini MR, Faria KC, Moreira PR, Morielle E. 2008. Assessing genetic variability in bat species of Emballonuridae, Phyllostomidae, Vespertilionidae and Molossidae families (Chiroptera) by RFLP-PCR. Genet & Molec Res 7: 1164-1178 [ Links ]
20. Matthias MA, Díaz MM, Campos KJ, Calderon M, Willig MR, Pacheco V, Gotuzzo E, Gilman RH, Vinetz JM. 2005. Diversity of bat-associated Leptospira in the Peruvian Amazon inferred by bayesian phylogenetic analysis of 16S ribosomal DNA sequences. Am J Trop Med Hyg 73: 964-974. [ Links ]
21. Moreno N, Agudelo P. 2010. Aplicación de las pruebas de PCR convencional simple y múltiple para la identificación de aislamientos de Leptospira sp. en Colombia. Rev Peru Med Exp Salud Públ 27: 548-556. [ Links ]
22. Pavlin BI, Schloegel LM, Daszak P. 2009. Risk of importing zoonotic diseases through wildlife trades, United States. Emerg Infect Dis 15: 1721-1726. [ Links ]
23. Romero MH, Sánchez JA, González LM. 2011. Revisión sobre la importancia de la fauna silvestre en la epidemiología de la leptospirosis. Biosalud 10: 112-122. [ Links ]
24. Rojas J, Céspedes M. 2008. Desarrollo del PCR REP1 para diferenciar serovares de Leptospìra sp y comparación con la prueba inmunológica MAT (ensayo microscópico de aglutinación) Rev Per Obst Enf 4: 72-78. [ Links ]
25. Rosario LA, Arencibia DF, Batista N, Jirón W, Suárez YE, Infante JF. 2012. Caracterización de aislamientos clínicos de Leptospira por métodos fenotípicos y moleculares en la República de Nicaragua. VacciMonitor 21: 6-12 [ Links ]
26. Rosadio RA, Véliz AA, Castillo HD, Yaya KL, Rodríguez AH, Rivera HG, Wheeler JC. 2012 Seroprevalencia a serovares de leptospiras patógenas en alpacas y vicuñas de los departamentos de Huancavelica y Ayacucho, Perú. Rev Inv Vet Perú 23: 350-356. [ Links ]
27. Smythe LD, Field HE, Barnett LJ, Smith CS, Dohnt MF, Symonds ML, Moore MR, Rolfe PF. 2002. Leptospiral antibodies in fying foxes in Australia. J Wildl Dis 38: 182-186. [ Links ]
28. Thirsa FB, Spichler A, Berardis EG, Husch AC, Fernandes M, Martos M, Mouriz ES, Veiga DR, Vinetz JM. 2010. The contribution of bats to leptospirosis transmission in São Paulo city, Brazil. Am J Trop Med Hyg 82: 315-317. [ Links ]
29. Zetun CB, Hoffmann JL, Silva RC, Souza LC, Langoni H. 2009. Leptospira spp. and Toxoplasma gondii antibodies in vampire bats (Desmodus rotundus) in Botucatu region, SP, Brazil. J Venom Anim Toxins Incl Trop Dis 15: 547-552. [ Links ]














