I. INTRODUCCIÓN
En el mundo de los micronadadores es usual que al observar ciertas células autopropulsadas con un microscopio tradicional y seguirlas con un software de tracking, las mismas exhiban trayectorias con formas oscilatorias. Un claro ejemplo de esto son los espermatozoides. Es posible realizar modelos predictivos de la dinámica de estas células, para los cuales se necesitan tener datos fehacientes de su movimiento. Estos modelos tienen múltiples aplicaciones biológicas, tanto en medicina reproductiva 1,2 como en la acuicultura o industria pecuaria. Por ejemplo, con modernos desarrollos micro-fluídicos 3, 4 se pueden realizar dispositivos miniaturizados del tipo “lab on a chip” con prometedoras aplicaciones clínicas.

Fig. 1: Trayectoria oscilatoria de un espermatozoide (puntos rojos) junto al camino medio calculado (cruces azules). Los puntos de la trayectoria son las posiciones del centro de la cabeza (disco morado) en el tiempo. El punto final de la trayectoria es el disco morado. En el zoom se muestra la distancia que corresponde a la amplitud de cabeceo ALH (barras marrones) y donde se toman los cruces para la frecuencia de cabeceo BCF (cuadrados verdes).
Para generar estos modelos es habitual que a partir de las trayectorias 2D se calculen parámetros de movimiento. Hay tres parámetros en particular que se utilizan en la clínica 5 y en diferentes modelos 1,6. Primero, la VAP, average path velocity, que es la velocidad de un camino medio. El mismo se calcula a partir de la trayectoria seguida, eliminando la oscilación o “cabeceo” típico de la misma, para seguir el desplazamiento medio del centro de masa de la célula, como se muestra en la Fig. 1. En ésta, los puntos rojos son las posiciones del centro de la cabeza del espermatozoide en cada imagen, mientras que los puntos azules corresponden al camino medio calculado. El segundo parámetro es la BCF, beat cross frequency, y se define como la frecuencia con la cual se cruzan el camino medio y la trayectoria original. Por último, la ALH, amplitude of lateral head displacement, que se mide como la máxima separación entre ambos caminos.
Con el fin de realizar este análisis de movimiento, es estándar emplear ciertos softwares llamados Computer Assisted Sperm Analysis, CASA, los cuales recuperan las trayectorias de los espermatozoides y permiten el cálculo de estos parámetros de movilidad 7. Diseñados originalmente para ser utilizados en espermatozoides humanos, estos son muy útiles y se emplean para varias especies. Una dificultad que presentan, por ejemplo, es su generalidad. Sus resultados dependen de los parámetros de configuración, por ejemplo, de detección y adquisición de datos, lo que da lugar a que las mediciones entre distintos laboratorios puedan variar 8. Esta variabilidad sucede particularmente con poblaciones que demuestran distribuciones heterogéneas de sus parámetros entre individuos; incluso estos parámetros pueden cambiar con el tiempo. Esta situación plantea un desafío significativo para su análisis. Como resultado, puede que la configuración del software por defecto no sea la óptima para toda la población, o que no sea representativa para todo tiempo. Adicionalmente, no existe un protocolo estándar a seguir para realizar las mediciones, como tampoco una disposición experimental sugerida. Por esta razón se pueden encontrar ciertos problemas a la hora de realizar un modelado objetivo de la dinámica de estas células.
Con el objetivo de obtener datos de calidad y representativos de la movilidad poblacional espermática, en este trabajo se presenta un algoritmo que busca analizar simultáneamente múltiples trayectorias con los parámetros adecuados para cada célula. En suma, su propósito es llevar a cabo un análisis eficaz de una gran cantidad de datos de forma automatizada. Esta herramienta de trabajo espera ofrecer múltiples aplicaciones a la industria de la salud humana, ganadería o acuicultura.
II. MÉTODOS
Preparación de muestras
Se utilizaron espermatozoides acuáticos (aquasperm), dada la variabilidad de los parámetros que presentan a lo largo de su tiempo de vida media. Las muestras fueron extraídas y preparadas por colaboradores de Aqua Analytix SpA de la acuicultura de Chile (comunicación privada). Dichas muestras se conservan refrigeradas entre 4-6°C por menos de 24 horas, antes de realizar las grabaciones de su movimiento. En el caso de espermatozoides humanos, M. Bettera cedió datos de su tesis doctoral en física, titulada “Dinámica de espermatozoides humanos micro-confinados” (FaMAF-UNC, a ser publicada en digesto UNC).
Obtención de trayectorias
Se filman videos de 400 fps de un segundo de duración con una cámara Zeiss Lab A1 y objetivo Zeiss A Plan ph1 10X/0.25 de los espermatozoides acuáticos. Para espermatozoides humanos se utiliza una cámara Andor Zyla VSC-10051 10X y se grabó a 100 fps 9.
Para realizar el seguimiento de los espermatozoides se utiliza el software BioTracker desarrollado por el grupo de materia Blanda y Activa en FaMAF-UNC 10, 11. Este programa está diseñado para reconstruir las trayectorias de micronadadores en general, pudiéndolo adaptar según la configuración de parámetros que se utilicen, y analizar ciertos aspectos de sus trayectorias, como cambios en su dirección o el largo de trayectoria, entre otros. El mismo consiste en tres etapas, cada una con su archivo de salida. En este trabajo se utiliza en particular la segunda de éstas, generada con el programa Linker, el cual conecta las posiciones de los micronadadores de distintas imágenes para recrear las trayectorias.
Algoritmo de ventana automática
En primer lugar, para calcular el camino medio se toma el promedio de los puntos de la trayectoria real dentro de cierta ventana temporal. Por ejemplo, si se utiliza una ventana temporal de diez cuadros, cada posición del camino medio será un promedio de las posiciones de los cinco puntos (o tiempos) anteriores y los cinco posteriores. De esta forma se obtiene una posición o camino medio de la trayectoria seguida. El valor de esta ventana temporal varía en función del tipo de trayectoria y de sus parámetros de movimiento. Para calcular la velocidad del camino medio (VAP) se utiliza diferencia centrada de tres puntos.
El método para seleccionar la ventana para cada trayectoria es el siguiente: se desarrolló un programa en Python que utiliza una ventana temporal inicial, la cual sigue siendo en cierta forma arbitraria, y genera un camino medio inicial. A partir de éste se cuenta aproximadamente el número de puntos por período de la trayectoria. Luego se vuelve a calcular el camino medio utilizando como ventana esta cantidad de puntos por período y se calcula el valor promedio de la VAP a lo largo de toda la trayectoria. Se suman dos puntos extras a la ventana temporal y se calcula nuevamente este valor promedio de la VAP. Al cambiar el camino medio y seguir teniendo la misma duración temporal, cambia la velocidad, y si este cambio es negativo, se acepta la ventana. Nuevamente, se suman un par de puntos a la ventana y se repite el cálculo. Se continúa de esta forma hasta que el cambio en la velocidad media sea positivo, lo que significa que el camino medio aumentó su distancia total y la ventana no es la correcta. Se busca un mínimo local de velocidad. De esta manera, con tan sólo un parámetro de entrada libre, el programa selecciona una ventana que se adapte a cada trayectoria celular.
III. RESULTADOS
Ventana Automática

Fig. 2: Trayectoria de un espermatozoide seguido por un segundo. En(a)y (b) el camino medio fue calculado con ventanas temporales distintas, de 28 y 36 puntos, respectivamente. El camino medio de (a) es considerado erróneo, mientras que el de (b), correcto. La velocidad media del track se indica en cada figura.
En la Fig. 2, se muestra una trayectoria tomada cerca del final de la vida media del espermatozoide acuático. En las Fig. 2(a) y Fig. 2(b) los caminos medios son calculados con ventanas de tiempo diferentes, de 28 y 36 puntos, respectivamente. La ventana temporal de 36 puntos en la Fig. 2(b), la cual fue seleccionada automáticamente por el algoritmo, da como resultado un camino medio correcto, según una inspección visual directa y criteriosa. Una ventana con un bajo número de puntos mantiene la oscilación, mientras que un número de puntos excesivo puede sobre ajustar. Por otro lado, en el camino medio de la Fig. 2(a) que se calculó a partir de la ventana temporal de 28 puntos, se pueden observar oscilaciones. Éstas son las que hacen que la velocidad del camino medio aumente, por lo que minimizar la velocidad es equivalente a “suavizar” el camino.
En cuanto a la eficacia del algoritmo, utilizando un procesador de escritorio Intel Core i7 de duodécima generación, el tiempo promedio de cálculo para 53 trayectorias de 400 puntos es de 0.9 segundos.
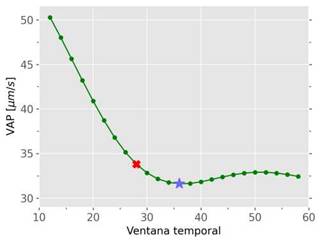
Fig. 3: Velocidad promedio del camino medio (VAP) de la trayectoria de la Fig. 2, en función de la ventana temporal utilizada para calcular el mismo. Con una cruz roja se indica la ventana temporal utilizada para el camino medio “incorrecto”, mientras que con una estrella azul el camino medio “correcto”.
En la Fig. 3 se presenta como varía el promedio de la velocidad de todo el camino medio, en función del valor de la ventana temporal utilizada para calcular el mismo. Este gráfico fue realizado a partir de la trayectoria de la Fig. 2, y las ventanas temporales utilizadas en la Fig. 2(a) y Fig. 2(b) se muestran con los mismos símbolos de “Incorrecto” y “Correcto”. Al comienzo del gráfico el camino medio sigue la trayectoria oscilando junto a esta. Mientras se aumenta el número de puntos con el cual se promedia el camino, este se suaviza y la velocidad media disminuye, hasta llegar a un mínimo local. Si se continúa aumentando la ventana temporal, se toman valores de otros períodos de forma incompleta y la velocidad aumenta, por lo que se generan estas oscilaciones del camino medio nuevamente.
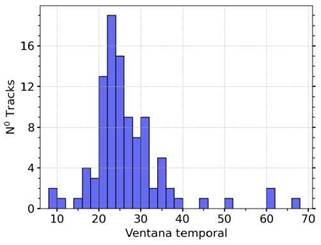
FIG. 4: Distribución de las ventanas temporales calculadas de forma automática para 98 trayectorias, seguidas (duración temporal) hasta por un segundo.
Por otro lado, en cada extremo de las trayectorias, hay un total de media ventana temporal de posiciones a las que no se les calcula su correspondiente posición del camino medio. Consecuentemente, al incrementar el valor de la ventana temporal utilizada, la cantidad de puntos en los “bordes” de la trayectoria que no cuentan con un punto correspondiente en el camino medio también aumenta. Como mencionamos anteriormente, el valor óptimo de la ventana temporal está relacionado con la cantidad de puntos por período. Es por esta razón que este efecto de “borde”, por lo general, es del orden de medio período de la trayectoria.
Si se compara la velocidad del camino medio con una ventana temporal de 20 puntos, 40.9 ^m/s, con la velocidad a partir de una ventana de 30 puntos, 32.8 ^m/s, hay una diferencia de aproximadamente un 20%. Es por esto que un cambio en la ventana temporal utilizada puede generar cambios notorios en los parámetros calculados, y una elección correcta de la misma tiene un papel importante en el análisis de este tipo de trayectorias.
En la Fig. 4 se presenta la distribución de ventanas elegidas de forma automática para una muestra de 98 trayectorias, seguidas por un tiempo de hasta un segundo. Se puede observar una alta heterogeneidad en la elección de la ventana, siendo la menor de 8 puntos y la mayor de hasta 68. En general, los espermatozoides acuáticos tienen una vida media móvil muy corta, de segundos, que decae heterogéneamente en las distintas células espermáticas de una misma muestra seminal 12.
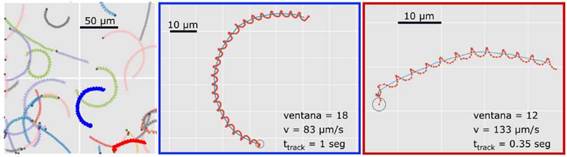
FIG. 5: Fragmento de todas las trayectorias seguidas durante un segundo. Con una ampliación ejemplificando sobre dos de ellas, marcadas en color rojo y azul, respectivamente. Estas trayectorias tienen distintas duraciones, ttrack, así como una diferencia de un orden de magnitud en sus velocidades.
Es por esto que la aplicación de una ventana temporal idéntica para todos estos caminos no sería representativa de la muestra. Suponiendo una ventana media de 22 puntos para todas las trayectorias, donde se ubica el pico de la Fig. 4, hay 16 trayectorias cuyas ventanas temporales difieren en más de 10 puntos de la seleccionada automáticamente. Teniendo en cuenta la diferencia de 20 % mencionada anteriormente, es probable que estos tracks no sean analizados de una forma óptima, conformando aproximadamente el 16% de la muestra.
En la Fig. 5 se presentan algunas trayectorias que fueron seguidas a lo largo de un segundo de video. Además, se muestra una ampliación sobre dos células en particular, recuadradas del mismo color con el que se resaltan en la respectiva figura (en rojo y azul). Las velocidades exhiben un orden de magnitud de diferencia, así como duraciones distintas, siendo éstas un ejemplo de la sorprendente heterogeneidad que se puede encontrar en tan sólo un segundo de vida espermática. Aún así, con el algoritmo de la ventana automática estas trayectorias pueden ser analizadas de forma independiente, mostrando buenos resultados para medir los parámetros de movimiento.
IV. DISCUSIÓN
Una dificultad que se puede encontrar si se quiere analizar una población nueva de espermatozoides, es que en principio no se sabe cuál es el valor óptimo para la ventana temporal para cada trayectoria. Se deberían realizar las mediciones de forma manual, tratando cada trayectoria individualmente y dificultando el análisis de una gran cantidad de datos. O como mencionamos anteriormente, tomar un valor de este parámetro en común para toda la población y probablemente analizar de forma errónea cierta parte de la misma. Por lo que el desarrollo de una herramienta para automatizar el análisis aporta grandes ventajas a la hora de estudiar la dinámica de estas poblaciones. Además, el mismo funciona para espermatozoides con distintas velocidades, desde 30 nm/s como en la Fig. 2, hasta 130 nm/s como en la Fig. 5.
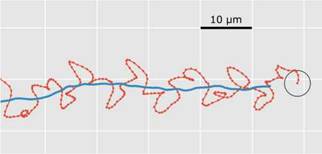
FIG. 6: Fracción de 3 segundos de una trayectoria de espermatozoide humano, seguido con una frecuencia de adquisición de 100 fps. Se utiliza una ventana temporal de 50 puntos y la velocidad promedio del camino medio es de 20 nm/s.
De los parámetros mencionados en la introducción, el algoritmo de este trabajo está pensado inicialmente para calcular la velocidad, aunque también mejora la precisión para la frecuencia y amplitud de cabeceo. Esto se puede observar en las oscilaciones en el camino medio de la Fig. 2, que pueden alterar la distancia máxima de separación entre ambos caminos.
El mismo algoritmo puede ser llevado a espermatozoides de otras especies. Por ejemplo, en la Fig. 6 se muestra una trayectoria de un espermatozoide humano con una velocidad de 20 nm/s. Cabe resaltar que esta velocidad media, así como su variabilidad, son mucho menores a las presentadas anteriormente. La misma puede tener un valor medio de 28nm/s, con una desviación estándar de 10nm/s 1. Éstas, aunque no tengan grandes variaciones en el tiempo, presentan ciertas irregularidades como las observadas en la Fig. 6, asociadas a su movimiento helicoidal en 3D. Ésto da lugar a una mayor complicación a la hora de encontrar una ventana adecuada. Como se mencionó al comienzo de este trabajo, estas trayectorias son seguidas en un microscopio tradicional, por lo que son proyecciones de un movimiento 3D a 2 dimensiones. Si se mide a pocos cuadros por segundo se observan simples oscilaciones, pero si se aumenta la velocidad de adquisición, entre 100 a 400 fps, en espermatozoides humanos se observan trayectorias oscilatorias altamente complejas, como las observadas en la Fig. 6.
Este comportamiento oscilatorio puede afectar la selección de la ventana. En este caso, la ventana temporal seleccionada por el algoritmo es de 50 puntos. Vale remarcar que a pesar de lo problemático de seleccionar la ventana para estas oscilaciones, nuestro algoritmo de todos modos logró encontrar un valor adecuado para el camino medio. Esto demuestra la capacidad de nuestro programa para adaptarse a diferentes especies, considerando que los parámetros que definen sus trayectorias varían, aunque todos los espermatozoides presentan un patrón de movimiento similar. El principio fundamental sigue siendo efectivo, resultando en una herramienta útil con amplio espectro de aplicabilidad.
V. CONCLUSIÓN
En este trabajo se presentó un algoritmo para analizar automáticamente trayectorias oscilatorias de múltiples micronadadores simultáneamente. El mismo funciona eficientemente, en cuanto a su automaticidad, para estimar los parámetros óptimos del algoritmo de análisis, que de otra manera pueden ser arbitrarios o difíciles de calcular para una gran cantidad de trayectorias. Resulta una herramienta útil al momento de estudiar la dinámica de poblaciones de espermatozoides que presentan una gran heterogeneidad en la distribución de sus parámetros, en función del tiempo. Su ventaja principal es la capacidad de adaptarse a los cambios de parámetros entre trayectorias, en lo que se centra la riqueza del desarrollo y sus futuras aplicaciones.














