Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista FAVE. Sección Ciencias veterinarias
On-line version ISSN 2362-5589
FAVE, Secc. Cienc. vet. vol.16 no.1 Santa Fe June 2017
COMUNICACIÓN CORTA
Detección molecular y análisis filogenético de Hepatozoon canis (Eucoccidiorida: Haemogregarinidae) en perros clínicamente sanos de Bahía Blanca (Buenos Aires)
Cicuttin GL*, De Salvo MN
Instituto de Zoonosis Luis Pasteur, Buenos Aires
* Correspondencia: Av. Díaz Vélez 4821, C1405DCD, CABA (Argentina). Correo electrónico: gcicuttin@gmail.com. Teléfono: +54 11 4958 9941. E-mail: gcicuttin@gmail.com
Editor: S. Nava
doi: https://doi.org/10.14409/favecv.v16i1.6651
Recibido: 5 Junio 2017.
Aceptado: 24 Junio 2017.
Disponible en línea: 29 Junio 2017
RESUMEN
Las enfermedades caninas causadas por protozoarios y transmitidas por garrapatas representan un importante problema en medicina veterinaria. El objetivo del estudio fue detectar molecularmente Hepatozoon spp., Babesia spp. y Theileria spp. en perros clínicamente sanos de distintas regiones de Argentina y analizar la diversidad genética de los hallazgos obtenidos. Se analizaron 163 muestras de ADN de sangre de perros (40 de Ciudad Autónoma de Buenos Aires; 33 de Bahía Blanca, Buenos Aires; 15 de Castelli, Chaco; 27 de Salsipuedes, Córdoba; 40 de Merlo, San Luis; y 8 de San Miguel, Corrientes). Mediante una PCR que amplifica un fragmento variable (460-540 pb) del gen ARNr 18S incluyendo la región V4 de los géneros Hepatozoon, Babesia y Theileria, el 12,1 % (4/33) de los perros de Bahía Blanca (Buenos Aires) resultaron positivos. Las secuencias obtenidas se identificaron como Hepatozoon canis y resultaron filogenéticamente similares a hallazgos en Sudamérica y en el resto del mundo. El estudio de H. canis en Argentina mediante técnicas moleculares de diagnóstico junto con el análisis filogenético resulta de suma importancia para conocer la situación de este patógeno en el país.
Palabras clave: Hepatozoon canis: Perros: Argentina
SUMMARY
Molecular detection and phylogenetic analysis of Hepatozoon canis (Eucoccidiorida: Haemogregarinidae) infecting healthy dogs from Bahia Blanca (Buenos Aires)
Tick-borne protozoan canine diseases represent a major problem in veterinary medicine. The aim of the study was to detect molecularly Hepatozoon spp., Babesia spp. and Theileria spp. in clinically healthy dogs from different regions of Argentina and to analyze the genetic diversity of the findings. DNAs extracted from 163 blood samples from dogs (40 from Buenos Aires city; 33 from Bahia Blanca, Buenos Aires; 15 from Castelli, Chaco; 27 from Salsipuedes, Córdoba; 40 from Merlo, San Luis; and 8 of San Miguel, Corrientes) were studied by PCR amplifying a variable fragment (460-540 bp) of the 18S rRNA gene including the V4 region of the genera Hepatozoon, Babesia and Theileria. Four dogs from Bahia Blanca (12.1%) were positive and the sequences obtained were identified as Hepatozoon canis and were phylogenetically similar to findings in South America and the rest of the world. The study of H. canis in Argentina with molecular methods together with phylogenetic analysis is important in order to know the situation of this pathogen in this country.
Key words: Hepatozoon canis: Dogs; Argentina
Introducción
Las enfermedades caninas transmitidas por garrapatas causadas por diferentes géneros de protozoos del Phylum Apicomplexa del Orden Piroplasmida (Babesia, Theileria y Cytauxzoon, entre otros) y Eucoccidiorida (Hepatozoon), representan un importante problema en medicina veterinaria (Greene, 2012).
La piroplasmosis canina es una enfermedad transmitida por garrapatas causada por diversas especies de los géneros Babesia y Theileria a nivel mundial. Se caracterizan por la presencia de fiebre, anorexia, anemia y esplenomegalia. La gravedad en el curso de la enfermedad depende de la especie de piroplasma, llegando a ser fatal en algunos casos (Ruiz et al., 2016; Greene, 2012).
Hepatozoon canis (Clase Conoidasida, Orden Eucoccidiorida, Familia Haemogregarinidae) fue descripto por primera vez en India en 1905 (Ruiz et al., 2013). Ha sido hallado infectando caninos en todo el mundo, con niveles de prevalencia muy disímiles (Greene, 2012; Linares, 2011). La transmisión ocurre por la ingestión de garrapatas de la especie Rhipicephalus sanguineus sensu lato infectadas con H. canis (Greene, 2012).
Las manifestaciones clínicas asociadas en los perros infectados son muy variadas, abarcando desde hallazgos casuales en animales aparentemente sanos hasta enfermedad debilitante con fiebre, letargia, pérdida de peso, anemia e hiperglobulinemia. El curso de la enfermedad suele ser prolongado, con aparición de signos clínicos en forma intermitente, con períodos de recuperación y recurrencia (Greene, 2012; Ruiz et al., 2013; Linares, 2011). Además, son frecuentes las coinfecciones con patógenos asociados a R. sanguineus s. l. como Ehrlichia canis, Anaplasma platys y Babesia canis (Greene, 2012; Ruiz et al., 2013).
En Argentina, sólo se notificó hasta el momento la circulación de Babesia vogeli y H. canis. Babesia vogeli fue confirmada molecularmente por primera vez en 2008 en caninos con signología compatible del Gran Buenos Aires (Eiras et al., 2008), siendo posteriormente hallada en otras regiones de Buenos Aires y Santa Fe (De Miguel et al., 2011; Ruiz et al., 2016). Hepatozoon canis fue descripto por primera vez en 1999 y ha sido notificado en las provincias de Buenos Aires, Chubut, Corrientes, Entre Ríos, La Pampa, Mendoza, Salta, San Luis y Santa Fe (Ruiz et al., 2013; Aráoz et al., 2016; Vezzani et al., 2017). Recientemente se reportó un caso clínico en Bahía Blanca (Aráoz et al., 2016). Sin embargo, la mayoría de los estudios realiza-dos en nuestro país utilizaron técnicas de microscopia óptica a partir de extendidos sanguíneos teñidos con distintas coloraciones (Linares, 2011; Aráoz et al., 2016; De Miguel et al., 2011; Ruiz et al. 2016; Vezzani et al., 2017).
El objetivo del presente estudio fue detectar molecularmente Hepatozoon spp., Babesia spp. y Theileria spp. en perros clínicamente sanos de distintas regiones de Argentina y analizar la diversidad genética de los hallazgos obtenidos.
Materiales y Métodos
Entre 2011 y 2016, en el marco de distintos proyectos sobre enfermedades bacterianas transmitidas por garrapatas, se recolectaron muestras sanguíneas de caninos domésticos clínicamente sanos de distintas áreas de Argentina: Ciudad Autónoma de Buenos Aires, Bahía Blanca (Buenos Aires), Castelli (Chaco), Salsipuedes (Córdoba), Merlo (San Luis) y San Miguel (Corrientes). Las muestras sanguíneas con anticoagulante EDTA fueron obtenidas por punción de vena cefálica o yugular con el consentimiento de los propietarios de los caninos. El ADN de la sangre entera fue extraído con AxyPrep Multisource Genomic DNA Miniprep Kit (Axygen Biosciences, EEUU) y High Pure PCR Template Preparation Kit (Roche, Mannheim, Germany) siguien-do las instrucciones de los fabricantes.
La detección de los géneros Hepatozoon, Babesia y Theileria fue realizada mediante PCR con los cebadores RLB-F2 (5´-GACACAGGGAGGTAGTGACAAG-3´) y RLB-R2 (5´-CTAAGAATTTCACCTCTGACAGT-3´) que amplifican un fragmento variable (460-540 pb) del gen ARNr 18S incluyendo la región V4, siguiendo las instrucciones de los autores (Matjila et al., 2008). Como control positivo se utilizó Babesia bigemina y como control negativo se usó agua libre de nucleasas.
Los productos amplificados fueron purificados con Wizard® SV Gel and PCR Clean-Up System (Promega, Madison, USA) y secuenciados en un secuenciador 3500 Genetic Analyzer sequencer (Applied Biosystems, Foster City, CA, EEUU).
Las secuencias obtenidas fueron editadas mediante el software BioEdit Sequence Alignment Editor (Hall, 1999) y alineadas con el program Clustal W (Larkin et al., 2007). Luego fueron comparadas con secuencias depositadas en GenBank y utilizadas para realizar el análisis filogenético mediante el método de máxima verosimilitud (ML, maximum-likelihood). El modelo de sustitución de mejor ajuste (best-fitting substitution model) fue determinado mediante el criterio de información bayesiano (bayesian information criterion) usando el modelo ML (Tamura 3-parameter) implementado en MEGA 6.0 (Tamura et al., 2013). Los valores de confianza fueron determinados mediante 1000 replicaciones de remuestreo (bootstrap).
Una secuencia consenso obtenida en este estudio fue depositada en la base de datos GenBank con el siguiente número de acceso: KY940658 (fragmento ARNr 18S de H. canis).
Resultados
En total se analizaron 163 ADNs extraídos de muestras sanguíneas (40 de CABA, 33 de Bahía Blanca, 15 de Castelli, 27 de Salsipuedes, 40 de Merlo y 8 de San Miguel).
El 2,5 % (4/163) de las muestras resultaron positivas mediante la técnica de PCR. Los perros positivos procedieron de Bahía Blanca (Buenos Aires) (12,1 %; 4/33).
Los cuatro productos amplificados pudieron ser secuenciados, resultando en un 100 % de similitud entre sí y con distintos hallazgos de H. canis en perros de Colombia (JN217101), España (AY461378), India (JN584477) y Taiwán (EU289222), 99,8 % con hallazgos en perros de Tailandia (DQ519358) y Venezuela (DQ439543), 98,4-99,7 % de Brasil (DQ198379,EU571737, DQ198378 y KF972441), 99,5 % de Venezuela (DQ439540) y 99,3 % de Sudán (DQ111754). El árbol filogenético construido con el fragmento del ARNr 18S se muestra en la Figura 1.
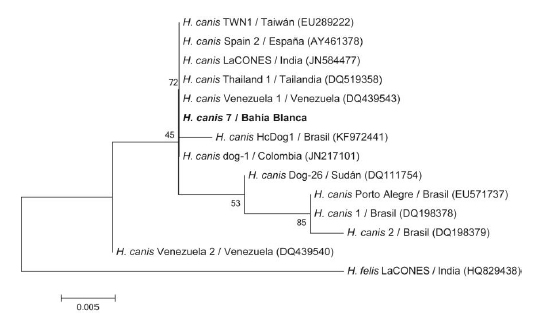
Figura 1: Árbol filogenético inferido por la comparación de secuencias parciales de ARNr 18S de Hepatozoon canis. Los números en los nodos son los valores de remuestreo. La barra de escala representa la diferencia en nucleótidos de las secuencias. Los números de acceso de GenBank están entre paréntesis. H.: Hepatozoon.
Discusión
Según nuestro conocimiento, este es el primer análisis filogenético de H. canis circulante en Argentina.
El nivel de infección encontrado en nuestro estudio en el área de Bahía Blanca (Buenos Aires), es similar a resultados previos a nivel mundial, considerando la amplia variación de las prevalencias existentes (Greene, 2012). La no detección en las otras áreas puede implicar la necesidad de estudiar un mayor número de muestras, así como la ocurrencia de distintas situaciones eco-epidemiológicas en las localidades más pequeñas con características suburbanas-rurales (Castelli, Salsipuedes, Merlo y San Miguel) y presencia de otras especies de garrapatas. Por otra parte, no se deTectaron los géneros Babesia y Theileria en nuestro estudio.
En Argentina, se ha sugerido que la hepatozoonosis canina se encuentra en expansión (Vezzani et al., 2017). Sin embargo, los estudios de la enfermedad a nivel poblacional son escasos, Vezzani et al. (2017) realizaron un amplio estudio sobre animales del sur del Gran Buenos Aires, encontrando una prevalencia de 2,3 % mediante observación microscópica de frotis sanguíneos; las demás publicaciones sobre el tema son relatos de casos o series de casos, principalmente como resultado de hallazgos casuales (Aráoz et al., 2016; Linares, 2011). Por otra parte, sólo dos estudios en Argentina utilizaron técnicas moleculares para el diagnóstico (Aráoz et al., 2016; Eiras et al., 2007) pero no existen secuencias disponibles de H. canis hallados en Argentina en GenBank, con lo cual la diversidad filo-genética de H. canis en nuestro país permanece prácticamente desconocida, así como tampoco se puede descartar la circulación en los perros de otras especies de Hepatozoon.
La diversidad genética de H. canis reportada en perros de todo el mundo varió entre 97 y 100 % para el ARNr 18S (Greene, 2012). En Argentina, Eiras et al. (2007) indicaron que sus hallazgos fueron similares en un 99 % con H. canis, mientras que Aráoz et al. (2016) sólo refieren que las secuencias permitieron determinar su hallazgo como H. canis al compararlas con las secuencias disponible en GenBnak pero no mencionan el porcentaje de identidad. En nuestro estudio, la secuencia del fragmento de ARNr 18S de H. canis fue idéntica a secuencias de distintas regiones del mundo (incluyendo Colombia), y similar a otras de países sudamericanos como Venezuela y Brasil. Sin embargo, exceptuando a Brasil, la información genética de H. canis para Sudamérica es muy limitada. De todas formas, se debe considerar que la comparación fue realizada en base a un pequeño fragmento génico (ARNr 18S), sien-do necesario ampliar el estudio a otros genes para una mejor caracterización y filogenia.
Las técnicas moleculares utilizadas en este estudio permitieron la detección de H. canis en perros clínica-mente sanos de Bahía Blanca, así como el análisis filo-genético de los hallazgos. Es necesario estudiar un mayor número de muestras de distintas áreas del país para investigar la prevalencia de la hepatozoonosis canina en nuestro país, siendo fundamental el uso de estas técnicas para caracterizar molecularmente los hallazgos en casos clínicos de la enfermedad.
Bibliografía
1. Aráoz V, Baneth G, Cáceres JP, Giannitti F. 2016. Infección por Hepatozoon canis en una perra con tumor venéreo transmisible en Bahía Blanca, Argentina. Vet. Arg. 34: 350 [ Links ]
2. De Miguel MC, Ferradas LA, Rosa A. 2011. Primer caso de babesiosis canina en Junín, provincia de Buenos Aires. En XI Congreso Nacional de AVEACA, Buenos Aires. p. 125. [ Links ]
3. Eiras DF, Basabe J, Mesplet M, Schnittger L. 2008. First molecular characterization of Babesia vogeli in two naturally infected dogs of Buenos Aires, Argentina. Vet. Parasitol. 157: 294-298. [ Links ]
4. Eiras DF, Basabe J, Scodellaro CF, Banach DB, Matos ML, Krimer A, Baneth G. 2007. First molecular characterization of canine hepatozoonosis in Argentina: Evaluation of asymptomatic Hepatozoon canis infection in dogs from Buenos Aires. Vet. Parasitol. 149: 275-279. [ Links ]
5. Greene CE. 2012. Infectious Diseases of the Dog and Cat. Ed. Elsevier, Missouri. 1354 pp. [ Links ]
6. Hall TA 1999. BioEdit: A user friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids Symp. Ser. 41: 95-98. [ Links ]
7. Larkin MA, Blackshields G, Brown NP, Chenna R, Mcgettigan PA, McWilliam H, Valentin F, et al. 2007. Clustal W and Clustal X Version 2.0. Bioinformatics 23: 2947-2948. [ Links ]
8. Linares C. 2011. Hepatozoonosis canina en la provincia de Mendoza, Argentina. Hallazgos clínicos y de laboratorio. Tesis. Facultad de Ciencias Veterinarias y Ambientales, Universidad Juan Agustín Maza. [ Links ]
9. Matjila PT, Leisewitz AL, Jongejan F, Bertschinger HJ, Penzhorn BL. 2008. Molecular detection of Babesia rossi and Hepatozoon sp. in African wild dogs (Lycaon pictus) in South Africa. Vet. Parasitol. 157: 123-127. [ Links ]
10. Ruiz MF, Zimmermann RN, Aguirre FO, Bono MF, Widenhorn NI. 2013. Hallazgo de Hepatozoon canis en caninos (Canis familiaris) en la ciudad de Esperanza, Santa Fe (Argentina). Revista FAVE - Ciencias Veterinarias 12: 15-20. [ Links ]
11. Ruiz MF, Zimmermannm RN, Giorgis F, Canteros E, Varisco B, Piazzese S, Aguirre F, Orcellet V. 2016. Piroplasmosis canina: Reporte de casos en dos ciudades de la provincia de Santa Fe. En IV Jornada de difusión de la investigación y extensión, Esperanza (Santa Fe). [ Links ]
12. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. 2013. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Mol. Biol. Evol. 30: 2725-2729. [ Links ]
13. Vezzani D, Scodellaro CF, Eiras DF. 2017. Hematological and epidemiological characterization of Hepatozoon canis infection in dogs from Buenos Aires, Argentina. Vet. Parasitol. Reg. Stud. Rep. 8: 90-93. [ Links ]














