PUNTOS CLAVE
• Los virus ARN se replican por acción de una ARN poli merasa que carece de exonucleasa correctora. En cada replicación se producen errores que originan mutantes con alteraciones en la proteína S (proteína spike de la superficie exterior del virus), la llave de ingreso del virus a la célula humana. Cuanto más se replican los virus, más mutan, y se hace más probable que aparezcan variantes que escapan al sistema inmune.
• Las vacunas y los anticuerpos neutralizantes manten drían su protección contra las variantes del SARS-CoV-2 porque actúan sobre múltiples sitios de la proteína S.
• Además, las vacunas parecen inducir aumento de célu las T helper y citotóxicas que, junto con los anticuerpos neutralizantes, pueden inducir protección.
Se ha comenzado a utilizar el término resistencia para describir la eficacia reducida de las vacunas contra algunas variantes del virus SARS-CoV-2. Se habla de “resistencia del SARS-CoV-2 a las vacunas y a los anticuerpos” por homología con el concepto de resistencia microbiana. Pero la resistencia de los microorganismos a los antibióticos y agentes quimioterápicos emerge durante el tratamiento por presión selectiva, mientras que las variantes “resistentes” de SARS-CoV-2 se difunden en la población independien temente del uso de vacunas. En este artículo extraemos algunas conclusiones basadas en el análisis de las simi litudes y las diferencias entre ambos tipos de resistencia.
La resistencia de bacterias y virus a los antibióticos
Las bacterias y los virus pueden presentar resistencia natural o adquirida a los fármacos empleados para su tratamiento, sean antibióticos o quimioterápicos. Por ejemplo: las bacterias Gram negativas tienen resistencia natural a la penicilina porque una capa de lipopolisacá ridos y proteínas que rodea la de peptidoglicano de su pared celular impide su penetración.
La resistencia adquirida, en cambio, emerge en enfermos tratados con un fármaco (sea antibiótico o quimioterápico) a partir de mutantes espontáneas producidas al azar ya antes del contacto con ese fármaco. Una vez en su presencia, esas mutantes resistentes continúan reproduciéndose mientras el resto es eliminado. Si se sigue administrando solo ese fármaco, las mutantes pueden llegar a formar una población tan grande como la original, pero com puesta enteramente por bacterias o virus resistentes. Por esa razón se emplean dos o más fármacos en el tratamiento antirretroviral para HIV-Sida, y cuatro en el tratamiento inicial de la tuberculosis. Se trata de eliminar las mutantes a un agente administrando simultáneamente otro, u otros, con mecanismos de acción diferentes entre sí (Fig. 1).
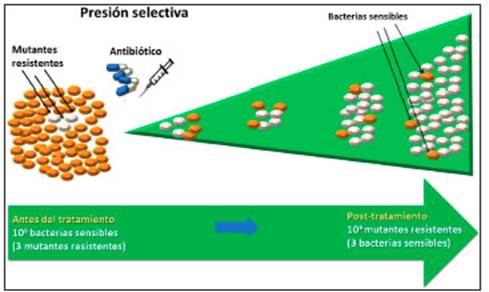
Fig. 1 Generación de resistencia bacteriana adquirida o secundaria. Imagen inspirada en: Mutations and selection, con permiso de ReAct- Action on antibiotics resistance (copyright). La figura original está disponible en: https://www.reactgroup.org/toolbox/understand/antibiotic-resistance/mutation-and-selection/
Mutaciones y virus ARN
El material genético de los coronavirus (como SARS-CoV-2) y los ortomixovirus (como influenza) es ARN y no ADN. Estos virus se replican y transcriben por acción de una ARN polimerasa dependiente de ARN (RdRp). A diferencia de las ADN polimerasas, que copian el ADN con alta fidelidad porque contienen un dominio de exo nucleasa correctora, las enzimas RdRp carecen de dicho dominio, por lo que la fidelidad de la transcripción no está asegurada y, en consecuencia, en la replicación del ge noma se producen errores frecuentes, con sustitución de nucleótidos. Ese es el origen de las mutantes que, según su aptitud física (fitness), pueden extinguirse si la muta ción genera una desventaja adaptativa, o bien, prosperar en la comunidad si la mutación provee una ventaja. Así se originan las “variantes de escape del sistema inmune”.
El caso de los virus influenza
Los virus influenza A y B, causantes de la gripe, mutan constantemente, en forma lenta o súbita. Los cambios genéticos lentos ocurren a la deriva (antigenic drift), con una frecuencia anual cuantificable. El sistema inmune no reconoce entonces la nueva variante de escape y es necesario modificar la fórmula de la vacuna. Además, los niveles de anticuerpos contra los tipos A y B del virus influenza declinan significativamente a los seis meses siguientes a la vacunación, y al año ya no son detectables. La eficacia de la vacunación varía entonces de acuerdo a la inmunidad en la población, las diferencias entre las cepas circulantes y las incluidas en la vacuna y la eventual aparición de nuevas variantes de escape1.
Pueden ocurrir también cambios mayores por despla zamiento antigénico brusco (antigenic shift), y originar un nuevo subtipo o mezcla genética que incluya genes de distintos virus animales. Eso ocurrió en 2009, cuando emergió un nuevo virus A(H1N1) por combinación de genes procedentes de los virus de la gripe porcina, aviar y humana. Y este virus esta genéticamente relacionado con el responsable de la pandemia anterior, la gripe “española”, que causó unos 50 millones de muertes en 1918-19202.
Coronavirus y mutaciones
Los coronavirus poseen ciertas características que los diferencian de otros virus ARN: su genoma es de tamaño llamativamente grande (26-32 kb) y, en ausencia de un dominio de exonucleasa en su RdRp, poseen una exo-ribonucleasa (ExoN) que ayuda a corregir los errores producidos durante la replicación. Como resultado, la frecuencia de mutaciones es menor en los coronavirus (incluido el SARS-CoV-2) que en otros virus ARN que carecen de la enzima ExoN.
Mutantes, variantes, cepas y linajes
Aunque los términos “mutante”, “variante” y “cepa” del virus son usadas indistintamente en las descripciones epidemiológicas del SARS-CoV-2, no significan lo mis mo. Las mutaciones son cambios en la secuencia de nucleótidos del genoma y los virus que las portan son llamados “mutantes”.
Cuando una “mutante” se selecciona a través de numerosos ciclos de replicación viral, emerge una “va riante” viral. Si la variación da como resultado un virus con características fenotípicas diferentes, la variante es denominada “cepa”. Cuando, por secuenciación genética, se halla que una nueva variante representa una rama di ferente en el árbol filogenético, eso significa el nacimiento de un nuevo “linaje”3.
No se conoce bien por qué algunas variantes llegan a predominar sobre otras. Pero las cuestiones de mayor interés conciernen a su potencial clínico: ¿se transmiten más?, ¿son más virulentas? y, sobre todo, ¿pueden eva dir la inmunidad inducida por vacunación o por infección anterior? Si es así, se las denomina “variantes de escape” y a veces también “variantes resistentes”.
Resultan de particular interés las mutaciones que alteran la proteína S (spike protein) porque es la puerta de entrada del SARS-CoV-2 a la célula huésped y, como tal, el blanco de las vacunas. La proteína S une el virus a su receptor celular ACE2 (angiotensin-converting enzyme 2), la enzima convertidora de angiotensina I tipo 2. Esa unión es mediada por el RBD (receptor binding domain), un dominio de unión al receptor situado en S (Fig. 2).

Fig. 2 Estructura del receptor ACE2 unido al dominio de unión al receptor (receptor-binding domain, RBD) de la proteína S del virus SARS-Cov2. La estructura crista lográfica fue creada a partir de una proteína quimérica entre el RBD de SARS-CoV-2 junto con el receptor ACE2 Humano (PDB_ID: 6VW1); la visualización de la estructura se realizó mediante el programa Chimera 1.10.2. Fuente original del PDB: Shang J, Ye G, Shi K, et al. Structural basis of receptor recognition by SARS-CoV-2, Nature 2020; 581: 221-4.
A partir de marzo de 2020, las variantes de SARS-CoV-2 que contienen la sustitución D614G (de ácido aspártico a glicina en la posición 614) de la proteína S se expandieron rápidamente en forma simultánea en distintas regiones y, en pocos meses, pasaron a ser la forma dominante del virus en todo el mundo. ¿A qué se debió esto? ¿Pudo ser al azar? Varias investigaciones, en humanos y en modelos animales, mostraron que estas mutantes D614G pueden ubicar su dominio de unión RBD de modo de interactuar más eficientemente con el receptor ACE2. Esta mutación se asocia con una mayor carga de ARN viral en nasofaringe, lo que aumenta la transmisibilidad4,5.
¿Qué variantes del SARS-CoV-2 son importantes?
Una vez conocida la secuencia del SARS-CoV-2, el es fuerzo de las compañías farmacéuticas y la academia se dirigió, y con notable éxito, al desarrollo y la producción de vacunas. Pero también fueron apareciendo variantes del virus.
La variante de linaje B.1.1.7, identificada en el Reino Unido en octubre de 2020, muestra 9 cambios (deleciones y sustituciones) en la proteína S en comparación con la secuencia original. La variante B.1.351, descubierta en Sudáfrica el mismo mes, presenta 10 cambios. La variante P.1, procedente de Manaos, Brasil, fue identificada en diciembre de 2020 y muestra 12 cambios. Varios de es tos cambios son mutaciones en el RBD de la proteína S, cuya subunidad S1 es responsable de la interacción con la enzima ACE2 del receptor celular del SARS-CoV-2 en el huésped humano. La superficie de interacción del RBD con ACE2 es una pequeña secuencia de 25 aminoácidos situados en la punta del RBD y es el sitio para la unión de muchos potentes anticuerpos neutralizantes.
Las mutaciones en la superficie del RBD que interactúa con ACE2 en estas tres cepas variantes de SARS-CoV-2 son: N501Y (sustitución de asparagina por tirosina en posición 501) en B.1.1.7; K417N (de lisina por asparagina en posición 417) y E484K (ácido glutámico por lisina en posición 484) en B.1.351; y finalmente tres mutaciones en P.1: K417T (lisina por treonina en posición 417), E484K y N501Y6.
B.1.1.7, B.1.351, y P.1 fueron clasificadas por la OMS como “preocupantes” (of concern) en febrero de 2021, lo que se basó en su mayor transmisión, la reducción de la neutralización por anticuerpos tanto monoclonales como de suero de convalecientes, y también la menor capa cidad neutralizante del suero de sujetos vacunados7-9. La variante de linaje B.1.1.7, sin embargo, parece ser suficientemente sensible a los anticuerpos generados por las vacunas (al menos por las dos vacunas ARNm) y, en consecuencia, no se espera una reducción significativa de la efectividad de la vacunación frente a ella. En cambio, la variante de linaje B.1.351 ha mostrado ser hasta seis veces menos sensible a los anticuerpos neutralizantes inducidos por vacunación.
A estas variantes se agregó la de la India B.1.617, de rápida y amplia propagación, que fue declarada preocupante por la OMS en mayo. Su genoma tiene 13 mutaciones, 3 de ellas en el código de la proteína S. Los sueros de convalecientes y de personas vacunadas neutralizarían parcialmente esta variante10.
Sin embargo, se debe recordar que los títulos de neutralización in vitro no siempre reflejan la situación in vivo. Las vacunas inducen la producción de altos niveles de anticuerpos, y una caída en la potencia neutralizante puede no ser significativa. Además, es posible que las respuestas de inmunidad celular, dependientes de las células T, no se vean afectadas.
En su informe del 9/5/2021, el Consorcio Proyecto PAIS (MINCYT) ha observado en la ciudad de Buenos Aires (CABA) y el Gran Buenos Aires (GBA), una mayor frecuencia de detección de las variantes B.1.1.7 (Reino Unido), P.1 (Manaos) y de la denominada variante Andina (linaje C.37, mutación L452Q), en casos sin nexo epide miológico con turismo al exterior. Las frecuencias son: del 27% en CABA y del 13% en el GBA para la variante del Reino Unido; cerca de 32% para la variante de Manaos en ambos distritos, y del 33% (CABA) y el 49% (GBA) para la variante Andina. Hasta esa fecha no se había detectado la variante de Sudáfrica11.
También se observó que más del 90% de los virus SARS-CoV-2 que circulan actualmente en estas áreas poseen la mutación D614G en la proteína S que los dife rencian de los que circularon en la primera ola.
¿Cómo emergen las variantes de SARS-CoV-2?
No hay evidencias sobre las causas del éxito de las variantes del Reino Unido, de Sudáfrica ni de otras más recientes. Algunas hipótesis atribuyen su expansión ex plosiva a la presión selectiva ejercida por el tratamiento intermitente con antivirales, anticuerpos monoclonales, plasma de convalecientes, o por la persistencia en el organismo de grandes cantidades de virus, como ocurre en enfermos de COVID-19 inmunocomprometidos con larga evolución de la enfermedad12-14.
En áreas densamente pobladas con escasas medidas de prevención o protección, el virus se difunde libremente durante la pandemia y halla nuevos huéspedes donde re plicarse. Así aumenta también la probabilidad de generar mutaciones espontáneas con mejor fitness, que pasan a ser dominantes. India y Manaos ejemplifican esa situación epidemiológica.
Anticuerpos, sueros y vacunas
La proteína S es el blanco de los anticuerpos neutralizan tes los que, al unirse a la región RBD de esta proteína, impiden que el virus se una al receptor ACE2 de las cé lulas del huésped. Se ha mostrado que las mutaciones en la proteína S pueden disminuir o suprimir la actividad neutralizante de anticuerpos monoclonales, pero que la neutralización por suero inmune policlonal se mantiene. Dado que un suero policlonal, como el suero de convale ciente (o el suero equino hiperinmune), contiene múltiples anticuerpos contra diferentes partes de la proteína S, su acción se ve menos afectada por mutaciones puntuales que la de los anticuerpos monoclonales, que están dirigi dos contra un único determinante antigénico15-18.
Rol de la inmunidad celular
La inmunidad contra SARS-CoV-2 no se basa solo en anticuerpos, también juega un rol importante la inmunidad celular. Además de inducir el desarrollo de anticuerpos neutralizantes, las vacunas anti-COVID inducen el aumen to de las células T helper y T citotóxicas. Pero las pruebas serológicas que detectan anticuerpos circulantes, no son una medida de la actividad de las células T, ni siquiera de las células B de memoria. Por lo tanto, aunque la presencia de anticuerpos neutralizantes se correlacione con la protección conferida por la vacuna, no es su único biomarcador19.
Debe tenerse en cuenta que las pruebas serológicas solo detectan anticuerpos circulantes, no así la actividad de las células T, ni de las células B de memoria. Por lo tanto, aunque la presencia de anticuerpos neutralizantes se correlacione con la protección conferida por la vacuna, no es su único biomarcador19.
Consideraciones finales
Cuanto más se replican los virus, más mutan. Y cuanto más mutan, es más probable que aparezcan variantes dominantes que constituyan un problema. Lo mejor que podemos hacer para prevenirlo es tener menos enfermos circulando, menos virus circulando20-22.
Por evolución natural, algunos virus seleccionan las mutaciones que les permiten infectar, replicarse y trans mitirse con mayor eficiencia sin eliminar al huésped, lo que les facilita la propagación. En cambio, la circulación de otros de alta letalidad (como Ebola o hantavirus) se circunscribe a determinadas áreas geográficas. De acuerdo con esto, sería esperable que SARS-CoV-2 tienda a evolucionar a variantes más infecciosas, pero menos virulentas.
Las intervenciones de salud pública como máscaras, distanciamiento físico y limitaciones a las grandes reu niones siguen siendo efectivas, pero el control de una variante más transmisible requerirá la aplicación más estricta y generalizada de estas medidas.
En cuanto al rol de la vacunación, debido a que las vacunas actuales inducen una respuesta inmune poli clonal a múltiples sitios de la proteína S, se espera que mantengan una protección razonable y efectiva a pesar de los cambios en los sitios antigénicos presentes en las variantes del SARS-CoV-2.