Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista veterinaria
versión On-line ISSN 1669-6840
Rev. vet. vol.25 no.2 Corrientes dic. 2014
TRABAJOS DE INVESTIGACIÓN
Caracterización electroforética de proteínas de la "paja amarilla" (Sorghastrum setosum) incubada en rumen de bovinos
Slanac, A.L.1; Sgroppo, S.C.3; Kucseva, C.D.4; Balbuena, O.2
1Cátedra de Fisiología
2Cátedra de Nutrición y Alimentación, Facultad de Ciencias Veterinarias, UNNE.
3Cátedra de Bromatología, Facultad de Ciencias Exactas, Naturales y Agrimensura, UNNE.
4INTA Colonia Benítez (Chaco). E-mail: alslanac@vet.unne.edu.ar
Resumen
Con el objetivo de caracterizar las fracciones proteicas de Sorghastrum setosum durante el proceso de degradación ruminal, se tomaron 3 g de polvo acetónico obtenido a partir de muestras sometidas a distintos tiempos de incubación ruminal (0; 6; 12; 24; 48; 72 y 120 h). Las proteínas presentes en los residuos acetónicos fueron solubilizadas en buffer Tris pH 8 y cuantificadas por el método de Lowry. La caracterización se realizó por electroforesis desnaturalizante en placa (gel con gradiente de poliacrilamida 12% conteniendo SDS), de acuerdo al sistema de buffer discontinuo de Laemmli modificado (1970). El análisis cualitativo realizado a la hora cero mostró bandas de 87; 64; 58; 55; 51; 47; 46; 44; 42; 27 y 14 kDa. En el perfil proteico se detectaron bandas intensas de 46, 42 y 14 kDa, otras de menor intensidad de 64, 58 y 51 kDa, y un grupo de alto peso molecular en la región de 87 kDa. Se observaron modificaciones del perfil proteico a lo largo del proceso de degradación, detectándose nuevas bandas de alto y medio PM, así como la aparición e intensificación de bandas de bajo PM. Durante el proceso de degradación ruminal, se observó que las bandas de 24, 14 y 55 kDa estuvieron presentes durante las 120 horas de estudio; esta prolongada permanencia indica la escasa degradabilidad ruminal de esta gramínea.
Palabras clave: bovino, rumen, proteína, electroforesis, Sorghastrum setosum.
Electrophoretical characterization of proteins from Sorghastrum setosum ("sandysoil indiangrass") incubated in cattle rumendministration of selenium decreases lipid peroxidation in semen of Brahman bulls.
LWith the objective of characterize the protein fractions of Sorghastrum setosum ("sandysoil indiangrass") during its ruminal degradation, 3 g of acetone powder obtained from samples subjected to different ruminal incubation times were taken (0; 6; 12; 24; 48; 72 and 120 hours). Proteins present in the acetone residue were solubilized in Tris buffer pH 8 and quantitated by the Lowry method. The characterization was carried out by denaturing polyacrylamide gel electrophoresis (12% containing SDS) according to the modified Laemmli buffer system (1970). The qualitative analysis performed at zero hour showed intense bands of 87; 64; 58; 55; 51; 47; 46; 44; 42; 27 and 14 kDa. For the protein profiles, intense bands of 46; 42 and 14 kDa were detected, as well as others of lower intensity of 64; 58 and 51 and finally a group of high molecular weight in the region of 87 kDa. During the degradation process, modifications of the protein profile were observed, being detected new bands of high and half PM, as well as the appearance and increase of low PM bands. During the process of ruminal degradation it was observed that the bands of 24, 14 and 55 kDa were present during the 120 hours of study; this prolonged permanency indicates the scarce ruminal degradability of this gramineous.
Key words: cattle, rumen, protein, electrophoresis, Sorghastrum setosum.
Recibido: 29 julio 2014
Aceptado: 22 septiembre 2014
INTRODUCCIÓN
Las proteínas individuales que componen el total del proteoma de una planta en particular pueden ser separadas en geles de electroforesis, los cuales proporcion además el número y la cantidad relativa de la proteína en estudio7. La electroforesis discontinua SDS-PAGE es considerada como una herramienta de gran utilidad para estudios taxonómicos y genéticos en vegetales. Sin embargo su aplicación es limitada ya que se han observado, para algunas variedades, patrones electroforéticos heterogéneos en cuanto al número e intensidad de las bandas.
La proteína cruda es la fracción del forraje o alimento que está compuesta por una multitud de prótidos que difieren en la composición y secuencia de aminoácidos, así como por sus estructuras secundarias, terciarias y cuaternarias. Tales características pueden ayudar a aclarar la variación de la degradación ruminal17. Las estructuras terciarias y secundarias probablemente afecten la tasa y extensión de la hidrólisis ruminal evidenciable mediante la técnica de electroforesis, razón por la cual la proteína del forraje podría ser cuantificada usando la electroforesis y densitometría18. La desaparición de la proteína durante la incubación ruminal también sería monitoreable cuantitativamente usando electroforesis10, lo que facilitaría la formulación de dietas basadas en las proteínas metabolizables y absorbibles21.
La técnica SDS-PAGE es la más usada para separar y caracterizar proteínas para estimar el grado de diversidad genética28. Se demostró que esta técnica es confiable porque la proteína de almacenaje es en gran parte independiente de fluctuaciones ambientales4, 28. La electroforesis se emplea en muchos estudios para clasificar y evaluar la variabilidad genética, así como para comparar las variaciones de los monómeros que caracterizan a los genes que cifran varias proteínas. Se la utilizó en estudios sobre especies como: Amaranthus14, sorgo20, sésamo15.
En cultivos de tomates y calabazas la electroforesis de las hojas se utilizó para evaluar la variabilidad genética y caracterizar la proteína cruda en Lyncopersicon y Trichosanthes3. Mediante esta técnica, se puedo diferenciar la diversidad genética en la especie Leucaena29 y se caracterizaron las fracciones de proteínas de las hojas de Panicum virgatum, Andropogon gerardii y Bromus inermis, que no fueron degradadas en rumen; el análisis de los geles indicó tres fracciones de 24, 26 y 56 kDa. Especies de gramíneas C4 tienen ciertas fracciones de proteínas que les permiten permanecer por más tiempo sin ser degradadas en el rumen24 .
La técnica SDS-PAGE permitió la identificación de varios cultivares de cereales, diversas pasturas y otros vegetales22. En semillas de girasol se determinó cuantitativamente la presencia de cinco polipéptidos principales26. También se caracterizaron gluteínas de alto peso molecular en gluten de trigo en cultivares de Uruguay8; se realizó la caracterización proteómica del grano de frijol azufrado5 y se evaluaron las proteínas de semillas del género Passiflora L. Passifloraceae23 .
Análisis electroforéticos de material aislado de Lupinus por SDS-PAGE, muestran bandas entre 14,4 y 66,2 kDa, que serían proteínas que estarían mayoritariamente representadas por globulinas12. Las globulinas son una clase de proteínas muy heterogéneas, clasificadas como alfa, beta, gama y conglutina27. En investigaciones realizadas en proteínas de L. angustifolius y L. albus, se observó la presencia de una beta conglutinina, compuesta de monómeros de PM que oscilaron entre 17 y 64 kDa9.
El objetivo del presente trabajo fue realizar la caracterización por electroforesis de las fracciones proteicas de Sorghastrum setosum (paja amarilla) del nordeste argentino, durante la incubación ruminal en bovinos.
MATERIAL Y MÉTODOS
El material vegetal evaluado fue recolectado en un campo del nordeste argentino (latitud sur 27º10’; longitud oeste 58º57’). Las muestras de S. setosum fueron tomadas por medio de cortes con tijeras a 10 cm de altura de todo el material disponible en condición de pastoreo, dentro de un marco de 50 x 50 cm. El método utilizado fue el de exclusión del pastoreo por medio de alambrado perimetral, con cortes a los 15, 30 y 45 días del corte de limpieza, en cada una de las estaciones del año. Las muestras del pasto, se colocaron en bolsas de papel limpias y puestas a secar en estufa de aire forzado a 55ºC durante 48 a 72 h y luego molidas en molino de cuchillas con mallas de 2 mm.
Para la incubación ruminal, se utilizó la técnica de suspensión in situ de bolsas de dacrón, en dos novillos cruza cebú de 550 kg PV provistos de fístula ruminal, que fueron mantenidos en potreros con pastura natural. Los animales recibieron ad libitum un suplemento mineral que contenía 12% Ca, 8% P y microelementos vehiculizados en sal común. En cada bolsa, de 9,5 x 18 cm, con una porosidad de 50 um, se colocaron 5 g de material seco de la pastura, que fueron introducidas en el rumen en forma secuencial en los siguientes tiempos: 120; 72; 48; 24; 12; 6; 3 y 0 horas. Luego del tiempo de incubación correspondiente, las bolsas fueron retiradas del rumen y sumergidas en agua fría por 5 min, después lavadas bajo agua corriente y por último secadas en estufa a 60ºC durante 72 h. También fueron pesadas.
Con el fin de evaluar las fracciones proteicas de la pastura en estudio, se tomaron 1-3 g de material seco y se mezclaron con 50 ml de acetona enfriada a 4ºC, luego se filtró por Buchner y lavó con acetona hasta decoloración total del residuo. El residuo acetónico fue secado en estufa al vacío a 30ºC, obteniéndose así el polvo acetónico. La extracción de proteínas se llevó a cabo pesando 0.3 g del polvo acetónico anteriormente preparado y poniéndolo en contacto con 25 ml de buffer Tris pH 8 (Tris 100 mM, EDTA 1 mM, PVPP 0,6 mg/100 ml), al que se le adicionaron 200 μl del inhibidor de proteasas fluoruro de fenilmetilsulfonilo (PMFS) 1 mM.
La mezcla se mantuvo 3 h en agitación a temperaturas inferiores a 10°C y luego se centrifugó durante 15 min a 13500 rpm en una centrífuga refrigerada. Se filtró por gasa y al sobrenadante se le adicionaron 4 g de ácido tricloroacético, dejándolo en contacto durante 30 min a 10ºC con agitación constante. Luego el residuo se centrifugó 10 min a 13500 rpm y el precipitado se lavó 2 veces con 10 ml de ácido tricloroacético al 1% y 3 veces con acetona al 80%, antes de secarlo en desecador durante 24 h. Las proteínas presentes en los residuos acetónicos fueron solubilizadas en el buffer Tris de pH 8 y cuantificadas por el método de Lowry. Todas las determinaciones se efectuaron por triplicado. Se utilizaron geles desnaturalizantes SDS-PAGE, según protocolo anteriormente descrito16.
El precipitado obtenido en la etapa de extracción se disolvió en 200 μl de buffer (Tris 10 mM, EDTA 1 mM, SDS 1%, mercaptoetanol 5%, glicerol 40%, azul de bromofenol 0,02%, pH 8-9). Las muestras se sonicaron durante 10 min y se calentaron a ebullición durante 1 min, previo a la siembra en el gel de "stacking" a razón de 20 μl de muestra por calle. Para las corridas se utilizó el sistema de electroforesis vertical (Bio-Rad Mini-Protean II, Hercules, California, USA), empleando un gel concentrador de 4% de acrilamida y un gel de separación con 1 % de acrilamida.
Debido a que la mayoría de las proteínas vegetales contienen más aminoácidos de tipo ácido (ácidos aspártico y glutámico) que básico (lisina y arginina), los
puntos isoeléctricos de la mayoría de ellas se encuentran en un pH menor a 7, por ello la solubilidad generalmente es mayor en la zona alcalina, razón por la cual se usó buffer de pH > 8 para solubilizar las proteínas de las distintas muestras.
Las corridas se realizaron a 25 mAmp a temperatura ambiente, hasta la salida del frente de avance de los colorantes. Para la fijación de los geles se usó una solución de isopropanol 25% y ácido acético glacial 10% durante 30 min. Para la tinción se utilizó una solución de Coomasie Brilliant Blue R-250 al 0,02% durante 24 h y la decoloración de los geles se efectuó con una solución de alcohol metílico 25% y ácido acético glacial 10%. Se utilizó un kit Sigma de patrones de referencia (Dalton Mark VII-L Standard Mixture) con un rango de PM de 14 000 a 70 000 Da. Los geles de poliacrilamida fueron escaneados con una impresora HP 4200 y analizados por medio del software para análisis de geles2 WinXGel 1.8.
Para determinar el peso molecular de las proteínas separadas por electroforesis, se determinaron los valores de la distancia recorrida por cada una de las bandas (Rf) utilizando el software mencionado anteriormente y se calcularon según la ecuación obtenida para los patrones de pesos moleculares (Figura 1).
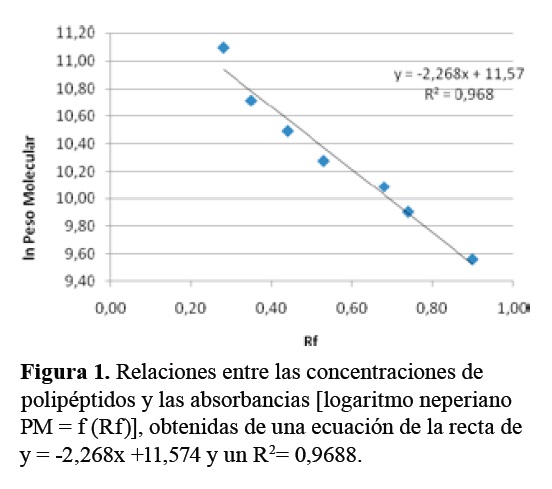
Figura 1. Relaciones entre las concentraciones de polipéptidos y las absorbancias [logaritmo neperiano PM = f (Rf)], obtenidas de una ecuación de la recta de y = -2,268x +11,574 y un R2= 0,9688.
RESULTADOS Y DISCUSIÓN
En el análisis cualitativo realizado mediante electroforesis por SDS-PAGE (Figura 2) a la hora cero, se encontraron proteínas de alto PM: 87; 64; 58; 55; 51; 47; 46; 44; 42; 27 y 14 kDa. En el perfil proteico del pasto obtenido por electroforesis en geles de acrilamida corridos en condiciones desnaturalizantes, se detectaron bandas intensas correspondientes a proteínas de pesos moleculares de 46, 42 y 14 kDa, además de otras de menor intensidad correspondientes a proteínas de 64, 58 y 51 kDa, y un grupo de proteínas de alto peso molecular en la región de 87 kDa. Estas bandas de proteínas de alto peso molecular se repiten en el resto de las muestras tomadas durante todo el proceso de degradación ruminal, valores éstos semejantes a los mencionados por la literatura, dado que en ensayos realizados con varias especies, coincidentemente en todos los forrajes se encontraron bandas en pesos moleculares de 15, 30,
45, 47 y 54 kDa18.
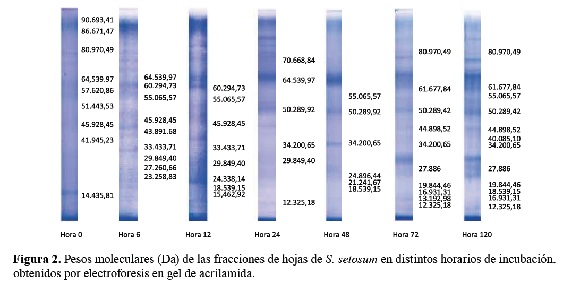
Figura 2. Pesos moleculares (Da) de las fracciones de hojas de S. setosum en distintos horarios de incubación, obtenidos por electroforesis en gel de acrilamida
En general, en el follaje de las gramíneas se observan dos bandas electroforéticas con mayor intensidad, que probablemente sean la RuBisCO (ribulosa-1,5- bisfosfato carboxilasa oxigenasa), proteína con mayor concentración en tejidos verdes y con peso de 56 kDa, y otras proteínas involucradas en procesos de reserva llamadas proteínas vegetativas de almacenamiento foliares (VSP), que se encuentran en las hojas. La masa de estas proteínas oscila entre los 27 y 47 kDa y son muy ricas en aminoácidos esenciales, como valina, lisina, prolina, metionina y triptófano, lo cual también fue observado en leguminosas6.
Luego de seis horas de digestión, se observaron las bandas de 65 y 46 kDa, al igual que en el tiempo inicial, a la vez que aparecieron nuevas proteínas a los 60; 55; 44; 33 (débil); 30, 27 y 23 kDa, así como un grupo debandas muy débiles de bajo peso molecular entre los 19 y 15 kDa. Doce horas después, continuó apareciendo la banda correspondiente a los 46 kDa que fuera detectada en tiempo cero. De las bandas que hicieron su aparición a las 6 horas de degradación, sólo se mantuvieron 4 de ellas y correspondieron a polipéptidos de 60; 55; 33 (débil) y 29 kDa. También se distinguieron nuevas bandas bien definidas de bajo peso molecular: 24; 18 y 15 kDa. A las 24 horas se observó la banda de 29 kDa que fue detectada por primera vez a las 6 horas de digestión, presentándose nuevas bandas a los 71; 64; 50; 34; 26 y 12 kDa.
Posteriormente, a las 48 horas de digestión, se observan las bandas de 50 y 34 kDa que continuaron apareciendo hasta finalizar el ensayo (120 horas). También se presentaron bandas a los 55; 25 y un grupo de bandas entre los 21 y 18 kDa. A las 72 y 120 horas de digestión, se observaron mayores cambios, dado queaparecieron bandas a los 62; 45; 28; 12; 19 y 17 kDa. Estas dos últimas bandas incrementaron su intensidad al avanzar el tiempo de digestión. También se detectaron nuevas bandas a las 120 horas de digestión a los 55; 40 y 18 kDa.
Se detectó la presencia de bandas muy intensas de 46, 41 y 14 kDa, bandas de menor intensidad de 64, 55, 57 y 51 kDa y un grupo de alto peso molecular de 87 kDa. Trabajando con pastos liofilizados, otros autores encontraron bandas entre 20 y 36 kDa18; estos forrajes no difirieron en la concentración nitrogenada y la distribución de proteínas individuales no varió entre cultivares, siendo semejantes a otros pastos, así también se observó que la fracción que más disminuyó fue la de 54 kDa a las 12 horas de degradación.
Las proteínas de 15, 30 y 54 kDa sumaron el 80% de las proteínas separadas electroforéticamente. Las fracciones de 15 y 54 kDa parecieran ser la mayor y menor subunidad de RuBisCO, y la de 30 kDa es la que podría estar asociada con el complejo clorofila-proteína25 del fotosistema 2. Las fracciones solubles de las proteínas de las hojas sumaron del 40 al 50% de las proteínas totales. La RuBisCO así identificada entre ellas representó la mayor proporción de las proteínas solubles totales en alfalfa, el resto fue una mezcla heterogénea de proteínas en el rango de 10 a 300 kDa13.
En las especies C4, del 8 al 23% del total de proteínas correspondió a la enzima RuBisCO. La pérdida de estas fracciones podría ser utilizada como indicador de integridad celular. En presencia de SDS, la proteína RuBisCO se disocia en una fracción con PM de 56 kDa y una subunidad más pequeña de PM de 16 kDa25.
En ensayos realizados con especies C3, se observó que las tasas de degradación ruminal eran diferentes. En ellas las fracciones de 24, 26 y 56 kDa, se localizaron dentro del cloroplasto del mesófilo; en las especies C4 la fracción de 56 kDa estaba localizada en el cloroplasto de las células de la vaina, que eran degradadas más lentamente1, conjuntamente con las fracciones de 24 y 26 kDa que están en el mesófilo de las células de la vaina. La disminución creciente de estas fracciones de proteínas en el tejido degradado de gramíneas megatérmicas, apoya fuertemente la hipótesis que éstas pueden ser protegidas de la degradación ruminal por células de la vaina24.
La fracción de proteína de 56 kDa que al parecer es la subunidad grande de la enzima RuBisCO, estuvo presente en la especie aquí estudiada hasta las 120 h de incubación ruminal; en cambio, la literatura menciona distintos comportamientos para otras pasturas como ser Panicum, donde estuvo presente hasta las 24 h, en Andropogon se la observó hasta las 16 h y en Bromus solo hasta las 8 h. En cambio otra fracción como la de 26 kDa, que fue observada hasta las 24 h en Panicum y Andropogon, coincidentemente con lo observado por nosotros, se detectó solamente por 8 h en Bromus 24 . La fracción de 24 kDa en S. setosum, estuvo presente hasta las 48 h de incubación ruminal, mientras que la literatura cita para Panicum 24 h de incubación, para Andropogon 8 h y para Bromus 4 h24.
En estudios realizados en semillas de girasol se encontró que el patrón electroforético exhibió cinco polipéptidos, en el rango de 65,5 a 42 kDa; esto fue comparado contra un patrón de ocho polipeptidos, dentro del cual se observó la fracción de 55 kDa, que pertenecería a RuBisCO y estaría presente en todos los vegetales 26 . En porotos pallar se registró la presencia de dos fracciones, una que tenía mayor presencia con PM de 14 a 50 kDa y otra con polipéptidos de PM entre 14 y 116 kDa; además, se reportó el hallazgo de varias bandas comunes entre ellas, probablemente debido a una separación incompleta de las fracciones11.
Análisis electroforéticos realizados en Lupinus, mostraron bandas entre 14,4 y 66,2 kDa12. En proteínas de L. angustifolius y L. albus se observaron monómeros con PM que oscilaron entre 17 y 64 kDa9. Estos valores son coincidentes con los hallados en nuestro trabajo, donde el rango encontrado para los diferentes monómeros se ubicó entre los 14 y 64 kDa, a excepción de dos polímeros, uno de ellos que surgió a las 24 h de incubación ruminal de PM de 12 kDa y otro de 86 kDa, que apareció en todos los horarios estudiados.
Otros investigadores sometieron a degradación ruminal tres genotipos de alfalfa y luego, por electroforesis bidimensional, caracterizaron las proteínas individuales en dos tiempos de incubación 45 y 120 min. Después de 45 min, 9 proteínas representaron el 75% del total, 12 proteínas representaron el 50% o menos y otras 5 proteínas fueron intermedias. Después de 120 min 4 proteínas representaron el 80%, 7 se presentaron en el rango de 80 y 50%, y otras 15 representaron menos del 50%7.
En trabajos anteriores se caracterizaron las proteínas solubles de la raíz de Lepidium peruvianum, observándose que para todos los ecotipos, los polipéptidos más abundante eran los de peso molecular bajo19. A su vez, el más prominente de los analizados, ostentaba un PM de 22,5 kDa, en segundo lugar un polipéptido de 17 kDa, y el tercer polipéptido más abundante tenía un PM de 15 kDa, todos ellos distribuidos en un rango de puntos isoeléctricos de 7,1 a 8,2. Si bien este estudio se realizó en raíces y no en hojas como el aquí presentado, los rangos de puntos isoeléctricos coinciden con las observaciones del presente ensayo, no así los PM que variaron dentro de un rango entre 12 a 87 kDa.
En conclusión, si bien la interpretación del patrón de bandas proteicas de S. setosum refleja variación con otras especies, la presencia de bandas observadas evidencia homogeneidad con gramíneas C4. Se observaron modificaciones en el perfil proteico a lo largo del proceso de degradación, detectándose la aparición e intensificación de bandas de menor PM y la aparición de bandas de PM superiores. Durante el proceso de degradación ruminal ensayado, se registró la presencia de bandas que permanecieron durante las 120 h de estudio, lo que permite inferir algún grado de protección que ayudaría a prolongar su permanencia sin ser degradada y por lo tanto una menor digestibilidad de esta gramínea.
1. Akin DE, Burdick D. 1981. Relationships of different histochemical types of lignified cell walls to forage digestibility. Crop Sci 21: 577-581. [ Links ]
2. Araujo C. 2000. WinXGel version 1.7. Facultad de Ciencias Bioquímicas y Farmacéuticas. Universidad Nacional de Rosario, Argentina. [ Links ]
3. Azeez MA, Morakinyo JA. 2004. Electrophoretic characterization of crude leaf protein in Lycopersicon and Trichosanthes cultivars. Afr J Biotech 3: 585-587. [ Links ]
4. Brahim NB, Salhi A, Chtourou N, Combes D, Marrakchi M. 2002. Isozymic polymorphism and phylogeny of 10 Lathyrus species. Gen Res & Crop Evol 49: 427-436. [ Links ]
5. Camacho MK et al. 2010. Caracterización proteómica de granos de frijol azufrado (Phamseolus vulgaris) cultivados en el estado de Sinaola. Ra Ximhai 6: 23-36. [ Links ]
6. Carvajal JI. 2010. Digestibilidad in vitro prececal y cecal de plantas forrajeras tropicales en nutrición de cerdos. www.bdigital.unal.edu.co/2190/1/7406002.2010 pdf [ Links ]
7. Chen ZZ, Huang J, Lai Z, Fan B. 2009. Biosynthesis of salicylic acid in plant. Plant Sign & Behav 4: 493-496. [ Links ]
8. Díaz P, Dallarizza M, Vázquez D, Castro M. 2006. Elementos de análisis cualitativo y cuantitativo en proteínas del gluten de trigo. Agric Técn (Chile) 66: 360-369. [ Links ]
9. Duranti M, Consonni A, Magni C, Sessa F, Scarafoni A. 2008. The major proteins of lupin seed: characterization and molecular properties for us as functional and nutraceutical ingredients. Trends Food Sci Technol 19: 624-633. [ Links ]
10. Fahmy WG, Aufrere J, Graviou D, Demarquilly C, El- Shazly K. 1991. Comparison between the mechanism of protein degradation of two cereals by enzymatic and in situ methods, using gel electrophoresis. Anim Feed Sci Technol 35: 115-130. [ Links ]
11. Gallego S, Pacheco J, Betancur D, Guerrero CL. 2004. Extracción y caracterización de las fracciones proteínicas solubles del grano de Phaseolus lunatus. Arch Latinoam Nutr 54: 78- 82. [ Links ]
12. Garzón P et al. 2008. Chemical physical properties of globulin and 1-congluten isolated at different pH values from Lupin albus. 12th Intern Lupin Conf. Fremantle, Australia. www.lupins.org/pdf/conference/2008/FoodandHealth.pdf [ Links ]
13. Grum DE, Shockey L, Weis WP. 1991. Electrophoretic examination of alfalfa silage proteins. J Dairy Sci 74: 146. [ Links ]
14. Illoh HC. 1990. An electrophoretic study of crude protein diversity in the seed of the genus Amaranthus. Nig J Botany 3: 151-158. [ Links ]
15. Isshiki S, Umezaki T. 1997. Genetic variations of isozymes in cultivated sesame. Euphytica 22: 375-377. [ Links ]
16. Laemmli UK. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature (London) 227: 680-685. [ Links ]
17. Messman MA, Weiss WP. 1994. Use of electrophoresis to quantify ruminal degradability of protein from concentrate feeds. Anim Feed Sci Technol 49: 25-35. [ Links ]
18. Messman MA, Weiss WP, Koch ME. 1994. Changes in total and individual proteins during harvest, storage, and ruminal fermentation of forages. J Dairy Sci 77: 492-500. [ Links ]
19. Monteghirfo M, Yarleque A. 2007. Caracterización de las proteínas totales de tres ecotipos de maca (Lepidium peruvianum G. Chacon) mediante electroforesis unidimensional y bidimensional. An Fac Med 68: 301-306. [ Links ]
20. Morakinyo JA. 1984. Genetic studies in the genus Sorghum. Tesis, Univ of Ife, Lle-Ife, Nigeria. http://www.nuc.edu.ng/nucsite/File/ILS%202003/ILS-120.pdf [ Links ]
21. National Research Council. 1985. Ruminant Nitrogen Usage. Natl Acad Sci Washington. http://www.nap.edu/openbook.php?isbn=030903597X
22. Onokpise OV. 1988. Coefficients of co-ancestry and in inbreeding of commoly grown muscadine grape cultivars. Amer J Enol and Vitic 39: 351-353. [ Links ]
23. Perez S, Escala M, Tillett S. 2007. Evaluación preliminar del patrón electroforético en una dimensión (SDS-PAGE) de proteomas de semillas en especies del género Passiflora
L. Passifloraceae. Rev Fac Agron LUZ (Venezuela) 24: 13-19. [ Links ]
24. Redfearn DD, Moser LE, Waller SS, Klopfenstein TJ. 1995. Ruminal degradation of Swithgrass, Big bluestem, and Smooth Bromegrass leaf portions. J Anim Sci 73: 598- 605. [ Links ]
25. Rutner AC, Lane MD. 1967. Nonidentical subunits of ribulose diphosphate carboxylase. Biochem Biophys Res Commun 28: 531-537. [ Links ]
26. Sammour RH, El-Shourbay MN, Abo-Shady AM, Abasery AM. 1995. Proteins of cottonseed (Gossypiun barbadense). Extraction and characterization by electrophoresis. Qatar Univ Sci J 15: 77-82. [ Links ]
27. Sirtori E, Resta D, Brambilla F, Zacheri C, Arnoldi A. 2010. The effects of various processing conditions on a protein isolate from Lupinus angustiolius. Food Chem 120: 496-504. [ Links ]
28. Ullah F, Nosheen A, Hussain I, Bano A. 2009. Base catalyzed transesterification of wild apricot Kernel oil for biodiesel production. Afr J Biotech 8: 3309-3313. [ Links ]
29. Wencomo HB, Díaz M, Álvarez A, Mora L, Coto O. 2008. Estudio del polimorfismo isoenzimático en el género Leucaena. Zoot Trop 26: 327-331. [ Links ] Revista Veterinaria.