Introducción
Scomber japonicus peruanus (Jordán & Hubbs, 1925), es una especie cosmopolita que en Perú y otros países se conoce como caballa. Habita en ambientes relativamente cálidos, a temperatura del agua entre 14°C y 23°C, y salinidad de 34,9%o a 35,2%o. En el Perú es la tercera especie más desembarcada1,2.
Gran parte de su captura se procesa como salado y se comercializa en lugares donde existe fuerte demanda, como en Tumbes, donde además de ser vendida en el mercado local, también se exporta al Ecuador, lugar donde es muy apreciada. La caballa salada se comercializa sin envasar por lo que está expuesta a contaminantes físicos, químicos y biológicos, con lo cual dejaría de ser apto para el consumo humano, según las exigencias de calidad del Codex Alimentarius3.
El salazonado de los alimentos cárnicos como el pescado es un método para conservarlos durante periodos más prolongados para su consumo, método que se ha utilizado desde tiempos remotos4. Tiene como objeto asegurar la penetración de la sal en el músculo para lograr deshidratar o eliminar agua intracelular del producto5, puesto que la sal introducida en el músculo del pescado ayuda a preservarlo de los procesos de descomposición bacteriana, así como retardar la actividad enzimática, ya que reduce la actividad del agua del pescado 2.
En general los alimentos son perecederos, por lo que necesitan ciertas condiciones de tratamiento, conservación y manipulación, siendo la principal causa de su deterioro el ataque por diferentes tipos de microorganismos (bacterias, levaduras y mohos)6,7,8. Los pescados medianamente salados presentan mejor resistencia al ataque de bacterias y pueden -al igual que los salpresos- absorber agua y aumentar de peso cuando son remojados para su desalado 2.
Existen bacterias halotolerantes así como halófilas que se desarrollan en los productos salados. El predo minio de determinado grupo de bacterias depende de varios aspectos tales como pH, temperatura, presión, oxígeno 9.
En pescado salado, se han indicado bacterias como: Staphylococcus aureus, Salmonella sp., Escherichia coli, Streptococcusfaecalis y Streptococcusfaecium10. Por su parte, en sardina salada-seca procesada artesanalmente, se reporta la presencia de Staphylococcus aureus, Salmonella sp. y Escherichia coli11, mientras que Pseudomonas salinaria y Pediococcus halophylus, se mencionan en pescado seco salado 9.
Para el Instituto Tecnológico Pesquero del Perú (ITP) 12, los requisitos para un buen salado de pescado son: humedad (43 a 52%) y cloruro de sodio (18 a 22%). Asimismo, se clasifica la calidad de la caballa de acuerdo a sus características organolépticas.
La identificación de bacterias que podrían hallarse en productos salados se puede realizar secuenciando su fragmento del gen 16S ARNr. Este gen es una sección del ADN que codifica para un polirribonucleótido de unos 1500 nt, a partir de cuya secuencia se puede obtener información filogenética y taxonómica, que conduce a conocer la secuencia de nucleótidos que la conforman,
Dicha secuencia es comparada contra bases de datos genómicas, por ejemplo GenBank, los cuales acumulan millones de secuencias de ADN para diferentes genes y organismos, a fin de poder identificar la secuencia más similar que corresponda a la especie problema 13,14.
Material y métodos
Se adquirieron 50 ejemplares de caballa salada del mercado de abastos de Tumbes, Perú, trasladándose a un depósito estéril y aséptico en la Facultad de Ingeniería Pesquera y Ciencias del Mar de la Universidad Nacional de Tumbes.
1. Determinación de humedad y cloruros
Humedad. Con una balanza digital (Sartorius TE2145 con precisión ±0,0001 g), se pesó 5 g del músculo de cada ejemplar de caballa y se llevó a estufa a 105°C por 3 a 4 horas. La muestra se retiró de la estufa y se colocó en un desecador hasta que se enfriara. El porcentaje de humedad se obtuvo con la fórmula:
Cloruro de sodio. Con la balanza digital Sartorius se procedió a pesar 2 g del músculo de cada ejemplar de caballa. A continuación, la muestra fue triturada en un vaso de precipitación agregándosele 100 ml de agua destilada para homogenizar el contenido, tratando de que el músculo se disgregue completamente para liberar el volumen de NaCl, filtrándose el contenido en un filtro whatman, para separar el músculo triturado del líquido, dejándose decantar por un minuto.
Se tomó 10 ml del líquido y se diluyó en 100 ml de agua destilada (dilución 1:10, factor dilución f=1/10), luego con un dosificador, se agregó una gota de solución al 1% de fenolftaleína. Si la muestra quedó en color rosa, estuvo lista para titular, caso contrario si quedó transparente se debió agregar gota a gota solución 0,1 N de NaOH hasta que la solución se tornara ligeramente rosa. Finalmente se agregó una gota de solución de cromato de potasio al 5%. La solución debió tornarse a un tono amarillo.
Una bureta se cargó con una solución de 0,1 N de AgNO3, hasta un valor exacto en la escala de la bureta (volumen inicial Vi). En un vaso de precipitados se colocó la muestra liquida con NaCl obtenido del músculo del pescado salado y se puso debajo de la bureta que pendía de un soporte universal.
Luego con la bureta se agregó gota a gota la solución de AgNO3, observándose primero la formación de un precipitado blanquecino de cloruro de plata (AgCl) y finalmente la titulación terminó al producirse un precipitado color rojo ladrillo de cromato de plata (Ag2CrO4). En la escala de la bureta se anotó el volumen final (Vf). El gasto (G) se determinó por diferencia entre el volumen final e inicial (Vf - Vi). Para obtener la concentración de NaCl se utilizó la siguiente fórmula:
2. Aislamiento de cepas bacterianas
De cada caballa se extrajo una muestra del músculo de entre 0,5 y 1 g, colocándose cada muestra en un tubo falcon de 15 ml. Las muestras se homogenizaron utilizando un macerador metálico, agregándose 5 ml de solución salina al 10%.
A partir de esta mezcla y sin realizar diluciones, se sembraron por barrido, inóculos de 100 pl en placas petri con agar tripticase soya (TSA), incubándose a temperatura ambiente por 24 horas. De cada una de las colonias obtenidas se tomó una muestra y se volvió a sembrar en placas con agar TSA a fin de obtener una cepa más purificada.
Una vez obtenidas, las cepas purificadas se guarda ron en medio sólido de agar TSA para posteriormente poder realizar la extracción del ADN de las diferentes cepas que se obtuvieron.
3. Secuenciamiento de bacterias en el músculo de caballa usando un fragmento del gen 16S ARNr
Extracción del ADN. Se tomó una placa de petri en la que se cultivó en TSA con 15% de NaCl, una cepa bacteriana aislada de la caballa, la que se dejó crecer por 72 h. Se tomó un microtubo de centrífuga y se taró, luego se retiraron las colonias bacterianas de la placa de petri y se transfirieron al tubo, hasta que se obtuvo una masa de colonias con peso entre 14 a 20 mg.
Seguidamente se añadió al microtubo, 800 pl de solución fisiológica (0,85% de NaCl) y se suspendieron las colonias bacterianas en la misma. Se centrifugó a 10 000 rpm por 2 min, luego se retiró el sobrenadante invirtiendo el microtubo. A continuación, se añadió al microtubo 500 pl de solución de buffer de fosfato salino (PBS) 1X, nuevamente se resuspendieron las colonias en dicha solución, posteriormente se llevó a centrifugación a 10 000 rpm por 2 min retirándose el sobrenadante por inversión del microtubo
Posteriormente se añadió 200 pl de solución de tris EDTA (TE) 1X, nuevamente se resuspendieron las colonias en dicha solución para lo cual se hizo uso de un vórtex. El microtubo fue llevado a baño maría, a temperatura de ebullición (95 a 100 °C), por 10 min, luego se colocó en congelación a -20°C por 5 min.
El microtubo se llevó a centrifugación a 10 000 rpm por 2 min. Finalmente, se tomó 10 pl del sobrenadante y se llevó a un nuevo microtubo de centrífuga y se le añadió 90 pl de agua ultrapura. Este microtubo fue llevado a congelación a -20°C en un congelador.
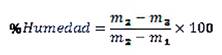
Tabla 1 Cálculo de volumen de reactivos para la preparación del mix necesario para la realización de la PCR xincluye dNTPs, pero no incluye MgCl2(cat N° S113).
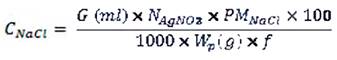
Tabla 2 Porcentaje de humedad y NaCl del músculo de los ejemplares de caballa en los meses muestreados Humedad y NaCl figuran en promedio ± desvío estándar.
Amplificación del fragmento del gen 16S ARNr. Se usaron los iniciadores 27F (5’-AGAGTTT- GATCMTGGCTC-3’) y 1492R (5'-GGTTACCTTG- TTACGACTT-3'), los cuales producen una amplificación de 1465 pb 15. La amplificación se realizó preparando una solución mix para realizar la reacción de PCR, teniendo en cuenta el número de cepas bacterianas a las cuales se les extrajo el ADN. A estos, se les adicionó una reacción más para el control negativo y otra para el control positivo (Tabla 1).
La amplificación del ADN se realizó en un termociclador (marca Applied Biosystems modelo SimpliAmp termal Cycler) que fue programado basado en el protocolo utilizado por Pérez 17, que consiste en: i) predesnaturalización a 95°C por 1 min; ii) 30 ciclos de las siguientes etapas: 94°C por 30 s (desnaturalización), 50°C por 40 s (hibridación) y 68°C por 30 s (polimerización); e iii) polimerización final a 68°C por 7 min.
Para la verificación de la amplificación, se hizo migrar los amplicones en geles de 120 ml de agarosa al 2% conteniendo 5 pl de bromuro de etidio, se usó como tampón de migración TAE 1X. En los pozos de dichos geles se colocó 1 pl de tampón de depósito y 9 pl de cada amplicón. La migración se realizó a 68 V durante 45 min.
Conjuntamente se hizo migrar un marcador de peso molecular de 100 a 3000 pb. Los geles fueron visualizados utilizando un transiluminador (marca Spectroline, modelo 3494-3123).
Identificación de cepas bacterianas aisladas de caballa salada. La bacterias aisladas del múscu lo de caballa fueron identificadas mediante búsqueda de secuencias similares en la base de datos GenBank, siendo el criterio para identificar la cepa a nivel de especie, al menos 99% de similitud con la secuencia comparada; mientras que para identificar a nivel de género, que tenga al menos una similitud de 97%.
Resultados
Porcentaje de humedad y cloruro de sodio en el músculo de los ejemplares de caballa salada. El porcentaje promedio de la humedad del músculo de los ejemplares de caballa tuvo un valor mínimo de 32,13% y un máximo de 70,94%, mientras que el porcentaje de NaCl estuvo entre 11,87 y 15,31%, con un rango de concentraciones entre 5 y 25,5%. Ambos se muestran en la Tabla 2.
Identificación de cepas bacterianas aisladas de caballa salada. La Tabla 3 muestra las especies bacterianas que se identificaron mediante la búsqueda de secuencias similares en la base de datos GenBank, determinándose 13 cepas de las que fue posible obtener secuencias de ADN.
Las cepas que fueron posibles de identificar pertenecieron a sólo tres géneros: Staphylococcus (siete cepas), Salinivibrio (dos cepas) y Halomonas (una cepa). Sólo cuatro cepas pudieron ser identificadas a nivel de especie, todas ellas del género Staphylococcus, siendo ellas: S. equorum (tres cepas) y S. nepalensis (una cepa).
Discusión
Las cepas bacterianas que fueron identificadas mediante secuenciación y búsqueda en la base de datos Genbank correspondieron a géneros y especies reportados previamente como flora bacteriana halófila, como: Staphylococcus sp., Staphylococcus nepalensis, Staphylococcus equorum , Salinivibrio sp. y Halomonas sp.16,17 han reportado presencia de varias cepas de Staphylococcus sp., entre ellas S. nepalensis y S. equorum en corvinas saladas y fermentadas en China, así como en mariscos salados en Corea, siendo por tanto especies que forman parte de alimentos con alto contenido de sal.
Adicionalmente, en una guía de calidad de caballa salada, se señala que las bacterias antes indicadas forman parte de la flora de este producto 12. Por su parte, se menciona que en alimentos fermentados y con alto contenido de sal, se identificaron Staphylococcus equorum como especie dominante 18. Esta bacteria productora de aminas, también se ha detectado con frecuencia en productos cárnicos fermentados con alto nivel de sal en Europa.
Entre las aminas biogénicas, la histamina y la tiramina son peligrosas para la salud humana debido a sus propiedades vasoactivas y psicoactivas, aunque ninguna de las enzimas implicadas en la descarboxilación de histidina y tiramina fueron identificadas en los genomas de las cepas de S. equorum.
Por otra parte, Halomonas sp. han sido reportadas como parte de la flora bacteriana de mariscos salados en Corea 17 , mientras que Salinivibrio sp. ha sido encontrada como flora bacteriana de productos de pescado salado 19. También se afirma que Salinivibrio es un género bacteriano capaz de vivir en concentraciones de NaCl de 0,5 a 20% 20, lo que le faculta a desarrollarse en ambientes hipersalinos y en carnes saladas.
En resumen, todas las especies bacterianas identificadas en esta investigación corresponden a géneros presentes en productos con alto contenido de sal. En esta investigación, la humedad porcentual promedio se encontró en 62,73±3,02% en junio; 62,72±10,34% en agosto y 47,7±5,53% en septiembre, coincidiendo con lo mencionado por quienes refieren que un buen pesca do salado debe contener entre 45 y 52% de humedad 12.
En el caso de este trabajo, solo 15 de los 50 ejemplares analizados (30%) estuvieron dentro del rango manifestado por investigadores 12, con lo cual el 70% restante no tuvo una humedad adecuada, lo cual genera que dichos productos reduzcan su vida en anaquel.
En el presente trabajo la concentración de NaCl en el músculo de caballa salada estuvo entre de 11,87±4,67% y 15,31±3,01%, valores inferiores al recomendado por el ITP (2001) quien indica que un buen músculo de pes cado salado debe contener entre 18% y 22% de ClNa. Ello difiere contra quienes manifiestan que al 15% de NaCl no hay crecimiento de Staphylococcus nepalensis 21 , mientras que hasta un 10% sí lo hay.
En conclusión, mediante la secuenciación de un fragmento del gen 16S ARNr y la búsqueda en la base de datos Genbank, se determinaron las especies: Staphylococcus sp, Staphylococcus equorum, Staphylococcus nepalensis, Salinivibrio sp y Halomonas.