Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Boletín de la Sociedad Argentina de Botánica
versión On-line ISSN 1851-2372
Bol. Soc. Argent. Bot. vol.57 no.4 Córdoba dic. 2022
http://dx.doi.org/10.31055/1851.2372.v57.n4.38230
10.31055/1851.2372.v57.n4.38230
Genética y evolución
Variabilidad del acervo génico y parámetros germinativos en poblaciones naturales de Enterolobium contortisiliquum (Leguminosae) del Noreste Argentino
Gene pool variability and germinative parameters in natural populations of Enterolobium contortisiliquum (Leguminosae) from Northeastern Argentina
Carla G. Martinotto
María E.Barrandeguy
Alejandra L.Goncalves
María V.García
1. Universidad Nacional de Misiones. Facultad de Ciencias Exactas, Químicas y Naturales. Posadas, Misiones, Argentina
2. Instituto de Biología Subtropical - Nodo Posadas (UNaM - CONICET).
3. Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET - Argentina).
4. Instituto de Botánica Darwinion, Labardén 200, San Isidro, Buenos Aires, Argentina.
*vgarcia@fceqyn.unam.edu
Summary
Background and aims: Enterolobium contortisiliquum is a South American native forest tree. Gene pool variability and germinate variables were estimated in populations from Northeastern Argentina in order to analyze the relationship among gene flow, mating system, and dispersal.
M&M: Individuals from Eldorado and Ituzaingó populations and seeds from Ituzaingó were genotyped by means of five and four nuclear microsatellite loci, respectively. Germinate variables were determined while genetic diversity and population structure were characterized. Indirect gene flow and pollen-mediated gene flow were estimated. Nuclear genetic variability differentiation at locus and gene pool levels was determined.
Results: The analyzed populations showed high genetic diversity. At least one or two pollen donor trees by fruit were detected. Also, the absence of population genetic structure as a consequence of high pollen and seed-mediated gene flow was detected. The seeds showed the highest genetic differentiation at locus and gene pool levels.
Conclusions: The high gene flow levels detected contribute to maintaining the gene pool variability while the high genetic diversity, the absence of both inbreeding and population genetic structure together with pollen-mediated gene flow support allogamy as the main mating system in the analyzed populations.
Key words: Genetic structure, genetic variability, germination, microsatellites, pollen donors, timbó.
Resumen
Introducción y objetivos: Enterolobium contortisiliquum constituye un recurso forestal nativo sudamericano. En el presente trabajo se estimó la variabilidad del acervo génico y se evaluaron variables germinativas en poblaciones del Noreste argentino de esta especie para analizar la relación entre el flujo génico, el sistema de fecundación y la dispersión.
M&M: Se genotipificaron individuos de las poblaciones Eldorado e Ituzaingó y semillas de Ituzaingó mediante cinco y cuatro loci microsatélites nucleares, respectivamente. Se determinaron variables germinativas, se caracterizó la diversidad genética y la estructura genética poblacional, se estimaron los niveles de flujo génico de manera indirecta, se identificó la presencia de alelos heredados vía polen y se determinó la representatividad de la variabilidad genética nuclear a nivel de locus y de acervo génico.
Resultados: Las poblaciones analizadas presentaron elevada diversidad genética. Se detectó el aporte, como mínimo, de uno o dos árboles donantes de polen por fruto y ausencia de estructura genética poblacional como consecuencia de elevados niveles de flujo génico mediado por polen y semillas. Las semillas presentaron la mayor diferenciación respecto a su complemento tanto a nivel de locus como de acervo génico.
Conclusiones: Los elevados niveles de flujo génico detectados contribuyen al mantenimiento de la variabilidad del acervo génico mientras que, la detección de elevados niveles de diversidad genética, valores nulos del coeficiente de endogamia, ausencia de estructura genética y alelos heredados vía polen a partir de una fuente externa a las poblaciones estudiadas, respaldan que la alogamia es el sistema de fecundación dominante en ellas.
Palabras clave: Donantes de polen, estructura genética, germinación, microsatélites, timbó, variabilidad genética.
Introducción
El conocimiento de los procesos que configuran la diversidad genética poblacional a lo largo de las generaciones constituye uno de los aspectos fundamentales en el manejo sostenible de los recursos genético-forestales. En ecosistemas boscosos fragmentados, la pérdida de diversidad genética en poblaciones aisladas de especies forestales se produce a una tasa mayor que en poblaciones continuas (Sork & Smouse, 2006). Sin embargo, las poblaciones de especies leñosas que presentan polinización mediada por animales pueden resultar menos susceptibles a los efectos de la fragmentación del hábitat (Breed et al., 2015) lo cual, sumado a características tales como la longevidad de los individuos, la elevada diversidad genética dentro de las poblaciones y las posibles tasas elevadas de flujo génico mediado por polen, confieren a las especies forestales la capacidad de hacer frente a las consecuencias negativas de la fragmentación (Andrianoelina et al., 2009). En especies forestales con elevados niveles de flujo génico mediado por polen, los límites de una población no son sencillos de ser identificados, dado que cualquier individuo que aporte polen a la población se convertirá en miembro de la misma, siendo el tamaño poblacional potencialmente mayor que el representado por el conjunto de árboles del cual se puedan colectar semillas (Hattemer, 2005). Esta característica de las poblaciones de especies forestales demanda inferir el número de posibles donantes de polen que contribuyen al acervo génico poblacional mediante el análisis de estadios tempranos (semillas). De esta manera, el análisis de la variabilidad genética de los individuos adultos y de los posibles donantes de polen mediante el análisis de las semillas permite establecer la contribución relativa de cada uno de estos estadios al acervo génico de la población en estudio, ya que la estimación de su variabilidad genética brinda información de utilidad para la sostenibilidad de los recursos genéticos a través del tiempo (Gillet et al., 2005).
Enterolobium contortisiliquum (Vell.) Morong, "timbó", "oreja de negro" o "pacará", es la única especie del género presente en Argentina. Su distribución geográfica es disyunta y está fuertemente asociada a los Bosques Secos
Estacionales Neotropicales de Sudamérica -BSEN - (Prado, 2000). Es abundante en las Selvas de Transición de la Provincia Fitogeográfica de las Yungas (Cabrera, 1976), consideradas como parte del Núcleo Pedemontano de los BSEN (Prado, 2000). Es común en las Selvas Marginales de la Provincia Fitogeográfica Paranaense (Cabrera, 1976), unidad perteneciente al Núcleo Misiones de los BSEN (Prado, 2000). También ocurre en bosques en galería dentro de la Provincia Chaqueña (Cabrera, 1976) que se corresponden a los llamados Bosques de Transición Austro-Brasileños del Núcleo Misiones (Prado, 1993), donde crece sobre llanuras aluviales de los principales ríos (Morello & Adámoli, 1974). Se trata de árboles de fuste recto y tronco de hasta 30 m de altura y 1,60 m de diámetro en las formaciones densas (Fig. 1A), siendo sus hojas alternas y bipinnadas (Tortorelli, 2009; Hoc et al., 2015) (Fig. 1B). Sus flores son blancas, a veces algo verdosas y se disponen en un racimo doble piramidal. Cuando se produce la antesis se distinguen dos morfos florales: flores perfectas (donantes y receptoras de polen) y flores estaminadas (donantes de polen), presentando ambos morfos las anteras abiertas con políadas expuestas y un nectario anular prominente en la base del tubo estaminal (Fig. 1C). De esta manera, la inflorescencia constituye una unidad de atracción de polinizadores tales como abejas de tamaño mediano (Hoc et al., 2015) y polillas, las cuales pueden transportar grandes cargas de polen, visitar inflorescencias de un elevado número de árboles y cubrir grandes distancias (Moreira et al. 2015). Los frutos son de tipo legumbre bacoide, simples, secos, de color negro en la madurez, indehiscentes y polispérmicos, presentando en promedio entre 12 y 15 semillas (Fig. 1D). Las semillas son aplanadas y oblongas, con testa lisa, córnea, bicolor, glabra, poco pulida a opaca, de color marrón oscuro (Barretto & Ferreira, 2011) (Fig. 1E). Los frutos constituyen la unidad de dispersión tanto vía endozoocoria, mediante la intervención de mamíferos silvestres y domésticos, que ingieren y deponen grupos de semillas (Abraham de Noir et al., 2002), como vía hidrocoria, principalmente en bosques en galería asociados a ríos o en relieves con pendiente que favorecen el movimiento de los propágulos (Gentry, 1993; Hunter, 1989).
Los estudios genético-poblacionales que incluyen variables asociadas a la germinación y al establecimiento de las plántulas aportan información relevante para el manejo sostenible y la conservación de las poblaciones de especies forestales nativas, en las cuales la dormición de las semillas es una característica frecuente. La dormición física de las semillas de E. contortisiliquum se produce por la impermeabilidad del tegumento (Abraham de Noir et al., 2002), con lo cual, tratamientos pre-germinativos de escarificación pueden ser utilizados para inducir la germinación, simulando procesos que ocurren de manera natural, como por ejemplo el recorrido de las semillas por el tracto digestivo de los mamíferos (Alexandre et al, 2009). Los estudios de germinación son relevantes para evaluar los efectos de los cambios en el uso del suelo sobre la permanencia de las poblaciones. Ha sido descripto que los frutos de E. cyclocarpum provenientes de árboles que habitan áreas de pasturas producen en promedio un menor número de semillas, las cuales a su vez presentan menor vigor, que los frutos de árboles de bosques continuos de los BSEN, en los que se ha registrado un menor tiempo de germinación de semillas (Rocha & Aguilar, 2001a). Del mismo modo, estudios recientes en E. contortisiliqumm revelaron que el tiempo de germinación es afectado por el disturbio del hábitat, detectando un tiempo medio de germinación menor en áreas preservadas de los BSEN en relación a áreas con impacto antrópico (Moreira et al., 2021).

Fig. 1. Características morfológicas de Enterolobium contortisiliquum. A: Individuo adulto y detalle de la corteza; B: Hoja compuesta bipinnada; C: Inflorescencia; D: Fruto; E: Semillas. Escala= A: 1 m; B-D: 1 cm; E: 0,5 cm. Imágenes de propia autoría y adaptadas de Ilustraciones de Museo Botánico (FCEFyN - UNC), de Flora Argentina, Plantas Vasculares de la República Argentina (www.floraargentina.edu.ar) y de Argentina nativa (https://faunayfloradelargentinanativa.blogspot.com/2011/02/timbo-enterolobium-contorstiilquum.html).
Diferentes estudios genético-poblacionales desarrollados en poblaciones de E. contortisiliquum localizadas en Brasil central han detectado diversidad genética elevada mediante el empleo de marcadores microsatélites nucleares (SSRnu) y estructura genética poblacional moderada empleando marcadores ISSR, la cual ha sido explicada como consecuencia de la presencia de barreras geográficas que limitarían el flujo génico (Moreira et al., 2012; Moreira et al, 2015).
En el presente trabajo, la estimación de la variabilidad genética intra e interpoblacional y la evaluación de variables germinativas de E. contortisiliquum en poblaciones del Noreste argentino permitió analizar la relación entre el flujo génico, el sistema de fecundación y la dispersión. Atendiendo tanto a la biología reproductiva de la especie como a la limitada movilidad de los propágulos, que incluye dispersión de semillas por endozoocoria y polinización mediada por insectos, la hipótesis de este trabajo plantea que las poblaciones analizadas presentan estructura genética poblacional como consecuencia de flujo génico restringido. Así, para analizar la variabilidad del acervo génico de E. contortisiliquum se consideraron individuos adultos y semillas provenientes de dos poblaciones naturales del Noreste argentino con el objeto de determinar variables germinativas, caracterizar la variabilidad genética de los diferentes estadios, determinar la estructura genética y la representatividad de la diversidad genética poblacional y estimar de manera indirecta el flujo génico entre las poblaciones estudiadas.
Materiales y Métodos
Sistema de estudio
El área de estudio se situó en el Noreste argentino en la Provincia Fitogeográfica Paranaense del dominio Amazónico (Cabrera, 1971; Cabrera, 1976). En esta región, se analizaron individuos de la localidad Eldorado, ubicada en el Noroeste de la provincia de Misiones, y de la localidad Ituzaingó ubicada en el Noreste de la provincia de Corrientes (Fig. 2). La distancia lineal que separa ambas poblaciones es de aproximadamente 250km. Los individuos fueron geo-referenciados mediante el sistema de posicionamiento global (GPS) mediante un receptor Garmin eTrex® 20* (precisión de ± 3m). Se analizó material vegetativo de 28 individuos adultos de E. contortisiliquum (18 individuos de Eldorado y 10 de Ituzaingó), como así también tejido embrionario radicular obtenido desde tres semillas de dos frutos maduros cosechados a partir de cinco árboles madre de Ituzaingó. Se unificó el material procedente de tres semillas por fruto a los efectos de contar con un volumen de tejido adecuado para la extracción de ADN.
Análisis de variables germinativas
Se extrajeron las semillas de los frutos maduros colectados en Ituzaingó y se registró el número de semillas por fruto descartándose las semillas vanas y/o dañadas. Las semillas viables fueron almacenadas a temperatura ambiente en bolsas de papel al resguardo de la humedad y la luz hasta el establecimiento de los ensayos de germinación.
Se establecieron sendos ensayos de germinación a partir de semillas con y sin tratamiento pregerminativo. El ensayo con semillas pre-tratadas se estableció a partir de semillas sometidas a escarificación mecánica empleando papel de lija, mediante el cual las semillas fueron pulidas en el área opuesta al embrión hasta dejar expuesto el cotiledón. Los ensayos se llevaron a cabo a temperatura ambiente (entre 22 y 26°C) y un fotoperíodo de 12h según lo recomendado por Lozano et al. (2016). Se registró el número de semillas germinadas por árbol madre mediante observaciones diarias siguiendo como criterio de germinación la emergencia radicular (Lozano et al., 2016). Los ensayos se mantuvieron hasta cinco días después de observar el último evento de germinación, es decir hasta que la totalidad de los tratamientos alcanzó un número constante de semillas germinadas según lo recomendado por Cruz-Silva & Rosa (2011).
![]()
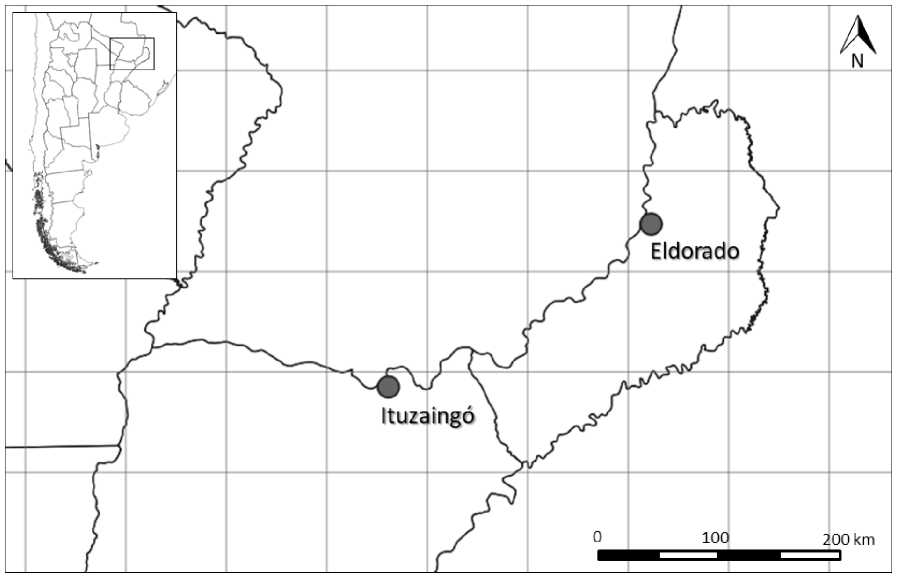
Fig. 2. Localización geográfica de las poblaciones de Enterolobium contortisiliquum analizadas.
Para cada árbol madre se consideraron variables germinativas mediante la estimación de los siguientes parámetros propuestos por Lozano-Isla et al. (2019): Germinabilidad (G): porcentaje de semillas que completan el proceso de germinación:
G= ioo
donde n., es el número de semillas germinadas en el z'-ésimo tiempo y N el número total de semillas en cada unidad experimental, es decir semillas con y sin tratamiento pre-germinativo. Tiempo medio de germinación (t): se calcula como la media ponderada' del tiempo de germinación (hora, día u otra unidad de tiempo):
T _ Zt=l njtj
S?= i"¡
donde t.es el tiempo desde el inicio del experimento a la /'-ésima observación; es el número de semillas germinadas en el /'-ésimo tiempo y k corresponde a la última observación. Coeficiente de variación del tiempo de germinación (CVt): indica la uniformidad de la germinación o la variabilidad en relación con el tiempo medio de germinación: 100
donde St es la desviación estándar del tiempo de germinación. Tasa media de germinación: (V):
![]()
![]()
ft =
Sí : «i
corresponde al recíproco del tiempo medio de germinación: _ _ i
í
índice de sincronización (Z): corresponde al grado de superposición en el tiempo de genninación:
^ _ H £?11,2
_ N
siendo: y
![]()
donde C , _ es la combinación de semillas
«1,2
germinadas en el /'-ésimo tiempo. La sincronía de germinación de una semilla con otra supone Z=1. Índice de incertidumbre (U): esta medida es una adaptación del índice de Shannon y mide el grado de incertidumbre asociado a la distribución de la frecuencia relativa de germinación: siendo donde f corresponde a la frecuencia relativa de germinación, al número de semillas germinadas en el /'-ésimo tiempo y k al último día de germinación. La comparación múltiple de medias de los parámetros de germinación de semillas se realizó empleando la prueba Student-Newman-Keuls (SNK) con un nivel de significancia estadísticap < 0,05. Las estimaciones se realizaron empleando el paquete de R GerminaR mediante la aplicación web GerminaQuant for R (Lozano-Isla et al, 2019).
Anál/s/s de var/ables genét/cas
Los individuos adultos de ambas poblaciones fueron genotipificados empleando cinco loc/ microsatélites nucleares (SSRnu) específicos de Enterolob/um cyclocarpum (Jacq.) Griseb.: Ency-4, Ency-8, Ency-17, Ency-24, Ency-33 (Peters et al, 2008), transferidos a E. contort/s/l/quum por Moreira et al. (2012), mientras que el tejido embrionario fue genotipificado empleando cuatro de estos SSRnu (Ency-4, Ency-8, Ency-24 y Ency-33), debido a la elevada proporción de datos perdidos en el locus Ency-17. Las reacciones de amplificación se realizaron en un volumen final de 14pl empleando 0,5ng/pl de ADN genómico; 1X de buffer KCl; entre 1,12-1,40mM de MgCl2 en función del locus analizado; 1,25mM de cada dNTP; 1U de Taq polimerasa; entre 0,33-0,60pM del cebador reverse, entre 0,20-0,40pM del cebador forward en función del locus analizado y 0,2 pM del cebador universal M13 marcado con un fluoróforo (FAM o HEX) en su extremo 5'.
La genotipificación de los individuos y del tejido embrionario se realizó mediante el método de los tres cebadores propuesto por Schuelke (2000) modificado por Goncalves et al. (2013). La genotipificación de los amplicones se llevó a cabo por medio de electroforesis capilar en un secuenciador automático ABI 3730 XL (Macrogen, Inc., Corea del Sur) empleando el marcador de peso estándar GS500-ROXTM (Applied Biosystems®). Los diferentes alelos fueron identificados y codificados a partir del tamaño de los fragmentos usando el programa Peak Scanner 1.0 (Applied Biosystems®).
La amplificación se realizó en dos etapas: 35 ciclos empleando la temperatura de hibridación (TA) específica de los cebadores SSRnu y ocho ciclos empleando la TA del cebador universal M13. Las reacciones de amplificación fueron realizadas en un termociclador con gradiente de temperatura (Biometra TProfessional Standard) a temperatura de hibridación fija de 54°C para los loc/ Ency-24 y Ency-33, de 56°C para los loc/ Ency-4 y Ency-17 y de 58°C para el locus Ency-8.
La diversidad genética se caracterizó mediante la estimación de los siguientes parámetros: Frecuencias alélicas por locus, Número promedio de alelos por locus (Na), Número efectivo de alelos por locus (Ne), Número promedio de alelos únicos (Nau), Heterocigosis observada (HO) y Heterocigosis esperada (He) calculada como:
He = í-'Zixf
donde x corresponde a las frecuencias alélicas (Nei, 1987). Estas estimaciones se realizaron empleando el programa GenAlEx versión 6.5 (Peakall & Smouse, 2012). Las estimaciones de riqueza alélica permiten independizar a las estimaciones de números de alelos del sesgo que puede introducir el tamaño de la muestra (Gillet et al., 2005). Por ello, se estimaron los siguientes parámetros: Riqueza alélica (R) y Riqueza de alelos únicos (Rau) (Gillet et al., 2005). Estas estimaciones se realizaron empleando el programa ADZE versión 1.0 (Szpiech et al., 2008).
La fuente de polen fue identificada considerando los alelos presentes en tres semillas de cada fruto y los genotipos multilocus de cada árbol madre. Este enfoque implica la inferencia de los alelos heredados por vía paterna directamente desde la detección de alelos por cada fruto y su comparación con los genotipos maternos (Godoy & Jordano, 2001). Debido a que el material empleado para la extracción de ADN genómico total fue tejido embrionario proveniente de tres semillas del mismo fruto podrían ser detectados más de dos alelos por locus. Así, se espera detectar por fruto, como mínimo, un alelo por locus (árboles madre homocigotas autofecundadas o con donantes de polen genotípicamente idénticos al árbol madre) y como máximo cinco alelos por locus (árbol madre heterocigota con donantes de polen genotípicamente diferentes al árbol madre).
El grado de estructura genética poblacional se analizó mediante el índice de fijación FST (Wright, 1965), el cual fue estimado a partir de un análisis de varianza molecular (AMOVA) testando la hipótesis nula de ausencia de estructura genética (Stewart & Excoffier, 1996). Este análisis fue realizado de manera global empleando dos modelos definidos atendiendo diferentes niveles de subdivisión jerárquica para analizar la distribución de la variabilidad genética: Modelo 1: Entre poblaciones y entre individuos dentro de poblaciones. Modelo 2: Entre poblaciones, entre individuos dentro de poblaciones y dentro de individuos. A partir de este último modelo se estimó el índice para inferir los efectos de la endogamia, es decir del cruzamiento entre individuos más emparentados que la media de la población (Hartl & Clark, 2007). La significancia estadística del índice de fijación fue testada con un valor de p obtenido mediante 1023 permutaciones de los genotipos dentro y entre poblaciones para el 95% de confianza (p<0,05). Este análisis fue desarrollado utilizando el programa Arlequín 3.5 (Excoffier & Lischer, 2010).
La estructura genética poblacional fue evaluada mediante algoritmos de agrupamiento Bayesiano para determinar el número más probable de clusters (K) a los cuales los individuos son asignados empleando únicamente información genética (genotipos multilocus). Para este análisis se empleó un modelo de tipo admixture (admixture ancestry model) implementado en el programa STRUCTURE 2.3.4 (Pritchard et al., 2000). Además, se empleó un modelo de agrupamiento espacial de individuos incorporando al agrupamiento no espacial el término correspondiente a la probabilidad a priori de la posición geográfica de cada individuo. Para determinar el número de clusters que presenta la mayor probabilidad a posteriori se testaron valores de K clusters comprendidos en un rango de uno a seis (K = [1-6]) realizando 10 repeticiones para cada valor de K. Este análisis se realizó empleando el programa TESS (Chen et al., 2007; Durand et al., 2009).
La representatividad de la diversidad genética nuclear fue estimada a partir de la diferencia entre la composición genética de cada población y de las semillas en relación a la composición genética de su complemento mediante la estimación del índice de diferenciación genética de Gregorius (Gregorius, 1984). Además, se estimó el índice de diferenciación subpoblacional (5) para representar el nivel medio de diferenciación entre las poblaciones y las semillas (Gregorius & Roberds, 1986). De esta manera, se caracterizó cada sitio de muestreo y las semillas mediante la diferencia entre su composición genética y la de su complemento, considerando como nivel de análisis al acervo génico en el cual los elementos genéticos corresponden a todos los alelos y a nivel de locus considerando los loci SSRnu estudiados en este trabajo. Para estos análisis se utilizó el programa GSED versión 3.0 (Gillet, 2010). Finalmente, el flujo génico (Nm) fue estimado empleando el método de alelos raros, incluyendo una corrección para el tamaño de la muestra implementando el programa GenePop v 2.4 (Rousset, 2008).
Resultados
Variables germinativas
En semillas sin escarificación se evidenciaron menores porcentajes de germinación (G entre 0 y 58%) en relación a los porcentajes registrados en semillas con escarificación (G entre 67 y 100%) (F = 864,39; p < 0,0001) (Tabla 1). El primer evento de germinación fue detectado a los cinco días y la última detección fue a los 11 días de iniciado el ensayo, que se dio por finalizado luego de transcurridos cinco días sin detección de eventos de germinación. En ese intervalo, las semillas sin tratamiento presentaron mayor frecuencia de germinación en t= 7-9 días, mientras que en las semillas con tratamiento pre-germinativo T = 5,386,75 días (Fig. 3). El coeficiente de variación del tiempo (CV) fue bajo tanto para las semillas sin tratamiento pre-germinativo (CVt = 20,48) como para las semillas con tratamiento pre-germinativo (CVt= 9,16-17,40), dada la uniformidad en el tiempo de germinación (Tabla 1). La tasa media de velocidad de germinación (v) fue mayor para semillas con tratamiento pre-germinativo (Fig. 3). Se detectó una moderada a baja sincronización (Z) y valores bajos de incertidumbre (U) (Tabla 1).
Tabla 1. Valores medios y desviaciones estándar de los parámetros de germinación de semillas de Enterolobium contortisiliquum considerando dos tratamientos pre-germinativos: sin escarificación (0); con escarificación (1). G: Porcentaje de semillas que completan el proceso de germinación; CV'. Coeficiente de variación del tiempo; Z: Índice de sincronía; U. Índice de incertidumbre; a. ausencia de diferencias estadísticamente significativas entre medias de acuerdo a las pruebas de comparación múltiple de medias SNK.
| Tratamiento pre- | Árbol | N° de semillas | G | cvt | Z | U |
| germinativo | germinadas | |||||
| 0 | T21 | 1 | 4,17 | 0 | 0 | 0 |
| 0 | T22 | 0 | 0 | 0 | 0 | 0 |
| 0 | T23 | 14 | 58,33 | 20,48 ± 7,34 a | 0,22 ± 0,11 a | 1,78 ± 0,58 a |
| 0 | T24 | 0 | 0 | 0 | 0 | 0 |
| 0 | T25 | 1 | 4,17 | 0 | 0 | 0 |
| 0 | T28 | 0 | 0 | 0 | 0 | |
| 0 | T29 | 0 | 0 | 0 | 0 | 0 |
| 1 | T21 | 23 | 95,83 | 11,48 ± 3,45 a | 0,35 ± 0,09 a | 1,52 ± 0,30 a |
| 1 | T22 | 16 | 66,67 | 9,16 ± 2,48 a | 0,48 ± 0,13 a | 1,06 ± 0,34 a |
| 1 | T23 | 24 | 100 | 10,86 ± 4,24 a | 0,44 ± 0,21 a | 1,27 ± 0,65 a |
| 1 | T24 | 24 | 100 | 15,98 ± 6,34 a | 0,35 ± 0,04 a | 1,48 ± 0,24 a |
| 1 | T25 | 24 | 100 | 17,4 ± 2,45 a | 0,39 ± 0,03 a | 1,42 ± 0,19 a |
| 1 | T28 | 24 | 100 | 15,13 ± 5,85 a | 0,30 ± 0,11 a | 1,62 ± 0,33 a |
| 1 | T29 | 22 | 91,67 | 12,35 ± 0,72 a | 0,54 ± 0,20 a | 1,06 ± 0,34 a |
Variables genéticas
Los cinco loci SSRs analizados resultaron polimórficos. Se detectó un total de 27 alelos considerando las dos poblaciones estudiadas. El número de alelos estuvo comprendido entre 2 y 14 para los loci Ency-33 y Ency-4, respectivamente (Fig. 4). En cuanto a la diversidad genética, la población Eldorado presentó un mayor número promedio de alelos por locus y número efectivo de alelos en relación a Ituzaingó (Tabla 2). En ambas poblaciones NE fue inferior a NA indicando la presencia de alelos a baja frecuencia, en tanto que Nau presentó valores promedio de 2,20 y 0,80 en Eldorado e Ituzaingó, respectivamente (Tabla 2). En conjunto, considerando los cinco loci, la heterocigosis observada (HO) fue mayor a la heterocigosis esperada (HE) evidenciando un elevado número de individuos heterocigotas en ambas poblaciones (Tabla 2). La población Eldorado presentó mayores valores de diversidad genética (HE=0,51; ^=3,38 y RAU=1,77) que la población Ituzaingó (HE=0,48; R=3,00 y RAU=1,39) (Tabla 2).
Por su parte, el análisis genético a nivel de semillas fue realizado desde cinco árboles madre: T21, T22, T24, T25 y T29, ya que no fue posible obtener ADN de calidad del tejido embrionario de semillas de los árboles madre T23 y T28. Considerando los cuatro loci en conjunto, se reveló el aporte de, al menos, un donante de polen con alelos distintos al árbol madre por fruto (Tabla 3). A nivel de los frutos del árbol madre T22 se identificó el aporte de, al menos, dos árboles donantes de polen mediante el registro de hasta tres alelos no detectados en el árbol madre por locus (Tabla 3).

Fig. 3. Tiempo medio de germinación (t) y Tasa media de velocidad de germinación (v) de siete genotipos de Enterolobium contortisiliquum considerando semillas con y sin tratamiento pre-germinativo. Las letras diferentes indican diferencias estadísticamente significativas entre medias de acuerdo a las pruebas de comparación múltiple de medias SNK.
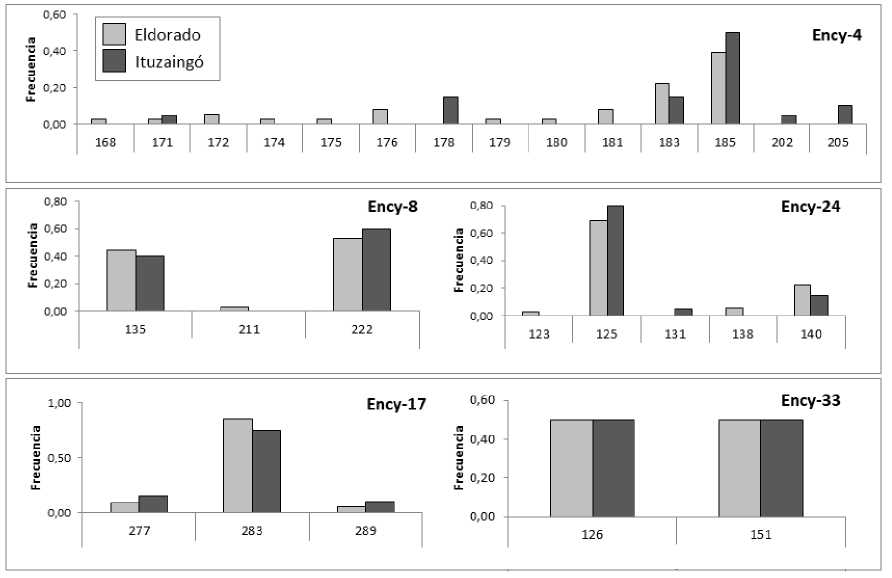
Fig. 4. Frecuencias alélicas por locus en las poblaciones de Enterolobium contortisiliquum estudiadas.
Mediante el análisis de varianza molecular (AMOVA) en ambos modelos se detectó que el mayor porcentaje de variación estuvo contenido en el nivel menos inclusivo: En el Modelo 1, dentro de poblaciones (100%) y en el Modelo 2, dentro de individuos (100%). A partir del Modelo 2 se estimó el índice FIS el cual presentó un valor de 0 al igual que el índice FST, indicando ausencia de endogamia y de estructura genética poblacional, respectivamente. Este último resultado es también respaldado por el modelo no espacial del análisis Bayesiano, el cual no evidenció estructura genética poblacional (Fig. 5A). Mientras tanto, a partir del análisis Bayesiano espacial, se identificó K
Tabla 2. Caracterización de la diversidad genética en las poblaciones de Enterolobium contortisiliquum analizadas para los cinco loci SSRs.
| Locus | Eldorado | Ituzaingó | ||||||||||||||
| N | Na | Ne | Nau | HO | R | Rau | N | Na | Ne | Nau | HO | R | Rau | |||
| Ency-4 | 18 | 11,00 | o LO | 0,88 | 0,77 | 10 | 6,00 | 3,22 | 0,90 | 0,69 | ||||||
| Ency-8 | 17 | 3,00 | 2,10 | 0,94 | 0,52 | 10 | 2,00 | 1,92 | 0,80 | 0,48 | ||||||
| Ency-17 | 17 | 3,00 | 1,35 | 0,29 | 0,26 | 10 | 3,00 | 1,68 | 0,30 | 0,40 | ||||||
| Ency-24 | 18 | 4,00 | 1,86 | 0,61 | 0,46 | 10 | 3,00 | 1,50 | 0,40 | 0,33 | ||||||
| Ency-33 | 18 | 2,00 | 2,00 | 1,00 | 0,50 | 10 | 2,00 | 2,00 | 0,80 | 0,50 | ||||||
| Media | "vi O) o | 4,60 | 2,36 | 2,20 | 0,74 | 0,51 | 3,38 | 1,77 | O O o | 3,20 | 2,06 | 0,80 | 0,64 | 0,48 | 3,00 | 1,39 |
| SE | 0,24 | 1,63 | 0,54 | 1,49 | 0,13 | 0,08 | 0,00 | 0,73 | 0,30 | 0,58 | 0,12 | 0,06 | ||||
Se indica N: tamaño de la población, NA. número promedio de alelos por locus, NE. número efectivo de alelos por locus, Nau: número promedio de alelos únicos por locus, HO. heterocigosis observada, HE: heterocigosis esperada, R . riqueza alélica, Rau: riqueza de alelos únicos, SE. Error estándar.
Tabla 3. Genotipos multilocus de los árboles madre y perfiles alélicos detectados a partir de tejido embrionario de Enterolobium contortisiliquum por fruto. Se indica: . Alelos detectados en el árbol madre; o Alelos no detectados en el árbol madre.
| Árbol T21 | Árbol T22 | Árbol T24 | Árbol T25 | Árbol T29 | ||||||||
| Locus | Alelos | Hojas | Frutos | Hojas | Frutos | Hojas | Frutos | Hojas | Frutos | Hojas | Frutos | |
| T21 | 21.1 21.2 | T22 | 22.1 22.2 | T24 | 24.1 24.2 | T25 | 25.1 25.2 | T29 | 29.1 | 29.2 | ||
| 183 | . | |||||||||||
| 185 | . | .. | . | .. | . | .. | . | .. | . | . | . | |
| 191 | o | |||||||||||
| 198 | o | |||||||||||
| 199 | o | |||||||||||
| 201 | o | |||||||||||
| Ency-4 | 202 | . | . | |||||||||
| 203 | o | oo | ||||||||||
| 205 | . | . | . | |||||||||
| 207 | o | |||||||||||
| 220 | o | |||||||||||
| 224 | o | |||||||||||
| 225 | o | |||||||||||
| 132 | o | |||||||||||
| 135 | . | oo | . | .. | . | . | . | . | . | |||
| Ency-8 | 149 | o | ||||||||||
| 151 | o | |||||||||||
| 170 | oo | o | ||||||||||
| 222 | . | .. | . | .. | . | . | . | .. | . | . | . | |
| 108 | o | o | ||||||||||
| 125 | . | .. | . | . | . | .. | . | .. | . | . | . | |
| 128 | o | |||||||||||
| Ency-24 | 131 | . | . | |||||||||
| 140 | . | .. | o | o | o | o | ||||||
| 142 | o | |||||||||||
| 148 | o | |||||||||||
| 112 | oo | |||||||||||
| 116 | o | |||||||||||
| Ency-33 | 120 | o | ||||||||||
| 126 | . | . | . | .. | . | .. | . | . | . | |||
| 151 | . | .. | . | .. | o | . | . | |||||
| 159 | o | oo | o | |||||||||
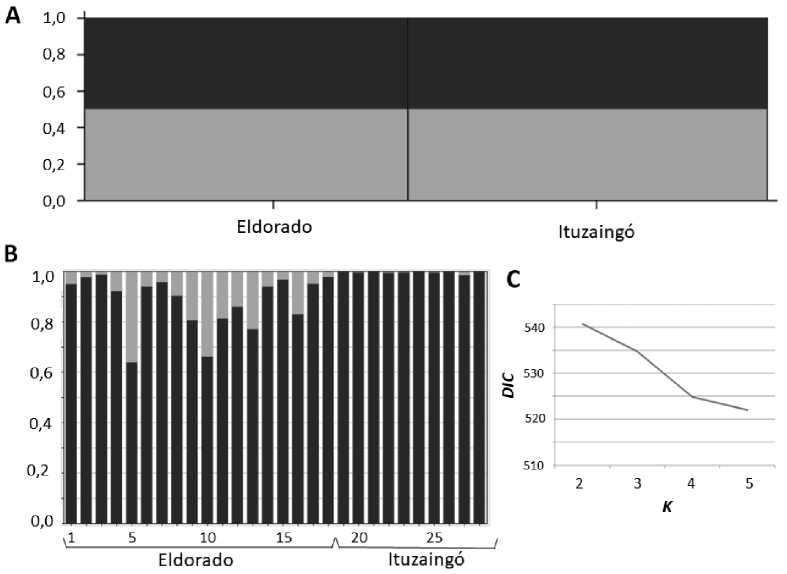
Fig. 5. Determinación de la estructura genética poblacional mediante inferencia Bayesiana. A: Inferencia Bayesiana no espacial; B: Inferencia Bayesiana espacial. Se indica población: Individuos del 1 al 18: Eldorado; Individuos del 19 al 28: Ituzaingó; C: Determinación de Kmáx en base a DIC vs. K.
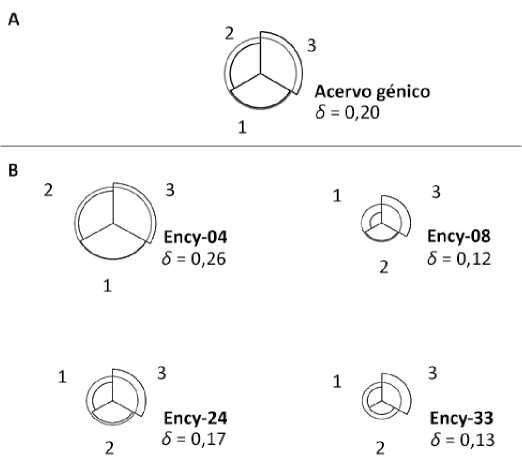
Fig. 6. Diferenciación genética a nivel de A: acervo génico y B: por locus mediante los marcadores Ency-4, Ency-8, Ency-24 y Ency-33. El radio correspondiente a lay-ésima subpoblación es proporcional a D, y el ángulo, proporcional al tamaño relativo c se ha igualado para todas las subpoblaciones. El círculo posee un radio proporcional a la diferenciación subpoblacional ó. Se indican Grupos: 1: Eldorado, 2: Ituzaingó y 3: Semillas.
máx
= 2 como número real de clusters genéticos a nivel global (Fig. 5C), detectándose una baja proporción de admixture o contribución de diferentes acervos génicos en individuos dentro de la población Eldorado y baja o nula estructura genética poblacional (Fig. 5B).
A nivel de locus, considerando los loci Ency-4, Ency-8, Ency-24 y Ency-33 de forma independiente y a nivel de acervo génico, las semillas presentaron mayor diferenciación genética respecto a su complemento. Las poblaciones Eldorado e Ituzaingó presentaron valores de D. menores al índice de diferenciación subpoblacional 5 (Fig. 6A). Eldorado resultó el sitio con mayor representatividad de la diversidad genética a nivel de los loci Ency-8 y Ency-24, mientras que a nivel del locus Ency-4 Ituzaingó presentó mayor representatividad. Ambos sitios de muestreo presentaron niveles equivalentes de diferenciación genética respecto al complemento a nivel del locus Ency-33 (D. = 0,098) evidenciando la presencia de alelos compartidos para ese locus (Fig. 6B).
El número efectivo de migrantes estimado a partir de la frecuencia de alelos raros en las poblaciones luego de la estandarización del tamaño de la muestra presentó un valor a nivel global de Nm = 2,70, siendo la frecuencia de alelos raros a nivel global p(1) = 0,05.
Discusión
El acervo génico de las poblaciones de E. contortisiliquum analizadas en el presente trabajo se caracteriza por una elevada diversidad genética, ausencia de estructura genética poblacional y ausencia de endogamia, atributos genético-poblacionales que en conjunto pueden ser explicados por los elevados niveles de flujo génico, el aporte de múltiples donantes de polen y las elevadas tasas de germinación de semillas.
A partir de las variables de germinación evaluadas, los valores máximos de porcentaje de germinación fueron obtenidos en semillas escarificadas mecánicamente. El porcentaje global de germinación en semillas sin escarificar fue de 10%, en tanto que el de las semillas escarificadas fue de 93%. Estos resultados son similares a los informados por Salazar et al. (2000), quienes indican que los porcentajes de germinación en semillas de E. contortisiliquum sin tratamiento pregerminativo pueden alcanzar hasta 22%, mientras que en semillas con tratamiento pre-germinativo los porcentajes de germinación varían entre 95 y 100%. En el presente trabajo se evidenció que el tratamiento pre-germinativo aplicado no sólo es necesario para romper la dormición de las semillas de E. contortisiliquum, sino que también acelera el proceso de germinación.
El estudio de la biología reproductiva de E. contortisiliquum ha sido abordado en un escaso número de trabajos (Hoc et al., 2015). Moreira et al. (2015) postulan que esta especie podría presentar fecundación cruzada debido, en parte, a estimas previas de heterocigosis que resultaron en HO > HE y HO=0,66 (Moreira et al., 2012). Estimas similares fueron obtenidas en el presente trabajo (HO = 0,69; HE =0,49), evidenciando elevada heterocigosis y diversidad genética, así como la presencia de alelos a baja frecuencia, dado el menor número efectivo de alelos en relación al número de alelos en ambas poblaciones analizadas. De esta manera, la diversidad genética detectada podría estar relacionada con elevadas tasas de fecundación cruzada en esta especie, las cuales han sido evidenciadas mediante la detección de más de un donante de polen por fruto. Al comparar el perfil alélico de las semillas por fruto con los genotipos multilocus de los árboles madre se identificaron alelos no detectados en cada árbol madre, lo cual reveló el aporte, como mínimo, de uno o dos árboles donantes de polen por fruto. La presencia de estos alelos identificados en las semillas podría ser consecuencia de un aumento de alelos nuevos originados por mutación, o bien incorporación de alelos a la población por flujo génico reciente mediado por polen a partir de individuos adultos no incluidos en el presente estudio que han aportado alelos al acervo génico, con lo cual el tamaño de las poblaciones de E. contortisiliquum podría ser mayor al considerado, destacando el rol de los polinizadores tales como las polillas que presentan largas distancias de vuelo.
La distribución de la variabilidad genética entre los diferentes niveles jerárquicos se caracterizó por un mayor porcentaje de variación contenido en el nivel jerárquico menos inclusivo en los dos modelos empleados, es decir, dentro de poblaciones y dentro de individuos. Además, se detectó ausencia de estructura genética tanto mediante el índice de fijación (Fst = 0) como mediante la implementación de algoritmos de inferencia Bayesiana, de manera que no se presentan diferencias en las frecuencias alélicas entre Eldorado e Ituzaingó. El flujo génico ininterrumpido entre los sitios de muestreo (Nm = 2,70) indica que en las poblaciones estudiadas los efectos del flujo génico son suficientes para contrarrestar los efectos de la deriva genética.
Si bien en el Noreste argentino los ambientes donde habita la especie en estudio se ven afectados por el avance de la frontera agrícola y la deforestación, aún se conservan grandes fragmentos de bosques nativos remanentes, de modo que las poblaciones estudiadas podrían estar interconectadas por flujo génico entre poblaciones intermedias. El modelo de flujo génico paso a paso (stepping-stone) entre poblaciones (Slatkin, 1993) explica los patrones de ausencia de estructura genética en casos en los cuales las poblaciones se presentan en múltiples parches de hábitat con diversos grados de conectividad, determinados en gran medida por el tamaño de los parches y la distancia geográfica que los separa (Le Corre & Kremer, 1998; Balkenhol et al., 2016). Ha sido descripta una elevada conectividad entre poblaciones de E. contortisiliquum como consecuencia de la influencia pasada de la dispersión de frutos mediada por la megafauna extinta, la dispersión de semillas por roedores y la dispersión de polen mediada por polinizadores con largas distancias de vuelo (Moreira et al, 2015). Por su parte, Hunter (1989) propone que semillas de E. cyclocarpum podrían ser dispersadas por hidrocoria, debido a que a diferencia de otras especies de la tribu Mimosoideae las del género Enterolobium incluida E. contortisiliquum, presentan vainas enroscadas sobre sí mismas formando espirales que se transportan mejor por el agua que las vainas rectas y alargadas. Además, estos frutos no se hunden en el agua permaneciendo a flote durante varios días y se humedecen rápidamente, proporcionando un ambiente que puede promover la germinación de las semillas (Hunter, 1989). En las especies ribereñas con dispersión por hidrocoria las semillas pueden recorrer varios kilómetros de distancia a través del agua propiciando flujo génico continuo y elevado tamaño efectivo poblacional (Imbert et al., 2003). De esta manera, la dispersión de frutos (y sus semillas) mediada por agua en la época de lluvias y/o por las corrientes de los ríos a lo largo de distancias considerables, puede explicar en parte, la elevada conectividad de las poblaciones (Gentry, 1993; Hunter, 1989). Estos factores influyen de manera significativa sobre la estructura genética espacial y sobre las tasas de fecundación cruzada de especies forestales de los BSEN (Goncalves et al. 2022), de manera que también influirían sobre la estructura genética y el sistema de fecundación de poblaciones de E. contortisiliquum distribuidas en estos bosques. Así, el tipo de dispersión de propágulos de E. contortisiliquum podría contribuir en la preservación de una elevada conectividad entre sus poblaciones. Considerando los elevados niveles de diversidad genética, así como también los valores de los índices de endogamia e índices de diferenciación genética detectados en el presente estudio se podría afirmar, en concordancia con Moreira et al. (2015), que el sistema de fecundación de E. contortisiliquum se caracteriza por una predominancia de fecundación cruzada de forma similar a lo descripto para E. cyclocarpum (Rocha & Aguilar, 2001b). De esta manera, el flujo génico juega un rol fundamental interconectando árboles espacialmente distantes o pequeños parches que actúan como puentes, contribuyendo a la viabilidad del conjunto de poblaciones, lo cual, sumado a la longevidad de los árboles de esta especie, podría atenuar las consecuencias genéticas de la creciente fragmentación del paisaje.
El conocimiento de la representatividad de la variabilidad genética facilita la identificación de unidades de conservación de la diversidad genética de especies forestales. En cada una de las unidades de conservación estará representada una fracción de la variabilidad genética de la especie y cada una de estas unidades representa un centro de diversidad genética (Manel et al., 2003). En el análisis de representatividad desarrollado en el presente estudio, tanto a nivel de locus como a nivel de acervo génico, las semillas presentaron la mayor diferenciación respecto a su complemento y, sumado a que ambas poblaciones presentaron elevados niveles de diversidad genética y baja diferenciación genética para los loci analizados, puede postularse que Eldorado e Ituzaingó podrían ser consideradas como posibles unidades de conservación de E. contortisiliquum en la región.
Conclusiones
Las poblaciones de E. contortisiliquum estudiadas presentan niveles elevados de diversidad genética nuclear siendo Eldorado la población que presenta la mayor diversidad. La población Ituzaingó se caracteriza por la contribución, como mínimo, de uno o dos donantes de polen por fruto. Las poblaciones no presentan estructura genética, como consecuencia de elevados niveles de flujo génico mediado por polen y por semillas. Los elevados niveles de diversidad genética identificados, los valores nulos del coeficiente de endogamia, la identificación de alelos heredados vía polen y la ausencia de estructura genética sustentan la alogamia como el sistema de fecundación dominante en las poblaciones estudiadas. En cuanto a la variabilidad genética en las semillas, un aumento de alelos nuevos originados por flujo génico a larga distancia mediado por polen explicarían la mayor diferenciación respecto a su complemento a nivel de locus y a nivel de acervo génico. De esta manera, la ausencia de estructura genética detectada queda explicada por los elevados niveles de flujo génico mediado por polen y por semillas, constituyendo un factor clave para contrarrestar las consecuencias genéticas de la creciente fragmentación del paisaje en el Noreste argentino.
Contribución de los Autores
CGM: Colecta del material, Metodología, Análisis de los datos, Redacción del manuscrito original, Redacción, revisión y edición del manuscrito final; MEB: Conceptualización, Colecta del material, Metodología, Análisis de los datos, Redacción, revisión y edición del manuscrito final; ALG: Colecta del material, Análisis de los datos, Redacción, revisión y edición del manuscrito final; MVG: Conceptualización, Colecta del material, Metodología, Adquisición de fondos, Administración del proyecto, Redacción, revisión y edición del manuscrito final.
Agradecimientos
El presente trabajo constituye parte de la tesis de grado de CGM para optar al título de Licenciada en Genética en la Facultad de Ciencias Exactas, Químicas y Naturales de la Universidad Nacional de Misiones (FCEQyN - UNaM). El trabajo fue parcialmente financiado con fondos correspondientes al Proyecto Especial FCEQyN - UNaM A-07: "Caracterización de recursos fitogenéticos de los campos del sur de Misiones" (Res. CS 1588/15) otorgado a MVG por el Centro de Investigación y Desarrollo Tecnológico de la FCEQyN - UNaM. Las autoras agradecen los aportes realizados en el proceso de revisión.
Bibliografía
ABRAHAM DE NOIR, F., S. BRAVO & R. ABDALA. 2002. Mecanismos de dispersión de algunas especies de leñosas nativas del chaco occidental y serrano. Quebracho 9: 140-150.
ALEXANDRE, R. S., F. G. GONQALVES, A. P. ROCHA, M. P. DE ARRUDA, & E. DE QUEIROZ LEMES. 2009. Tratamentos físicos e químicos na superajao de dormencia em sementes de Enterolobium contortisiliquum (Vell.) Morong. Rev. Bras. Cienc. Agrar. 4: 156-159. https://doi.org/10.5039/agraria.v4i2a6
ANDRIANOELINA, O., B. FAVREAU, L. RAMAMONJISOA & J. M. BOUVET. 2009. Small effect of fragmentation on the genetic diversity of Dalbergia monticola, an endangered tree species of the eastern forest of Madagascar, detected by chloroplast and nuclear microsatellites. Ann. Bot. 104: 1231-1242.
https://doi.org/10.1093/aob/mcp231 BALKENHOL, N., S. A. CUSHMAN, A. T. STORFER, L. P. WAITS. 2016. Landscape Genetics: Concepts, Methods, Applications. 1st ed. John Wiley and Sons Ltd, Chichester.
https://doi.org/10.1002/9781118525258.ch01 BARRETTO, S. S. B. & R. A. FERREIRA. 2011. Morphological aspects of fruits, seeds and seedlings of the Leguminosae Mimosoideae species: Anadenanthera colubrina (Vellozo) Brenan and Enterolobium contortisiliquum (Vellozo) Morong. Rev. Bras. Sementes 33: 223-232. https://doi.org/10.1590/S0101-31222011000200004
BERJAK, P, & N. W. PAMMENTER. 2002. Orthodox and recalcitrant seeds. En VOZZO J. A. (ed.) Tropical tree seed manual, pp 137-147. DCUSDA Forest Service, Washington.
BREED, M. F., OTTEWELL, K. M., GARDNER, M. G., MARKLUND, M. H., DORMONTT, E. E., & A. J. LOWE. 2015. Mating patterns and pollinator mobility are critical traits in forest fragmentation genetics. Heredity 115(2): 108-114. https://doi.org/10.1038/hdy.2013.48
CABRERA, A. L. 1971. Fitogeografía de la república Argentina. Bol. Soc. Argent. Bot. 14: 1-42.
CABRERA, A. L. 1976. Regiones fitogeográficas argentinas. Enciclopedia argentina de agricultura y jardinería. 2da ed. ACME S.A.C.I., Buenos Aires.
CHEN, C., E. DURAND, F. FORBES & O. FRANQOIS. 2007. Bayesian clustering algorithms ascertaining spatial population structure: a new computer program and a comparison study. Mol. Ecol. Notes 7: 747-756. https://doi.org/10.1111/j.1471-8286.2007.01769.x
CRUZ-SILVA, C. T. A., & A. P. M. ROSA. 2011. Tratamentos para superajao da dormencia em sementes de orelha-de-negro (Enterolobium contortisiliquum (Vell.) Morong). Varia Sci. Agrár. 2: 79-90.
DURAND, E., C. CHEN & O. FRANQOIS. 2009. TESS Version 2.3 Reference Manual. Disponible en: http:// membres-timc.imag.fr/Olivier.Francois/tess.html [Acceso: 11 November 2022].
EXCOFFIER, L. & H. E. L. LISCHER. 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour. 10: 564-567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
GENTRY, A. H. 1983. Dispersal ecology and diversity in neotropical forest communities. En K. KUBITZKI (ed.). Dispersal and Distribution: An International Symposium, pp. 303-314. Verlag Paul Parey, Berlin.
GILLET, E. 2010. GSED Genetic Structures from Electrophoresis Version 3.0 Data User's Manual. Abt. Forstgenetik und Forstpflanzenzüchtung, Universitat Gottingen, Gottingen.
GILLET, E., D. GOMORY & L. PAULE. 2005. Measuring genetic variation within and among populations at marker loci. En GEBUREK, T. & J. TUROK (eds.) Conservation and Management of forest genetic resource in Europe, pp 237-270. Arbora Publishers, Zvolen.
GODOY, J. A. & P. JORDANO. 2001. Seed dispersal by animals: exact identification of source trees with endocarp DNA microsatellites. Mol. Ecol. 10: 22752283. https://doi.org/10.1046/j.0962-1083.2001.01342.x
GONCALVES, A. L., M. E. BARRANDEGUY & M. V. GARCÍA. 2013. Método económico de genotipificación por fluorescencia de SSRs en Anadenanthera colubrina var. cebil. Actas del XLII Congreso Argentino de Genética. BAG, J. basic appl. genet. 24: 107.
GONCALVES, A. L., M. V. GARCÍA, M. E. BARRANDEGUY, S. C. GONZÁLEZ-MARTÍNEZ & M. HEUERTZ. 2022. Spatial genetic structure and mating system in forest tree populations from seasonally dry tropical forests: a review. Tree Genet. Genomes 18: 1-16. https://doi.org/10.1007/s11295-022-01550-1
GREGORIUS, H. R. & J. H. ROBERDS. 1986. Measurement of genetical differentiation among subpopulations. Theor. Appl. Genet. 71: 826-834. https://doi.org/10.1007/BF00276425 GREGORIUS, H. R. 1984. Measurement of genetic differentiation in plant populations. En GREGORIUS, H.R (ed.) Population Genetics in Forestry, pp. 276-285. Springer-Verlag, Berlín. https://doi.org/10.1007/978-3-642-48125-3_19 HARTL, D. L. & A. G. CLARK. 2007. Principles of population genetics. 4th ed. Sinauer Associates, Sunderland.
HATTEMER, H. H. 2005. On the appropriate size of forest genetic resources. En GEBUREK T, TUROK J (eds.) Conservation and Management of forest genetic resource in Europe, pp. 413-436. Arbora Publishers, Zvolen.
HOC, P. S. 2005. Inga MILL J. F. En: ANTON, A. M. & ZULOAGA F. O. (eds.) Flora Fanerogámica Argentina, fasc. 93, pp. 14-19. Córdoba: IMBIV (CONICET), Programa PROFLORA.
HOC, P. S., C. FLORES, E. LOZANO & A. ZAPATER. 2015. Preliminary studies on the reproductive biology of Enterolobium contortisiliquum. Res. Plant Biol. 5: 14-18.
HUNTER, R. J. 1989. Seed dispersal and germination of Enterolobium cyclocarpum (Jacq.) Griseb. (Leguminosae: Mimosoideae): Are megafauna necessary? J. Biogeogr. 16: 369-378. https://doi.org/10.2307/2845228 IMBERT, E. & F. LEFEVRE. 2003. Dispersal and gene flow of Populus nigra (Salicaceae) along a dynamic river system. J. Ecol. 91: 447-456. https://doi.org/10.1046/j.1365-2745.2003.00772.x
LE CORRE, V. & A. KREMER. 1998. Cumulative effects of founding events during colonisation on genetic diversity and differentiation in an island and stepping-stone model. J. Evol. Biol. 11: 495-512. https://doi.org/10.1046/j.1420-9101.1998.11040495.x
LOZANO, E. C., M. A. ZAPATER, C. MAMANI, C. B. FLORES, M. N. GIL & S. S. SÜHRING. 2016. Efecto de pretratamientos en semillas de Enterolobium contortisiliquum (Fabaceae) de la selva pedemontana argentina. Bol. Soc. Argent. Bot. 51: 79-87. https://doi.org/10.31055/1851.2372.v51.n1.14414
LOZANO-ISLA, F., O. E. BENITES-ALFARO & M. F. POMPELLI. 2019. GerminaR: An R package for germination analysis with the interactive web application "GerminaQuant for R". Ecol. Res. 34: 339-346. https://doi.org/10.1111/1440-1703.1275 MANEL, S., M. K. SCHWARTZ, G. LUIKART & P. TABERLET. 2003. Landscape genetics: combining landscape ecology and population genetics. Trends Ecol. Evol. 18: 189-197. https://doi.org/10.1016/S0169-5347(03)00008-9 MOREIRA, P. A., S. A. S. SOUZA, E. A. OLIVEIRA, N. H. ARAÚJO, G. W. FERNANDES & D. A. OLIVIERA. 2012. Charaterization of nine tranferred SSR markers in the tropical tree species Enterolobium contortisiliquum (Fabaceae). Genet. Mol. Res. 11: 2338-2342. https://doi.org/10.4238/2012.August.17.11 MOREIRA, P., M. M. BRANDAO, N. H. DE ARAUJO,
D. A. DE OLIVEIRA & G. W. FERNANDES. 2015. Genetic diversity and structure of the tree Enterolobium contortisiliquum (Fabaceae) associated with remnants of a seasonally dry tropical forest. Flora 210: 40-46. https://doi.org/10.1016/jflora.2014.10.005
MOREIRA, P.A, F. DE SIQUEIRA NEVES & J.A. LOBO. 2021. Consequences of tropical dry forest conversion on diaspore fate of Enterolobium contortisiliquum (Fabaceae). Plant Ecol. 222, 525535. https://doi.org/10.1007/s11258-021-01124-6 MORELLO, J. & J. ADÁMOLI. 1974. Las grandes unidades de vegetación y ambiente del Chaco argentino. Parte II. Provincia del Chaco. Serie Fitogeográfica del INTA, Buenos Aires.
NEI, M. 1987. Genet. Sel. Evol. Columbia University Press, New York.
PEAKALL, R. & P.E. SMOUSE 2012. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics 28: 2537-2539. https://doi.org/10.1093/bioinformatics/bts460
PETERS, M.B., C. HAGEN, D. W. TRAPNELL, J. L.HAMRICK, O. ROCHA, P. E. SMOUSE & T. C. GLENN. 2008. Isolation and characterization of microsatellite loci in the Guanacaste tree, Enterolobium cyclocarpum. Mol. Ecol. Resour. 8: 129-131. https://doi.org/10.1111/j.1471-8286.2007.01896.x PRADO, D. E. 1993. What is the Gran Chaco vegetation in South America? II. A redefinition. Contribution to the study of flora and vegetation of the Chaco. VII. Candollea 48: 615-629. https://doi.org/10.1017/S096042860000041X PRADO, D. E. 2000. Seasonally Dry Tropical Forest of tropical South America: From forgotten ecosystems to a new phytogeographic unit. Edinburgh J. Bot. 57(03): 437-461.
PRITCHARD, J. K., M. STEPHENS & P. DONNELLY. 2000. Inference of population structure using multilocus genotype data. Genetics 155: 945-959. https://doi.org/10.1093/genetics/155.2.945 ROCHA, O. J. & G. AGUILAR (2001a). Reproductive biology of the dry forest tree Enterolobium cyclocarpum (Guanacaste) in Costa Rica: a comparison between trees left in pastures and trees in continuous forest. Am. J. Bot. 88: 1607-1614. https:// doi.org/10.2307/3558405
ROCHA, O. J. & G. AGUILAR. 2001b. Variation in the breeding behavior of the dry forest tree Enterolobium cyclocarpum (Guanacaste) in Costa Rica. Am. J. Bot. 88: 1600-1606. https://doi.org/10.2307/3558404 ROUSSET, F. 2008. Genepop'007: a complete re-implementation of the genepop software for Windows and Linux. Mol. Ecol. Resour. 8: 103-106. https://doi. org/10.1111/j.1471-8286.2007.01931.x SALAZAR, R. 2000. Manejo de semillas de 100 especies forestales de América Latina. Proyecto de Semillas Forestales/Danida Forest Seed Centre. CATIE, Turrialba, Costa Rica.
SCHUELKE, M. 2000. An economic method for the fluorescent labeling of PCR fragments. Nat. Biotechnol. 18: 233. https://doi.org/10.1038/72708
SLATKIN, M. 1993. Isolation by distance in equilibrium and nonequilibrium populations. Evolution 47: 264279. https://doi.org/10.1111/j.1558-5646.1993.tb01215.x SORK, V. L., & SMOUSE, P. E. 2006. Genetic analysis of landscape connectivity in tree populations. Landsc. Ecol. 21: 821-836. https://doi.org/10.1007/s10980-005-5415-9
STEWART, C. N. & L. EXCOFFIER. 1996. Assessing population genetic stracture and variability with RAPD data: Application to Vaccinium macrocarpa (American cranberry). J. Evol. Biol. 9: 153-171. https://doi.org/10.1046/j.1420-9101.1996.9020153.x SZPIECH, Z.A., M. JAKOBSSON & N.A. ROSENBERG. 2008. ADZE: A rarefaction approach for counting alleles private to combinations of populations. Bioinformatics 24: 2498-2504. https:// doi.org/10.1093/bioinformatics/btn478
TOMMASI, F., C. PACIOLLA & O. ARRIGONI. 1999. The ascorbate system in recalcitrant and orthodox seeds. Physiol. Plant. 105: 193-198. https://doi.org/10.1034/j.1399-3054.1999.105202.x TORTORELLI, L.A. 2009. Maderas y bosques argentinos.
Buenos Aires, Argentina. Orientación Gráfica Editora. WRIGHT, S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution 19: 395-420. https://doi.org/10.1111/j.1558-5646.1965.tb01731.x