Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Ciencia y Tecnología
versión On-line ISSN 1851-7587
Rev. cienc. tecnol. no.14 Posadas jul./dic. 2010
GENÉTICA-BIOLOGÍA
Protocolo de extracción de DNA por salting-out para pequeños volúmenes de sangre
A modified salting-out protocol for DNA extraction from small blood sample volumes
Mario Alejandro Riera1, María Eugenia Rojas1, Pedro Darío Zapata1
1. Laboratorio de Biotecnología Molecular. Módulo de Bioquímica y Farmacia. FCEQyN, UNaM. Av. Mariano Moreno 1375, 3300. Posadas, Misiones, Argentina.
• Mario Alejandro Riera1 Es alumno avanzado de la carrera de Bioquímica. Se desempeña como Auxiliar Alumno de la Cátedra de Biología Molecular y Genética desde el año 2006. Participa del proyecto incentivado 16Q361 (Financiado por la ANPCyT PICTO 37029), habiendo participado además del proyecto financiado por la ANPCyT (PICT 05-15058). Realiza tareas de extensión y servicio en el Laboratorio de Biotecnología Molecular. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• María Eugenia Rojas1 Es alumna avanzada de la carrera de Bioquímica. Se desempeña como Auxiliar Alumno de la Cátedra de Biología Molecular y Genética desde el año 2006. Participa del proyecto incentivado 16Q361 (Financiado por la ANPCyT PICTO 37029), habiendo participado además del proyecto financiado por la ANPCyT (PICT 05-15058). Realiza tareas de extensión y servicio en el Laboratorio de Biotecnología Molecular. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• Pedro Darío Zapata1 Es Doctor por la Universidad de Alcalá de Henares. Se desempeña actualmente como Profesor Regular Adjunto en las Cátedras de Biología Celular y Molecular (Bioquímica), Genética Molecular (Lic. en Genética), Biología Celular (Lic. en Genética) y Biotecnología Molecular (Ingeniería Química, Bioquímica y Farmacia). Posee actualmente la Categoría III en el Sistema Nacional de Incentivos a los Docentes-Investigadores. En el área de Biomedicina ha sido beneficiario de subsidios de la ANPCyT: PICT 05-15058 y PICTO 37029. Dirige auxiliares, becarios de grado y posgrado. Posee publicaciones en revistas indexadas nacionales e internacionales y presentaciones a congresos nacionales e internacionales.
Resumen
Se desarrolló un protocolo modificado de extracción de DNA por salting-out a partir de muestras de sangre con volúmenes decrecientes (300; 100; 50; 25; 10; 5; 2,5; 1 y 0,5 µL). Se llevó a cabo posteriormente una amplificación de un fragmento de 345 pb del gen de la protrombina y de un marcador STR (short tandem repeat) utilizado comúnmente para estudios de filiación, mediante la reacción en cadena de la polimerasa, visualizándose los amplicones en un gel de poliacrilamida al 6,4 %. En todos los casos, la reacción de amplificación fue exitosa, demostrando así que el protocolo de extracción desarrollado permite obtener DNA en condiciones de pureza y cantidad suficiente a partir de pequeños volúmenes de sangre. De este modo, puede disminuirse la cantidad de muestra necesaria para realizar un estudio molecular, lo cual es fundamental en áreas como la genética forense donde la cantidad de muestra disponible puede ser limitada.
Palabras clave: Muestras de sangre; Extracción DNA; salting-out; PCR.
Abstract
A modified salting-out procedure for DNA extraction from decremental blood sample volumes was developed (300; 100; 50; 25; 10; 5; 2,5; 1 and 0,5 µL). A 345 bp human prothrombin gene fragment and a STR marker commonly used for filiation analyses (LPL) were then amplified by Polymerase Chain Reaction and visualized in a 6.4 % polyacrylamide gel. In all cases the amplification reaction was successful, showing that the DNA extraction method proposed here yields DNA of reasonable purity and quantity from small blood sample volumes. Therefore, the quantity of sample needed to perform a molecular study can be reduced, which is fundamental in certain areas such as forensic genetics, where the quantity of samples available could be limited.
Key words: Blood sample; DNA extraction; salting-out; PCR.
Introducción
La extracción de DNA a partir de muestras de distinta naturaleza, constituye la etapa previa de todo análisis genético. Obtener DNA relativamente puro y amplificable resulta fundamental para los posteriores usos a los que será destinado, en el marco de investigaciones de tipo forense, poblacionales, predisposición a enfermedades y otros. Esta tarea demanda una "puesta a punto" de las técnicas de extracción utilizadas, con el objeto de determinar las condiciones ideales en las que se obtendrá un máximo rendimiento. En general; los métodos de extracción de DNA tienen una serie de pasos básicos, que se cumplen independientemente del origen de la muestra. Estos son, a saber: disrupción celular (ruptura de la bicapa lipídica de las membranas celulares por tratamiento con detergentes, agentes quelantes que secuestran cationes estabilizantes de la membrana, etc.), eliminación de las proteínas (que constituyen los principales contaminantes del extracto), concentración del DNA (por precipitación con alcoholes), lavado (para eliminar restos de reactivos y solventes que puedan inhibir la Taq polimerasa) y resuspensión. [1, 2]
Para muestras de sangre en particular, distintos protocolos han sido descriptos y publicados, pudiendo diferenciarse básicamente dos alternativas metodológicas, las cuales difieren en la etapa de la purificación de las proteínas del extracto [3, 4]. Así podemos encontrar métodos que aprovechan las propiedades desnaturalizantes que tienen ciertos solventes orgánicos como cloroformo, fenol, etc. [1, 3], y a aquellos fundamentados en el conocido cambio de solubilidad que experimentan las proteínas cuando son sometidas a modificaciones en la concentración salina del medio [1, 5]. Así, en presencia de bajas concentraciones de sales, se verifica un ligero aumento en la solubilidad de las proteínas (salting-in) por aumento de la fuerza iónica del medio respecto de soluciones acuosas. Por el otro lado, en presencia de altas concentraciones de sales, la solubilidad de las proteínas disminuye y terminan por precipitar (salting-out) [6]. Este es el fundamento fisicoquímico de la precipitación de proteínas por salado. [7].
Sin embargo, estos protocolos requieren siempre una previa estandarización a las condiciones y recursos del laboratorio donde se lleven a cabo, así como también es necesario introducir modificaciones pertinentes a las situaciones especiales que pudieran plantearse. Entre ellas, la disponibilidad de muestra constituye un problema que puede presentarse con cierta frecuencia en estudios forenses o similares. Por ello, es de importancia determinar las condiciones bajo las cuales es posible la obtención de DNA en condiciones de relativa pureza, para ser empleado en los estudios genéticos posteriores a los que será destinado.
Materiales y métodos
Extracción de DNA por salting-out
Se obtuvo una muestra de sangre con anticoagulante (EDTA) y se tomaron alícuotas de 300; 100; 50; 25; 10; 5; 2,5; 1 y 0,5 µL de la misma. Cada una de estas alícuotas fue tratada con una solución de lisis de glóbulos rojos conteniendo Tris-HCl 10 mM pH 8, Triton X-100 1 % y Sacarosa 11 %; luego de lo cual se efectuó una centrifugación a 12000 rpm durante 3 min, descartándose el sobrenadante. Este paso de eliminación de las células anucleadas del extracto, se realizó 1, 2 o 3 veces según la alícuota utilizada (ver Tabla 1). Posteriormente se procedió a lisar los leucocitos mediante tratamiento con una solución de lisis conteniendo buffer de glóbulos blancos (Tris-HCl 10 mM pH 8; NaCl 400 mM; EDTA 2 mM), solución de Proteinasa K (Proteinasa K 1 mg/ml; SDS 1 %; EDTA 2 mM) y SDS 20 % en distintas proporciones. Se incubó una hora a 65 °C, agitando el preparado con vórtex cada 10 min. Seguidamente las proteínas fueron eliminadas del extracto mediante precipitación salina (salting-out) con acetato de potasio 3 M y posterior centrifugación a 12000 rpm por 5 min, para luego recuperarse el sobrenadante. Para mejorar la pureza, este paso fue realizado 2 veces. Luego, el DNA fue precipitado por adición de isopropanol y centrifugación a 12000 rpm durante 5 min. El pellet obtenido fue lavado con una solución de etanol 70 % y centrifugado a 12000 rpm durante 5 min, y se procedió a extraer todo el alcohol remanente. Finalmente, el pellet fue resuspendido en agua estéril. Los volúmenes utilizados de cada una las soluciones y reactivos, fueron ajustados en forma proporcional al volumen de la alícuota utilizada en la extracción (ver Tabla 1).
Tabla 1: Protocolo de extracción de DNA.
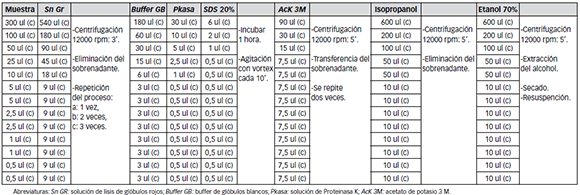
Abreviaturas
Sn GR: solución de lisis de glóbulos rojos; Buffer GB: buffer de glóbulos blancos; Pkasa: solución de Proteinasa K; AcK 3M: acetato de potasio 3 M.
Reacción en cadena de la polimerasa (POR)
La amplificación de un fragmento de 345 pb del gen de la protrombina (Sentido 5'-tctagaaacagttgcctggc-3' y antisentido 5'-atagcactgggagcattgaag c-3') se llevó a cabo en un volumen final de 25 µL por reacción con 4 µL de DNA y 20 pmoles de cada cebador (Invitrogen) en presencia de KCl; 1,5 mM MgCl2; 200 uM de cada dNTP (Promega) y 0,5 U de Taq polimerasa (Fermentas). El ciclado se realizó con una primera etapa de desnaturalización a 94 °C durante 3 min, seguido de 35 ciclos con segmentos de 20 seg a 94 °C, 50 °C y 72 °C respectivamente; para finalizar con una etapa de extensión a 72 °C durante 3 min.
La amplificación del marcador LPL se realizó utilizando el sistema GenePrint STR Systems (Silver Stain Detection) de Promega, en un volumen final de 12,5 µL por reacción con 2,5 µL de DNA y 1X de locus-specific Primer Pair; en presencia de 1X STR Buffer y 0,125 U de Taq polimerasa (Fermentas). El ciclado se realizó con una primera etapa de desnaturalización a 96 °C durante 2 min, seguido de 10 ciclos con segmentos de 1 min a 94 °C, 1 min a 60 °C y 1,5 min a 70 °C; y luego 20 ciclos con segmentos de 1 min a 90 °C, 1 min a 60 °C y 1,5 min a 70 °C para finalizar con una etapa de 30 min a 60 °C.
Gel de poliacrilamida
La visualización de los fragmentos amplificados por PCR se llevo a cabo mediante una corrida electroforética en un gel de poliacrilamida al 6,4 %, durante 90 min a 110 V, y luego fue teñido con nitrato de plata. [1].
Resultados
Todas las diluciones mostraron la presencia de una banda de amplificación correspondiente al fragmento del gen de laprotrombina, de 345 pb (ver Figura 1 y 2). Esto indica que las modificaciones introducidas al protocolo de extracción de DNA por salting-out, resultaron exitosas, logrando obtenerse DNA en condiciones de pureza adecuadas (libre de inhibidores) como para permitir la reacción de amplificación por PCR, utilizando a un fragmento del gen de la protrombina como DNA blanco o matriz.
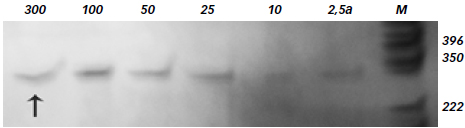
Figura 1. Amplificación del gen protrombina. Las alícuotas de 300, 100, 50, 25, 10 y 2,5 (a) µL fueron amplificadas con cebadores para un fragmento del gen de protrombina y separadas en un gel de poliacrilamida al 6,4 %. M: Marcador de peso molecular. La flecha señala la banda correspondiente al amplicón de 345 pb.
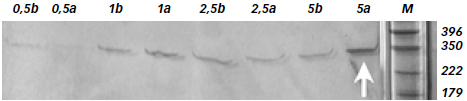
Figura 2. Amplificación del gen protrombina. Las alícuotas de 0,5 (a y b); 1 (a y b); 2,5 (a y b) y 5 (a y b) µL fueron amplificadas con cebadores para un fragmento del gen de protrombina y separadas en un gel de poliacrilamida al 6,4 %. M: Marcador de peso molecular. La flecha señala la banda correspondiente al amplicón de 345 pb.
Asimismo, para demostrar que las modificaciones introducidas al método de extracción por salting-out resultarían útiles en el campo de la genética forense, se realizó una PCR con un marcador STR usualmente utilizado para estudios de filiación: LPL. Los resultados de la reacción de amplificación se observan en la Figura 3, y evidencian la presencia de la banda correspondiente a uno de los alelos del marcador LPL.
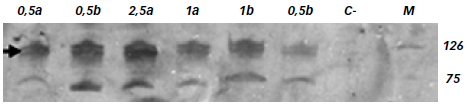
Figura 3. Amplificación del marcador microsatélite LPL. Las alícuotas de 0,5 (b); 1 (a y b); 2,5 (a), 5 (a y b) µL fueron amplificadas con cebadores para el marcador STR denominado LPL y separadas en un gel de poliacrilamida al 6,4 %. C-: control negativo. M: Marcador de peso molecular. La flecha señala la posición de los alelos correspondientes al marcador LPL (105-133 pb).
Discusión
Nuestros resultados ponen de manifiesto la gran sensibilidad que puede presentar la técnica de salting-out cuando es convenientemente adaptada al trabajo con pequeños volúmenes de sangre. De este modo, es posible reducir el volumen de muestra inicial utilizada en la extracción llegando hasta 0,5 µL de acuerdo a nuestros resultados.
Las técnicas de extracción de DNA disponibles en formato de kits comerciales no suelen contemplar la posibilidad de minimizar el uso de muestras cuando es necesario, situación que puede presentarse en casos forenses o similares. Además, no llegan al nivel de sensibilidad alcanzado con nuestro protocolo, considerando el volumen de muestra de sangre de partida, de acuerdo a lo especificado en los manuales correspondientes de los respectivos kits (QIAamp DNA Micro Kit [8], Genomic DNA Purification Kit [9], BLOODCLEAN DNA Purification kit [10], Invisorb Spin Blood Mini Kit [11]).
Asimismo, esto reviste también una importancia económica, teniendo en cuenta que las cantidades de los distintos reactivos y soluciones a utilizar por muestra, también resultan considerablemente menores a las ocupadas usualmente, como lo indica la Tabla 1; permitiendo que el costo del análisis por muestra sea inferior. Por otro lado, si bien existen kits especialmente diseñados para casos excepcionales de bajo volumen de muestra, debe contemplarse que los mismos son costosos, sobre todo en laboratorios donde el trabajo con pequeños volúmenes no es algo rutinario y adquirirlos demanda una amortización prolongada en el tiempo.
Esta técnica ha demostrado también ser capaz de adaptarse al campo de la genética forense y estudios de filiación, que necesitan de DNA de buena calidad obtenido a partir de pequeños volúmenes de muestra para amplificación de marcadores moleculares microsatélites, como los denominados short tandem repeat (STR).
Conclusiones
Las adaptaciones del método de extracción salting-out permitieron la obtención de DNA de calidad y pureza necesarias para la amplificación mediante PCR a partir de pequeños volúmenes de sangre, con un volumen mínimo de 0,5 µL de sangre de partida.
1. Cariaga Martinez A. E., Zapata P. D. El laboratorio de Biología Molecular. Una guía práctica. Colección: cuadernos de Cátedra. Editorial Universitaria de Misiones. Año 2005. [ Links ]
2. Parimoo S., P arimoo B. Sample Preparation Techniques in Analytical Chemistry. Volume 162. Cap. 6. Sample preparation in DNA analysis, p. 271-290. 2003. Disponible en http://www3.interscience.wiley.com/cgi-bin/bookhome/106561285/ [ Links ]
3. QIAGEN. Sample & Assay Technologies. Technical hints, applications, and protocols. Genomic DNA purification. P. 10-11 Disponible en http://www1.qiagen.com/literature/brochures/Gen_DNA_Pur/1019469_ BROS_DNYTi_INT_0502.pdf. [ Links ]
4. Austin M., Ordovas J., Eckfeldt J., Tracy R., Boerwinkle E., Lalouel J.M., Printz M. Guidelines of the National Heart, Lung, and Blood Institute Working Group on Blood Drawing, Processing, and Storage for Genetic Studies. American Journal of Epidemiology. Vol. 144. Number 5, p. 437-441. 1996. [ Links ]
5. Miller S.A., Dykes D.D. A simple salting-out procedure for extracting DNA from human nucleated cells. Nucleic Acid Research; 16(3): p. 1215; 1988. [ Links ]
6. Claros M., Avila C., Gallardo F., Canovas F. Bioquímica Aplicada. Diseño Experimental y Análisis de Datos; Septem Ediciones, 2001; Madrid España. p 36, 37. [ Links ]
7. Huan-xiang Zhou. Interactions of Macromolecules with Salt Ions: An Electrostatic Theory for the Hofmeister Effect. PROTEINS: Structure, Function, and Bioinformatics 61:69-78 (2005). [ Links ]
8. QIAGEN. Sample & Assay Technologies. 2003-2008. QIAamp DNA Micro Kit. For purification of genomic and mitochondrial DNA from small amounts of fresh or frozen blood, tissue, forensic samples, and dried blood spots. Disponible en: http://www1.qiagen.com/Products/GenomicDnaStabilizationPurification/QIAamp-System/QIAampDNAMicroKit.aspx?ShowInfo=1. [ Links ]
9. Fermentas. Life Sciences. Nucleic acid purification, kits: Genomic DNA Purification Kit. Disponible en http://www.fermentas.com/profiles/kits/pdf/coa_k0512.pdf. [ Links ]
10. BIOTOOL. BLOODCLEAN DNA Purification kit. Disponible en http://www.biotools.net/esp/productos/b5.htm. [ Links ]
11. Thistle Scientific. Invisorb Spin Blood Mini kit. Disponible en http://www.thistlescientific.co.uk/acatalog/Invisorb_Spin_Blood_Mini_kit.html. [ Links ]
Recibido: 05/10/08.
Aprobado: 11/05/10.