Introducción
El desierto de Atacama del norte de Chile, es uno de los desiertos más áridos del planeta, producto de la subsidencia del anticiclón subtropical, el cual es influenciado por la Corriente de Humboldt, la distancia del Atlántico, y el efecto del cordón de los Andes (Ritter et al, 2019). Recientemente, debido al cambio climático, las plantas provenientes de zonas desérticas cobran aún más relevancia, debido a que están adaptadas a entornos hostiles y con capacidad de tolerancia al estrés abiótico (Jiménez et al, 2017). En Chile, una de las pocas especies de árboles nativos adaptadas a las condiciones ecológicamente limitantes del desierto de Atacama es Geoffroea decorticans (Gillies ex Hook. & Arn.) Burkart (Fabaceae), comúnmente conocido como chañar (Contreras et al, 2018). En Argentina, el chañar está distribuido en diversas provincias, como Buenos Aires, Catamarca, Chaco, Córdoba, Corrientes, Entre Ríos, Formosa, Jujuy, La Rioja, Río Negro, La Pampa, Mendoza, San Juan, San Luis, Tucumán y Santa Fe, entre otras (Ireland & Pennignton, 1999).
El género Geoffroea Jacq. pertenece a la familia Fabaceae, y consta de cuatro especies aceptadas: Geoffroea horsfieldii (Lesch.) Oken, Geoffroea spinosa Jacq., Geoffroea violácea (Aubl.) Pers. y Geoffroea decorticans (Hook. & Arn.) Burkart y de ocho especies cuyo status taxonómico está aún sin resolver (Plant List, 2021). Por otro lado, se han observado diferencias intraespecíficas dentro de G. decorticans. Así, Lamarque et al. (2009), a través de un estudio del perfil electroforético de proteínas de semillas de G. decorticans, observaron grandes diferencias entre las variantes G. decorticans var. decorticans y var. subtropicalis, aclarando que estas variedades están menos relacionadas de lo que se creía, y sugirió la necesidad de estudiar más individuos en distintas regiones para confirmar su taxonomía.
De acuerdo a la Unión Internacional para la Conservación de la Naturaleza (UICN), a nivel mundial esta especie está clasificada como Preocupación Menor (Groom, 2012); mientras que en Chile la comisión de clasificación de especies del Ministerio de Medio Ambiente (17°proceso 2020-2021) la propuso como Vulnerable (MMA, 2021). Geoffroea decorticans es un recurso multipropósito utilizado para la alimentación, construcción de artefactos, muebles y viviendas (Giménez, 2004; Núñez et al., 2009; Castro, 2020; Ugalde et al., 2020). Los frutos de G. decorticans, pueden consumirse frescos y/o secos, o también ser utilizados para la elaboración de harina, bebidas alcohólicas y arrope, siendo este último producto un excelente jarabe para el tratamiento de la tos, además, la corteza, hojas y flores tienen propiedades medicinales como expectorantes (Costamagna et al, 2013; Echeverría et al., 2020). Por otro lado, las semillas poseen actividad anticoagulante e hipoglucemiante, presentando inhibidores de serina proteasa, los cuales son los más potentes aislados desde una fuente vegetal (Cotabarren et al., 2020). Además, las semillas de G. decorticans poseen propiedades nutritivas, constituyendo una buena fuente de nutrientes esenciales, como proteínas, carbohidratos y lípidos (Maestri et al., 2001;Cittadini et al., 2021). Actualmente en Chile, la valorización del arrope y harina de chañar como Producto Forestal No Maderero, se vincula directamente a las comunidades indígenas, siendo reconocido como un recurso patrimonial local (Várela et al., 2019).
Para conservar los recursos genéticos de esta especie, es necesario realizar más estudios de sus poblaciones, sin embargo, lamentablemente hoy existen pocos trabajos focalizados en determinar su diversidad y variabilidad genética. En este contexto, los microsatélites SSR (simple sequence repeat), son una buena alternativa como marcador codominante para realizar estudios de diversidad y estructura genética, debido a que exhiben una alta tasa de polimorfismo, son útiles para identificar hibridación, análisis de flujo génico y estudios de parentesco y mapeo genético (Vieira et al., 2016; Contreras et al., 2019, 2020a, 2020b). De acuerdo a Fox et al. (2019) en 2018 se publicaron aproximadamente 2.000 nuevos paneles de marcadores de microsatélites, sugiriendo que los microsatélites continúan siendo populares, y se vislumbra que seguirán utilizándose ampliamente en el futuro en estudios de conservación y ecología.
Los primeros microsatélites desarrollados en el género Geoffroea, fueron realizados en la especie G. spinosa, para caracterizar poblaciones de Perú, Argentina y Paraguay gracias a 10 loci SSR polimórficos (Naciri-Graven et al., 2005). Luego, usando 6 de estos microsatélites, un estudio profundizó la caracterización de la diversidad genética y diferenciación de poblaciones de G.
spinosa de la parte continental de Perú y las Islas Galápagos, revelando diferencias significativas entre las dos regiones geográficas (Caetano et al., 2012). Además, cinco de estos diez microsatélites polimórficos, descritos por Naciri-Graven et al. (2005), fueron transferibles a la especie G. decorticans, permitiendo realizar los primeros estudios genéticos de poblaciones de chañar establecidas en el Desierto de Atacama (Contreras et al, 2019). Sin embargo, creemos que es necesario desarrollar más marcadores SSR con el fin de dar más soporte a los análisis genéticos de poblaciones de G. decorticans. Para ello, se buscó identificar nuevos marcadores de ADN de tipo microsatélites mediante secuenciación NGS. El presente estudio tuvo como objetivo desarrollar marcadores SSR para G. decorticans a través de la plataforma Illumina, con el fin de realizar futuros estudios de diversidad, diferenciación genética y estructura genética de poblaciones de chañar.
Materiales y Métodos
Especie en estudio
Geoffroea decorticans, es un arbusto o árbol xerófilo, de entre 2 y 12 m de altura, de hojas pinnadas, con un fruto drupáceo y de flores amariposadas, que se reproduce mediante semillas y raíces gemíferas (Burkart, 1949; Echeverría et al, 2020), el cual se distribuye en el cono sur, específicamente en Argentina, Bolivia, Chile, Paraguay, Perú y Uruguay (Salinas et al, 2020). En chile, G. decorticans se distribuye en el desierto de Atacama, desde la Región de Arica-Parinacota hasta la región de Coquimbo, en un rango altitudinal de 100-2600 m (Rodríguez et al, 2018). Geoffroea decorticans dispersa su polen preferentemente vía polinización entomófila (polinización cruzada y reproducción de tipo xenogamía facultativa), sin embargo, también se ha observado autofecundación parcial (autogamia) (Eynard & Galetto, 2002).
Aislamiento de ADNy secuenciación
Se colectaron hojas frescas de G. decorticans de seis individuos de la Región de Arica-Parinacota (Valle de Azapa y Valle de Chaca), seis individuos de la Región de Tarapacá (poblado de Pachica y poblado de Matilla), seis individuos de Región de Antofagasta (San Pedro de Atacama), seis individuos de la Región de Atacama (Valle de Copiapó, Totoral y Valle Alto del Carmen) y seis individuos de la Región de Coquimbo (Valle de Elqui) (Tabla 1). La identificación taxonómica de los 30 individuos se realizó de acuerdo con las descripciones reportadas por Burkart (1949). Durante la recolección, las muestras se mantuvieron a 4 ° C; posteriormente, se almacenaron a -80 °C en el laboratorio. Se depositaron seis muestras en el herbario del Departamento de Silvicultura y Conservación de la Naturaleza de la Universidad de Chile (EIF, Código Index Herbariorum), bajo el siguiente número de voucher y localidad: EIF13800 (Valle de Chaca), EIF13803 (Valle de Chaca), EIF 13 807 (Valle de Azapa), EIF 13 809 (Valle de Azapa), EIF13812 (Valle de Copiapó) y EIF13815 (Valle de Copiapó).
El ADN se aisló de hojas mediante el protocolo de bromuro de cetil-trimetilamonio modificado (CTAB) descrito por Contreras et al. (2019). La calidad y concentración del ADN genómico extraído de las muestras se verificó mediante el uso de un espectrofotómetro de microvolúmenes Colibri (Titertek-Berthold, Pforzheim, Alemania). Para ello, se utilizó la relación de absorbancia a 260/280 nm para evaluar la pureza del ADN , cuyo valor óptimo es ~ 1,7 y, por otro lado, se utilizó la relación 260/230 como medida secundaria de pureza, cuyo valor aceptable se encuentra entre 2,0 y 2,2 (Demeke & Jenkins, 2010; Aleksic et al, 2012). El ADN extraído de chañar se cuantificó con un fluorómetro Qubit™ 3.0 y un Qubit™ dsDNA HS Assay Kit (Life Technologies, San Diego, CA) de acuerdo con el manual proporcionado por el fabricante. La muestra de ADN de chañar se almacenó a -80 ° C y la integridad del ADN se verificó con un bioanalizador Agilent 2100 (Agilent Technologies, San Diego, CA) antes de la secuenciación. A partir de una muestra de ADN de chañar, las bibliotecas de secuenciación se generaron mediante un kit TruSeq Nano DNA LT (Illumina, San Diego, CA). Las bibliotecas finales se realizaron en un bioanalizador Agilent 2100 para verificar la distribución y concentración del tamaño de los fragmentos. La secuenciación se realizó usando el servicio Genoma Mayor (Universidad Mayor, Chile) con una plataforma de secuenciación Illumina. Las secuencias se enviaron al Centro Nacional de Información Biotecnológica (National Center for Biotechnology Information, NCBI).
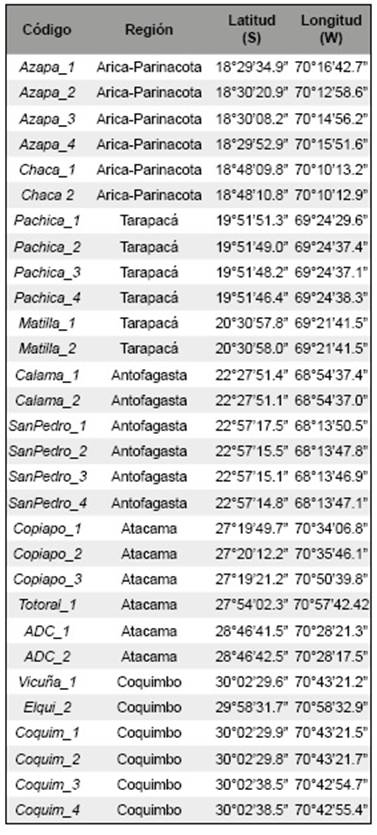
Tabla 1: Localización de muestras de Geoffroea decorticans utilizadas en el presente estudio y sus coordenadas geográficas en Chile.
Búsqueda de SSRs
Las lecturas de secuenciación sin procesar se sometieron a un riguroso proceso de filtrado. Se eliminaron las lecturas con > 10% de bases con una puntuación de calidad de Q <30 (control de calidad Q30), lecturas que representaban ARN no codificante, secuencias ambiguas representadas como “N”, lecturas vacías y contaminantes del adaptador. Para garantizar la precisión y validez de la búsqueda SSR, se filtraron y eliminaron los cóntigos que tenían menos de 300 pb. Las lecturas de las secuencias crudas sentido y antisentido se fusionaron mediante el uso de PEAR versión 0.9.4 (Zhang et al, 2014).
Búsqueda de loci SSR y diseño de cebadores
Se seleccionaron marcadores SSRs en todo el genoma ensamblado mediante el programa MISA (Thiel et al, 2003). Se buscó SSRs cuyos motivos comprendieran secuencias que van desde mono a hexanucleótidos y el número mínimo de unidades repetidas se estableció de la siguiente manera: diez unidades repetidas para mononucleótidos, seis para dinucleótidos y cinco para tri-, tetra-, penta- y hexanucleótidos. Se diseñaron pares de cebadores para los loci de SSR seleccionados utilizando el programa Primer3 (Rozen & Skaletsky, 2000). Los parámetros para el diseño del cebadores incluyó un tamaño de amplicón entre 90-230 pb, un tamaño de cebador entre 18-27 pb, una temperatura de hibridación entre 55-62 ° C y un porcentaje de GC entre 30 y 70%.
Evaluación de nuevos marcadores SSR por PCR
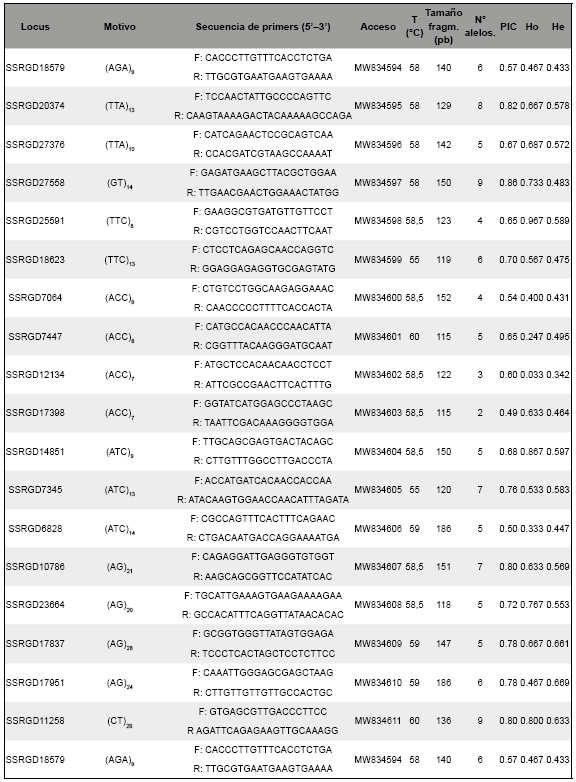
En total, se seleccionaron aleatoriamente 41 pares de cebadores y para la detección de polimorfismos estos se probaron en los 30 individuos de chañar procedentes de 5 sitios. La reacción en cadena de las polimerasas (Polymerase Chain Reaction, PCR) se llevó a cabo en un volumen de 16 pL que contenía 1X de SapphireAmp Fast PCR Master Mix (2X) (Takara-Clontech, EE. UU.), 16 ng de ADN genómico (5 ng/pL), 0,3 pM de cada cebador sentido y antisentido (5 pM) y agua libre de nucleasas. La amplificación por PCR del ADN se realizó en un ciclador térmico Labnet MultiGene OptiMax de acuerdo con el siguiente protocolo: desnaturalización a 94 °C durante 3 min; 40 ciclos de 94 °C durante 25 s, Ta °C (temperatura de hibridación, ver Tabla 3) durante 25 s y 72 °C durante 25 s; y una extensión final a 72 ° C durante 3 min. Los productos de amplificación se analizaron posteriormente mediante electroforesis en geles de agarosa al 3 % teñidos con tinción de ADN GelRed (10.000X, Biotium). Los tamaños de banda se aproximaron basándose en un marcador de ADN de 100 pb (Thermo Fisher).
Validación de marcador SSR
Se usaron 30 individuos de chañar para obtener productos PCR de 41 marcadores SSR y con ello determinar el tamaño de los alelos y el nivel de polimorfismo. Además, se seleccionaron al azar 18 marcadores SSR para confirmar la existencia de los motivos repetidos SSR, a partir de una muestra de ADN de chañar se prepararon productos SSR-PCR, los cuales luego fueron purificados con el Kit Wizard SV Gel and PCR Clean-Up System (Promega, Wisconsin, USA), secuenciados por Macrogen Inc. y analizados con el programa Chromas Pro v1 (Technelysium Pty, Ltd). Se analizó la presencia de alelos nulos en cada locus con el programa MICRO-CHECKER v.2.2.3 (van Oosterhout et al., 2004). Para cada locus, se evaluó el equilibrio de Hardy-Weinberg (HWE) y el desequilibrio de ligamiento (LD) con pruebas de permutación (10.000) entre todos los pares de pruebas de loci utilizando Arlequin v. 3.1 (Excoffier et al, 2005). Los análisis estadísticos de la información de los SSRs, incluido el número de alelos y la frecuencia de los alelos, se calcularon con el programa GenAlEx v. 6.5 (Peakall & Smouse, 2012). El contenido de información de polimorfismo (PIC) para cada locus SSR se estimó de acuerdo con la fórmula PIC = 1 - Epi2, donde pi es la frecuencia de los diferentes alelos detectados en un locus particular. Un valor de PIC de menos de 0,25 indica un polimorfismo bajo, un valor entre 0,25 y 0,5 indica un polimorfismo medio y un valor superior a 0,5 indica un locus altamente polimórfico (Botstein et al., 1980).
Resultados
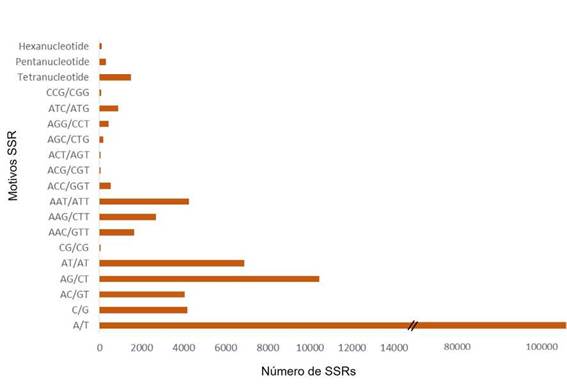
Se identificó un total de 144.117 loci de microsatélites entre los cóntigos ensamblados por el programa MISA (Tabla 2). Las repeticiones de mononucleótidos fueron las más abundantes, representando 109.988 (76,31%) del total de SSR; seguido de repeticiones de dinucleótidos (21.429; 14,86%), repeticiones de trinucleótidos (10.766; 7,47%), repeticiones de tetranucleótidos (1.504; 1,04%), repeticiones de pentanucleótidos (312; 0,21%) y repeticiones de hexanucleótidos (118; 0,08%). La longitud de SSR más frecuente para los mononucleótidos fue de 10 pb (41.104), mientras que la de los dinucleótidos y trinucleótidos fue de 6 pb (4.704) y 5 pb (4.312), respectivamente; en general, con respecto a las seis clases de motivos SSR, la cantidad de loci disminuyó a medida que aumentaba en el número de repeticiones (Fig. 1). De acuerdo con la distribución de microsatélites sobre la base del tipo de motivo, las repeticiones de mononucleótidos A/T estuvieron altamente representadas (105.806) en las secuencias de G. decorticans, mientras que el motivo C/G estaba escasamente representado de acuerdo al número de SSR (4.043) (Fig. 2). Entre las repeticiones en tándem de dinucleótidos, la frecuencia más elevada se observó para los dímeros AG/CT (10.459), seguidos de los dímeros AT/AT (6.879), AC/GT (4.043) y CG/CG (48); los motivos de repetición de trinucleótidos más comunes fueron AAT/ATT (4.244), seguidos de AAG/CTT (2.685), AAC/GTT (1.640), ATC/ATG (885), ACC/GGT (527) y AGG /CCT (437) (Fig. 2).
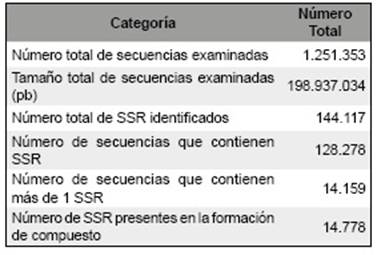
Tabla 2 Resultados de la búsqueda de microsatélites en Geoffroea decorticans, utilizando el software MISA.
De 41 pares de cebadores, un total de 38 (93%) presentaron productos y patrones aceptables de amplificación, sin embargo, tres pares de cebadores (7%; SSRGD10371, SSRGD20347, SSRGD9953) presentaron una débil intensidad de producto de amplificación (Tabla 3), no siendo finalmente considerados en los siguientes análisis. Se logró confirmar la presencia del motivo SSR en la secuencia de ADN de todos los productos de amplificación SSR-PCR (dieciocho seleccionados al azar); en nueve de ellos (SSRGD7064, SSRGD12134, SSRGD17398, SSRGD14851, SSRGD6828, SSRGD23664, SSRGD9382, SSRGD11733 y SSRGD20874) la secuencia de ADN se alineó muy bien a la secuencia de referencia (obtenida por NGS), sin embargo, en las demás secuencias (SSRGD7345, SSRGD4487, SSRGD23813, SSRGD8997, SSRGD8699, SSRGD18623, SSRGD17951, SSRGD17837 y SSRGD11258) se observaron ambigüedades con respecto a la secuencia de referencia. En cuanto a la presencia de alelos nulos, once loci mostraron evidencias de alelos nulos: SSRGD20374, SSRGD7447, SSRGD12134, SSRGD7345, SSRGD6828, SSRGD10786, SSRGD17951, SSRGD3584, SSRGD4487, SSRGD20874 y SSRGD8599. Dieciocho loci presentaron desviaciones significativas de equilibrio de Hardy-Weinberg (P < 0,05) y 11 loci (SSRGD7064, SSRGD7447, SSRGD12134, SSRGD7345, SSRGD6828, SSRGD17951, SSRGD3584, SSRGD16736, SSRGD20874, SSRGD8699 y SSRGD8599) no satisfacen el equilibrio de Hardy-Weinberg, reflejando un déficit en el número de heterocigotos. Las pruebas de desequilibrio de ligamiento fueron significativas en 78 pares de loci para todas las poblaciones (P < 0,05), pero después de la corrección de Bonferroni, ninguno de los pares de loci fue significativo. Los fragmentos amplificados variaron de 106 pb a 225 pb; y el número de alelos varió de 2 a 9, con un promedio de 4,7 (Tabla 3). El valor PIC de los 41 loci SSR osciló entre 0,32 y 0,86, con un promedio de 0,64 (Tabla 3), de los cuales 30 loci SSR exhibieron elevado polimorfismo, 8 loci presentaron moderado polimorfismo y ningún loci manifestó bajo polimorfismo (Tabla 3). Por otro lado, el nivel de la heterocigosis observada (Ho) fue entre 0,033 y 0,967, mientras que la heterocigosis esperada (He) fue entre 0,056 y 0,669. Las 41 secuencias SSR usadas en este estudio fueron depositadas individualmente en la base de datos de GenBank (desde el acceso MW834594 - MW834634), no obstante, las demás secuencias SSRs de G. decorticans (~128.000 secuencias SSR) se ingresaron en la página web “Sequence Read Archive (SRA)” del NCBI, bajo la identificación BioProject ID PRJNA719569 y acceso a BioSample SAMN18613292.
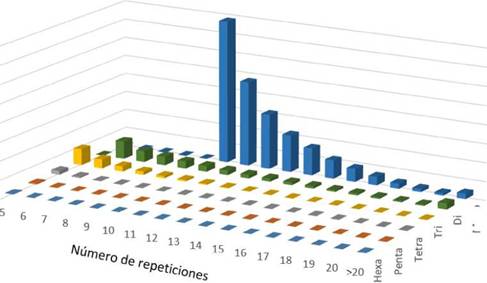
Fig. 1: Distribución de seis clases de motivos SSR (mono a hexanucleótidos) con diferentes números de repeticiones en Geoffroea decorticans.
Discusión
En el presente estudio, se desarrollaron marcadores SSR para G. decorticans a partir de 198
Mb de secuencias obtenidas de la plataforma de secuenciación Illumina. El primer estudio genético realizado en una especie del género Geoffroea utilizando marcadores SSR fue realizado por Naciri-Graven et al. (2005) y Caetano et al. (2012) en la especie G. spinosa. Mientras que los primeros estudios de diversidad genética en poblaciones de G. decorticans se realizaron utilizando marcadores ISSR y RAPD (Contreras et al., 2018) y luego a partir de la transferencia de marcadores SSR desde la especie G. spinosa (Contreras et al., 2019). Con el fin de obtener una mayor resolución en estudios de variabilidad y estructura genética de poblaciones de G. decorticans, era necesario un mayor número de marcadores SSR de la especie, ya que hasta el momento no se habían desarrollado marcadores codominantes específicos para G. decorticans. Más aún, los marcadores aquí desarrollados podrían ser transferibles a otras especies del género Geoffroea, como G. spinosa. Los avances realizados en “Next Generation Sequencing” (NGS) han proporcionado un nuevo escenario para la detección de microsatélites y en las últimas décadas se han desarrollado varios proyectos basados en NGS, generando una enorme cantidad de secuencias disponibles en bases de datos públicas (Vieira et al., 2016). En nuestro estudio se obtuvieron aproximadamente 128.000 secuencias SSR (el resto ~16.000 secuencias obtenidas fueron imperfectas) para G. decorticans mediante secuenciación NGS, lo cual constituyó un número elevado con respecto al número de secuencias SSR obtenidas en otras especies de la familia Fabaceae como Dalbergia odorifera (35.774 secuencias SSR; Liu et al., 2019) y Mimosa scabrella Benth (290 secuencias SSR; Saiki et al, 2017), y un número similar a Acacia koa (130.931 secuencias SSR; Lawson & Ebrahimi, 2018).
En Argentina, el chañar es uno de los árboles más característicos del Chaco Argentino (Giménez & Moglia, 2003). De acuerdo a Orrabalis (2014), el chañar está asociado a otros géneros, como Prosopis, Aphyllanthes y Schinus, entre otros, los cuales están establecidos en la Provincia Chaqueña, Provincia del Monte y Provincia del Espinal en una superficie de 67.490.000 ha, 43.380.000 ha y 33.000.000 ha, respectivamente. Mientras que en todo el territorio chileno la superficie total es de aprox. 2.000 ha (MMA 2021). Sin duda, estas diferencias de superficie hacen importante el estudio de poblaciones de chañar en Argentina, con el fin de construir la primera línea base de la estructura genética de esta valiosa especie. Los 38 loci SSR polimórficos obtenidos en este trabajo también podrían ser útiles para estudiar poblaciones de chañar en Bolivia, Chile, Perú, Paraguay y Uruguay. Además, estos marcadores codominantes podrían identificar y confirmar especies (Dai'nou et al., 2016), subespecies (Besnard et al., 2008) o quizás variantes del género Geoffroea, como por ejemplo G. decorticans var decorticans, G. decorticans var. subtropicalis descrita por Lamarque et al. (2009), o bien las especies G. horsfieldii y G. violaceae la cuales aún no están del todo definidas en el género (Plant List, 2021).
En el norte de Chile, particularmente en el Desierto de Atacama, recientemente se han realizado estudios con marcadores SSR específicos en especies de Fabáceas como Prosopis chilensis, Prosopis alba, Prosopis flexuosa (Moncada et al., 2019; Bessega et al., 2021) y Balsamocarpon brevifolium (Stoll et al., 2020), sin embargo, no se han estudiado marcadores SSR específicos para G. decorticans. En esta última especie, se realizó un estudio con cinco marcadores SSR en ocho poblaciones de chañar (84 individuos en total), de los cuales tres marcadores (Gsp.A149, Gsp.B284 y Gsp.A104) mostraron un elevado polimorfismo (PIC > 0,78) y elevado número de alelos (>9) (Contreras et al., 2019), mientras que en el presente trabajo, para 30 individuos, se observó un elevado polimorfismo (PIC > 0,78) en siete marcadores (SSRGD20374, SSRGD27558, SSRGD10786, SSRGD17837, SSRGD17951, SSRGD11258 y SSRGD12427) y también elevados valores (PIC > 0,5) en 31 marcadores SSR. Por lo tanto, el presente trabajo proporciona un elevado número de marcadores SSR polimórficos exclusivos para el estudio genético de poblaciones de G. decorticans, siendo interesante en el futuro considerarlos para estudios de estructura genética en poblaciones de países del cono sur, sobre todo donde se encuentra más poblado, como Argentina. De todas maneras, sería recomendable que en futuros estudios de diversidad y estructura genética de poblaciones naturales de Geoffroea decorticans se seleccione desde este estudio un set menor de marcadores SSRs y realizar un análisis con un mayor número de individuos, de manera de cuantificar los alelos con cebadores fluorescentes y procesarlos en un analizador de fragmentos. En ese caso, se recomienda utilizar el método de tres cebadores propuesto por Schuelke (2000) para economizar el marcado de los cebadores fluorescentes.
En conclusión, por primera vez se han desarrollado marcadores SSR putativamente neutros para la especie G. decorticans con el fin de apoyar estudios genéticos para la conservación de la especie. El presente estudio provee un conjunto de 38 nuevos marcadores SSR polimórficos, los cuales podrían servir como una herramienta efectiva para estimar diversidad genética, flujo de polen, estructura genética de poblaciones y para ser empleado en programas de mejora.
Contribución de los autores
RC diseñó la investigación, coleccionó el material de campo, dirigió la investigación y registró las muestras en herbario. MA, WHM y BD procesaron las muestras y realizaron los ensayos moleculares. RC, WHM y FC realizaron los análisis de los datos. RC, FC, WHM, MA y BD participaron en la revisión y escritura del manuscrito
Agradecimientos
Este trabajo fue financiado por la Universidad de Atacama en el marco del Proyecto DIUDA 19/18, 22380, y por el proyecto FIC BIP 30432984-0 del Gobierno Regional de Atacama. Agradecemos a la Corporación Nacional Forestal (CONAF) por el permiso de muestreo. Por otro lado, agradecemos los valiosos comentarios realizados por los evaluadores. Además, se agradece el apoyo de Isabel J. Arias, Roberto C. Jordán y César C. Jordán en las labores de muestreo.












 uBio
uBio