Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista argentina de endocrinología y metabolismo
versión On-line ISSN 1851-3034
Rev. argent. endocrinol. metab. vol.51 no.2 Ciudad Autónoma de Buenos Aires jun. 2014
REVISIÓN
microARNs: Nuevos actores en el metabolismode los lípidos
microRNAs: New Factors in Lipid Metabolism
Leonardo Salvarredi.
División Bioquímica Nuclear. Comisión Nacional de Energía Atómica
Correspondencia: Leonardo A. Salvarredi - División Bioquímica Nuclear - Departamento de Radiobiologia - Lab B221-2ºPiso-Edificio Tandar
Centro Atómico Constituyentes - Comisión Nacional de Energía Atómica - Av. Gral. Paz 1499 (B1650KNA) - San Martín-Buenos Aires
+54-011-67727186
Recibido: 07-04-2014
Aceptado: 23-04-2014
RESUMEN
Es reconocido el papel que cumplen los microARNs (miRs) en procesos celulares como la diferenciación, la apoptosis, la proliferación y su alteración en enfermedades como el cáncer. Sin embargo el conocimiento acerca de su función en el metabolismo de los lípidos y desórdenes asociados es escaso. Recientemente se ha visto que estas moléculas desempeñan un papel importante en la homeostasis lipídica, regulando a nivel postranscripcional la expresión de genes involucrados en este metabolismo. También se ha observado que las lipoproteínas, fundamentalmente las lipoproteínas de alta densidad (HDL) son capaces de transportarlos, permitiendo la comunicación celular entre tejidos distantes, estableciéndose así una regulación recíproca. La comprensión de los mecanismos regulatorios involucrados en estos procesos abre nuevas posibilidades al desarrollo de estrategias terapéuticas para el abordaje de desórdenes metabólicos y cardiovasculares. Rev Argent Endocrinol Metab 52:75-84, 2014
Los autores declaran no poseer conflictos de interés.
Palabras clave: MicroARNs; Lípidos; Lipoproteínas; Epigenética.
ABSTRACT
The role of microRNAs (miRs) in cellular processes such as differentiation, apoptosis, and proliferation as well as their alteration in diseases such as cancer are well known. However, there is little knowledge about the role of miRs in lipid metabolism and associated disorders. Recently, it has been shown that these molecules play an important role in lipid homeostasis by regulating post-transcriptional level expression of genes involved in this metabolism. It has also been observed that lipoproteins, mainly high-density lipoproteins (HDL), are capable of transporting miRs, enabling cellular communication between distant tissues, establishing a mutual regulation. Understanding the regulatory mechanisms involved in these processes opens up new possibilities for the development of therapeutic approaches for metabolic and cardiovascular disorders. Rev Argent Endocrinol Metab 51:75-84, 2014
No financial conflicts of interest exist.
Key words: MicroRNAs; Lipids; Lipoproteins; Epigenetic.
INTRODUCCIÓN
Durante la última década una clase de RNA no codificantes, llamados microARNs (miRs), comenzaron a ocupar un lugar crítico en la regulación de la expresión génica. De modo complementario a los mecanismos clásicos de regulación transcripcional, los miRs actúan a nivel postranscripcional. En su forma madura, producto de una compleja vía de procesamiento, son pequeñas moléculas de RNA de cadena simple (de unos 22 nucleótidos) que uniéndose específicamente a la región no traducida del extremo 3`de los RNA mensajeros (mRNAs 3`UTR, untranslated region) inducen su degradación o bloquean su traducción (Fig. 1). Se encuentran ampliamente conservados y ejercen efectos regulatorios en animales, plantas y protozoos(1,2). Descubiertos en C. elegans a la fecha se han identificados cientos de miRs en animales, plantas y virus(3).
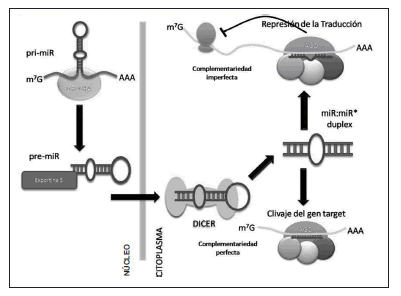
Figura 1. Biogénesis y función de los miRs. lnicialmente los miRs son transcriptos en transcriptos primarios de gran longitud. La secuencia del miR está contenida dentro de una estructura tipo hairpin de 60 a 80 nucleótidos que es clivada por acción de Ia m7G endonucleasa Drosha dando Iugar a un producto intermedio conocido como pre-miR. Este es transportado al citoplasma por medio de Ia exportina 5 donde es procesada por acción de Ia endonucleasa Dicer. Como resultado del clivaje se produce una estructura de doble cadena. Una de las cadenas es cargada selectivamente en el complejo de silenciamiento (RNA-induced silencing complex, RISC) y sirve de guía al complejo hacia el mRNA target induciendo su clivaje y degradación si la complementariedad miR:mRNA es perfecta o la represión de la traducción si la complementariedad es imperfecta
Un miR es capaz de regular simultáneamente distintos mRNAs, del orden de cientos(4) y se cree que todos los miRs identificados hasta el momento podrían modular la expresión de más de un tercio de los mRNAs codificados en el genoma(5-7). Asimismo cada gen puede ser regulado por más de un miR. De esta forma el potencial regulatorio de los miRs es enorme. Abarcan un amplio rango de procesos fisiológicos(8-13), y consecuentemente su desregulación está estrechamente ligada a distintos desórdenes y enfermedades humanas.
En las últimas décadas se ha ampliado la comprensión de los mecanismos involucrados en el metabolismo del colesterol y su participación en la progresión de enfermedades cardiovasculares. Los miRs se ubican actualmente como actores claves de la regulación de procesos homeostáticos.
Comprender cómo estas pequeñas moléculas contribuyen a los mecanismos involucrados en la homeostasis lipídica y su desregulación abre la posibilidad al desarrollo de nuevos blancos y estrategias terapéuticas para el abordaje de desórdenes metabólicos y enfermedades cardiovasculares.
HOMEOSTASIS DEL COLESTEROL
Mediante un proceso conocido como transporte reverso del colesterol (RCT, reverse colesterol transport) las lipoproteínas de alta densidad (HDL, high density lipoproteins) remueven el colesterol contenido en los macrófagos localizados en las paredes de los vasos y lo transportan hasta el hígado para su excreción. Este proceso comienza con la hidrólisis de ésteres de colesterol citoplasmático mediante la acción de hidrolasas y a través de lipasas lisosomales(14). El colesterol libre luego efluye desde la célula por difusión pasiva y, por transporte activo, a través de los transportadores ABC A1 (ABCA1) y G1 (ABCG1) es transferido a las lipoproteínas apoA1 y HDL respectivamente. Estos mismos transportadores intervienen en la biogénesis del HDL en el hígado y su maduración en el plasma(15).
Mientras adquieren colesterol y fosfolípidos las HDL atraviesan distintos eventos de remodelación bajo la acción de colesterol transferasas (LCAT, lecithin-cholesterol acyltransferase) o lipasas hepáticas y/o endoteliales que en su curso van alterando la composición y el tamaño de las lipoproteínas(16). Las HDLs pueden intercambiar el colesterol por triglicéridos procedente de lipoproteínas que contienen la proteína apoB por medio de la acción de la proteína transferidora de ésteres de colesterol (CETP, cholesteryl ester transfer protein). De esta forma el colesterol puede ser tomado por el hígado a través del receptor de lipoproteínas de baja densidad (LDLR, low density lipoprotein receptor)(16).
En el último paso de la vía canónica del transporte reverso las HDLs trasladan su contenido lipídico al hígado donde puede ingresar a través del receptor SR-BI (scavenger receptor class B, type I)(17). Una vez en el hígado el colesterol que ha ingresado tanto a través de los receptores de LDL o los SR-BI puede ser oxigenado y convertido en sales biliares que luego serán secretadas al intestino a través de transportadores canaliculares(18).
Tanto el proceso de eflujo y remoción en condiciones de exceso de colesterol como la biosíntesis y la internalización de colesterol exógeno están regulados por la expresión de distintos genes. Una familia importante de proteínas son las proteínas de unión a elementos regulados por esterol (SREBPs, ER-bound sterol regulatory element-binding proteins)(19). Esta familia de proteínas consiste en 3 proteínas codificadas por los genes Srebp-1 y Srebp-2. El gen Srebp-1 genera por remoción de intrones (splicing) 2 transcriptos alternativos, Srebp-1a y Srebp-1c. Las proteínas SREBPs difieren en su expresión tejido específico, la selectividad de los genes blanco y la potencia relativa de sus dominios de transactivación. SREBP1c regula la transcripción de genes involucrados en el metabolismo de los ácidos grasos, como la sintasa de ácidos grasos (FASN, fatty acid sinthase). SREBP2 y SREBP1a regulan la transcripción de genes asociados al colesterol, como la reductasa HMGCR (3-hydroxy-3-methylglutaryl coenzyme A reductase), la cual cataliza un paso limitante en la biosíntesis del colesterol, y el receptor de LDL, el cual importa colesterol desde la sangre(19).
Otro paso importante en la transcripción para el mantenimiento de la homeostasis del colesterol lo constituyen los receptores X del hígado (LXR, liver X receptors), que regulan la respuesta al exceso de colesterol(20) LXRα y LXRβ son factores de transcripción activados por ligando pertenecientes a la familia de receptores nucleares de hormonas y son activados por metabolitos endógenos oxidados que derivan del colesterol, llamados oxisteroles. Estos factores de transcripción son activados en respuesta a niveles elevados de colesterol e inducen la expresión de proteínas involucradas en la absorción de colesterol, transporte, excreción y eflujo, incluyendo a los transportadores ABCA1 y ABCG1(20).
PAPEL DE LOS MIRS
Muchos genes involucrados en la biogénesis de HDL, el eflujo celular del colesterol, el ingreso selectivo al hígado desde las lipoproteínas y el transporte biliar son potenciales blancos para los miRs. Algunos son capaces de regular múltiples genes de la vía e inversamente algunos genes claves pueden ser regulados por distintos miRs.
Hasta el momento los miRs 122 y 33 han sido identificados como reguladores claves del metabolismo de los lípidos (Fig.2 ). Recientemente se ha reportado que otro miR, el miR30c cumple también una función en la homeostasis lipídica(21).

Figura 2. Regulación de Ia homeostasis de lípidos por miRs. Las puntas de flecha rectangulares indican Ia represión postranscripcional de los genes regulados por el miR33. La represión de genes por parte de los miR22 y 30 c produce un incremento y una reducción de los niveles plasmáticos de LDL respectivamente.
miR 122
Es el miR más abundante en el hígado, comprende el 70 % de la totalidad de miRs expresados y está altamente conservado en distintas especies. Inicialmente fue estudiado en procesos de respuesta a estrés o depleción de aminoácidos. Sin embargo. se desconocía su papel en la fisiología normal del hígado. Posteriormente, en estudios in vivo en ratón se realizaron análisis funcionales(22,23). Silenciando específicamente al miR por medio de oligonucleótidos antisentido seguido de un análisis de expresión por microarrays se observó la desregulación de más de 100 genes blanco conteniendo sitios de unión específicos para el miR122. A nivel funcional se observó una reducción de los niveles de colesterol totales del plasma. En ratones con dieta rica en grasas se demostró una reducción del contenido de triglicéridos en hígado y un aumento de la boxidación de ácidos grasos. Estas observaciones resultaron coherentes con la represión por parte de este miR de la enzima FASN. Para hacer extensivos estos resultados a otros modelos, se practicaron estudios de silenciamiento en primates no humanos. Con oligonucleótidos antisentido se realizaron estudios in vivo inhibiendo al miR122 en monos verdes Africanos(24) y chimpancés(25). Se observó en forma similar a lo sucedido en el modelo murino, una reducción en los niveles de colesterol totales en un rango de entre el 20 al 30 %.
La aplicación clínica de estos resultados hoy está allanada por estudios que involucran al miR122 como parte de una estrategia terapéutica en pacientes con hepatitis C. El miR122 contiene 2 sitios de unión a la región 5` no codificante del genoma del virus, que resultan esenciales para la acumulación y propagación del virus en hepatocitos(26,27). En estudios realizados en primates no humanos donde se silenció al miR se observó una mejora en la patología y reducción en la viremia(25). Estos resultados han impulsado la primer aplicación clínica basada en miRs sobre pacientes con hepatitis C que a la fecha se encuentra en fase II(28).
miR 33
Aunque la localización genómica del miR 33 había sido reportada en 2004(29) las consecuencias funcionales de su papel en el metabolismo de los lípidos no se conocieron sino hasta 2010(30-32). Tres laboratorios por distintas caminos determinaron que este miR constituía un regulador clave en el metabolismo del colesterol. Se observó por medio de un análisis de expresión por microarrays que la expresión del miR estaba regulada por el contenido de colesterol en macrófagos(32). En condiciones de depleción o enriquecimiento de colesterol se determinó que el miR estaba positiva o negativamente regulado respectivamente, correlacionado a la expresión del gen Srebf2 (Sterol Regulatory Element-Binding Factor 2). También se observó en ratones que la dieta rica en colesterol alteraba los niveles de expresión del miR. Estos datos sugirieron una coregulación de ambos genes. En otro estudio por análisis in silico se observó que el miR estaba localizado en un intrón del gen Srebf2. Más interesante aún, este grupo mostró explicando la corregulación de ambos genes, que el miR33 se cotranscribe junto a Srebf2 tanto en macrófagos como hepatocitos(30,31).
Para explicar el efecto del silenciamiento del miR33 sobre los niveles de colesterol total se analizaron genes blanco del miR que pudieran estar asociados al metabolismo del colesterol. El principal candidato encontrado resultó ser el transportador ABCA1, responsable del movimiento de colesterol al medio extracelular. La región 3`UTR del mRNA de ABCA1 contiene 3 sitios de unión al miR33 (en sus dos isoformas miR33a y miR33b). El análisis funcional mostró que la sobreexpresión del miR reprimió la expresión de ABCA1 e inversamente la inhibición del miR resultó en un incremento de la expresión del transportador(30-32). La mutación de los sitios de unión del miR en la región 3`UTR de ABCA1 restauró los niveles de expresión(30-32). Producto de la inhibición también se observó en macrófagos y hepatocitos, conjuntamente al aumento de expresión del transportador, un aumento del eflujo de colesterol a apoA1. De modo inverso la sobreexpresión del miR produjo una disminución del eflujo de colesterol a apoA1(30-32).
Además de ABCA1, se observó en el modelo murino que el miR33 inhibe la expresión del transportador ABCG1, responsable del transporte de colesterol hacia HDL(30-32). Llamativamente, la región 3`UTR del mRNA de ABCG1 en ratón contiene 2 sitios de unión al miR33, ausentes en humanos. Esto explica porqué la sobreexpresión del miR en células de origen humano no inhibe la expresión del transportador. De modo inverso, la proteína NPC1 (Neimann Pick C1) a cargo del transporte del colesterol desde compartimentos lisosomales a otros compartimentos, y que actúa coordinadamente con ABCG1 en el eflujo de colesterol a HDL, es reprimida por el miR33 en células de origen humano pero no en murinas(32). Esto podría deberse a que el mRNA humano contiene 2 sitios de unión al miR y el murino solo uno de ellos. De esta forma se demostró la versatilidad que poseen los miRs para producir un efecto similar en diversas especies regulando distintos genes blanco pertenecientes a la misma vía.
Como consecuencia de la regulación de los transportadores ABCA1 y ABCG1 se pensó que esto debería tener un efecto sobre los niveles de HDL circulantes. Mediante el silenciamiento in vivo del miR33 en ratones se observó un aumento del 25 % en los niveles plasmáticos de esta lipoproteína. De modo inverso al sobreexpresar el miR se determinó una disminución entre el 25-30 % en los niveles de HDL circulante(30-32).
Dando más sustento al papel regulatorio del miR33 sobre los niveles de HDL circulantes Horie y col. (2010) utilizaron ratones knock out para la expresión del miR33, manteniendo intacta la expresión de Srebf2. Observaron un aumento de la expresión de ABCA1 y un aumento de entre el 25-40 % en los niveles de HDL(33).
Además de promover el eflujo de colesterol y la biosíntesis de HDL, estudios en ratón demostraron que el miR33 es capaz de regular la secreción biliar en el hígado(34). El miR regula a través de la unión a la región 3`UTR los transportadores ABCB1 y ATP8B1, ubicados en las membranas canaliculares y claves en la secreción biliar. En experimentos in vivo también se observó una alteración en la secreción biliar y el contenido de esteroles(34).
Por otra parte, se observó que genes involucrados en la boxidación de ácidos grasos como CPT1a, CROT y HADHB(35) presentan sitios de unión altamente conservados para el miR33. Se determinó la unión específica del miR33 a los sitios de unión de la región 3`UTR de estos genes y la represión de CPT1a y HADHB(32).
Todos estos resultados sustentan con mayor fuerza el papel clave del miR33 en la regulación del transporte reverso de colesterol y en consecuencia su potencial terapéutico en enfermedades cardiovasculares como la aterosclerosis. En ratones knock out para el receptor de LDL (Ldlr-/-) con placas ateroscleróticas establecidas, la inhibición del miR33 produjo una regresión de las lesiones ateroscleróticas, reduciendo el tamaño de las placas, el contenido lipídico y de macrófagos y la expresión de genes proinflamatorios(36). En otro estudio en macrófagos peritoneales de ratones knock out para miR33 y apoE (mir33-/- ;apoE-/-) se observó un incremento en el eflujo de colesterol a apoA1 y HDL respecto a macrófagos con la expresión normal del miR(37). En el mismo estudio los ratones fueron alimentados por 14 semanas con una dieta conteniendo un 0,15 % de colesterol y se observó en los ratones knock out para el miR33 una reducción del 20-25 % en el tamaño de las placas y el contenido lipídico respecto a los ratones con la expresión del miR intacta(37).
La proyección terapéutica del miR33 alcanza también la función de las células bde los islotes del páncreas. Se ha observado que en individuos con una combinación de defectos en estas células y en los lípidos plasmáticos los altos niveles de colesterol afectan la función de las células by la tolerancia a la glucosa. La inhibición del miR33, con el incremento de la expresión de ABCA1, resultó en un aumento de la remoción del colesterol de estas células y la restauración de los niveles normales de insulina(38).
miR30c
Aunque los niveles de expresión de este miR en hígado son relativamente bajos respecto a otros tejidos Soh y col. (2013) plantearon una amplia evidencia respecto a su papel en la homeostasis lipídica. El hallazgo de este miR surgió en la búsqueda de inhibidores de la proteína transferidora de triglicéridos microsomales (MTP, microsomal triglyceride transfer protein). Esta proteína, involucrada en el ensamblaje de precursores de lipoproteínas de baja densidad, interactúa y lipidifica a la proteína apoB, haciendo de ella un blanco terapéutico para reducir los niveles plasmáticos de los lípidos. Sin embargo, la utilización de inhibidores ha dado lugar a efectos secundarios como la esteatosis y el incremento en los niveles plasmáticos de transaminasas. Mediante el análisis in silico se encontró que los miembros de la familia del miR30 contenían sitios de unión a los transcriptos de MTP y además se encontraban conservados entre los vertebrados. Estudios funcionales in vitro demostraron que, de los 3 miembros de la familia, debido a la presencia de sitios de interacción suplementarios al sitio de interacción principal(39), solo el miR30c tenía un efecto sobre la actividad y los niveles de expresión de la proteína blanco. Se observó in vitro que la sobreexpresión e inhibición del miR no solo reducía y aumentaba, respectivamente, los niveles de expresión y actividad de la proteína sino que, como producto de estos cambios, también variaban del mismo modo, los niveles de secreción de apoB en el medio. En estudios in vivo, en ratones en los que se sobreexpresaba o inhibía la expresión de los miRs en hígado, se observó también un incremento y reducción respectivamente en los niveles de expresión de la proteína blanco y en los niveles plasmáticos de colesterol y triglicéridos, como resultado de la reducción en los niveles de secreción de lipoproteínas no HDL. Se observó además que estos cambios se producían sin variar los niveles plasmáticos de transaminasas. Cuando en el mismo experimento se analizaron los niveles de colesterol y triglicéridos, pero esta vez en los homogenatos de hígado, contrariamente a lo esperado, no se observó un incremento en la concentración hepática de los mismos. Se buscaron posibles genes blanco del miR en vías de oxidación y síntesis de triglicéridos y fosfolípidos. In vitro, la sobreexpresión del miR redujo entre otros genes los niveles de expresión de la enzima lisofosfatidilglicerol aciltransferasa (LPGAT1, lysophosphatidylglycerol acyltransferase 1), que por estudios de silenciamiento mostró tener un rol en la lipogénesis de novo sin afectar la actividad de MTP. Para estudiar el efecto in vivo, ratones knock out para MTP fueron transfectados con el miR30c y no se observaron cambios en los niveles plasmáticos del colesterol y los triglicéridos, ni en el colesterol hepático, pero sí en los niveles de los triglicéridos, ácidos grasos y fosfolípidos. Esto indicó entonces que el miR era capaz de reducir los niveles de triglicéridos hepáticos independientemente de la actividad de MTP. Para resaltar el alcance terapéutico de este miR se estudio el efecto sobre la aterosclerosis. Ratones knock out para apoE se transfectaron con el miR, con el inhibidor o con un control scrambled. Se observó una reducción y un incremento en las concentraciones plasmáticas de los lípidos y de la proteína apoB, sin cambios en los niveles de las transaminasas, en los ratones en los que la expresión del miR estaba aumentada o disminuida respectivamente. Asimismo se observó una reducción y un incremento en el número y tamaño de placas ateroscleróticas y en la tinción de macrófagos en las uniones cardíacas de la aorta en los ratones en los que el miR estaba aumentado y disminuido respectivamente.
De esta manera los autores demostraron que el miR30c era capaz, al disminuir la expresión de MTP, de reducir los niveles plasmáticos de colesterol y triglicéridos sin producir efectos secundarios, reducir los niveles hepáticos de los triglicéridos, disminuyendo la lipogénesis de novo al reducir la expresión de algunos genes blancos como LPGAT1 y finalmente reducir también la formación de placas ateroscleróticas en ratones knock out para apoE.
miRs circulantes
El alcance regulatorio de los miRs va mas allá de la célula de origen. Diversos estudios han reportado la existencia de miRs circulantes en el plasma y si bien en un principio se pensaba que era un proceso pasivo y azaroso producto de la muerte celular, hoy se sabe que en realidad es principalmente un proceso activo, controlado y específico, aunque hasta el momento los mecanismos involucrados en su secreción resultan desconocidos. Los miRs pueden ser transportados en microvesículas, exosomas, cuerpos apoptóticos, proteínas libres y en lipoproteínas HDLs(40) (Fig. 3). Hay una amplia variedad de trabajos que establecen una relación estrecha entre estos miRs y distintas patologías(41-43). Esto ha generado un interés particular en la utilización de estas pequeñas moléculas como biomarcadores de distintas patologías.
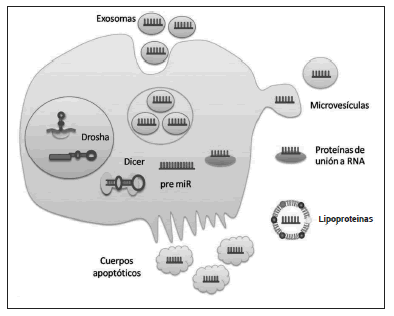
Figura 3. miRs circulantes. Los miRs pueden ser secretados a Ia circulación en distintas vesículas lipídicas como exosomas, microvesiculas y cuerpos apoptóticos o pueden encontrarse fuera de las vesículas pero unidas a lipoproteínas o a proteínas de unión a RNA
Las HDLs son capaces de transportar miRs endógenos hasta células receptoras por medio de un mecanismo que involucra a los receptores SR-BI(44). Se ha observado además que las HDL de pacientes con hipercolesterolemia familiar (FHC) presentan mayor abundancia y riqueza de miRs que aquellos procedentes de pacientes normales(44) El análisis de los miRs contenidos en las HDLs mostró que el miR223 se encuentra altamente enriquecido y llamativamente este miR se encuentra en forma abundante en células receptoras con la consiguiente reducción en los niveles de dos genes target: RhoB y EFNA1. Inclusive cuando se trataron hepatocitos con estas HDLs se observó una regulación negativa de 91 genes, 79 de los cuales eran blancos tentativos de los 22 miRs más abundantes contendidos en las lipoproteínas(44). La heterogeneidad de efectos vasculares de los HDLs en la regulación del óxido nítrico y funciones antioxidantes, antiinflamatorias y antitrombóticas hace pensar que esto puede deberse al perfil de miRs contenidos en las lipoproteínas.
De esta manera, los miRs no solo regulan genes clave del metabolismo de los lípidos, particularmente involucrados en la homeostasis del colesterol y los niveles de HDL circulantes, sino que estas mismas lipoproteínas son responsables de su transporte y en última instancia de sus efectos regulatorios en tejidos distantes.
Inclusive se ha reportado que los miRs no solo ejercen efectos regulatorias a distancia entre tejidos distantes de un organismo sino que pueden hacerlo entre distintos organismos, inclusive de distintos reinos. En un trabajo reciente(45) se describió que un miR de origen vegetal, abundante en arroz, el miR168a, aumentó su nivel en plasma tras la ingesta de arroz, y fue capaz de unirse al mRNA de la proteína adaptadora 1 del receptor de LDL, inhibir su expresión y como consecuencia de ello alterar los niveles de colesterol en suero. Este trabajo evidencia cómo un miR de origen vegetal en la dieta es capaz de regular la expresión de un gen target en mamíferos.
CONCLUSIONES
Los miRs regulan una amplia variedad de procesos fisiológicos incluyendo al metabolismo de los lípidos. Consecuentemente su desregulación está también asociada a dislipidemias. Como se describe anteriormente, se han identificado tres miRs que regulan la homeostasis de los lípidos: los miRs122, 33 y 30c. Se ha demostrado que la expresión de estos miRs está determinada por cambios en el ambiente celular, incluyendo los niveles de colesterol, y estos procesos se correlacionan con la inducción de otros genes que regulan la homeostasis lipídica. Los estudios funcionales, en los cuales se silencia por distintas vías la expresión de los miRs tanto in vitro como in vivo, incluyendo a modelos primates no humanos muestran resultados muy promisorios. Además, teniendo en cuenta la diversidad de genes involucrados en estos procesos y de acuerdo a las predicciones bioinformáticas, se espera que en los próximos años sea aún mayor la cantidad de miR asociados a estos procesos regulatorios.
Sin embargo, la aplicación clínica de este conocimiento tiene por delante importantes desafíos. Debe tenerse en cuenta, en primer lugar, que un miR es capaz de regular simultáneamente la expresión de una amplia variedad de genes. En términos fisiológicos, esto significa que cuando se silencia o sobreexpresa un miR se puede estar inhibiendo la expresión de un gen blanco de interés conocido y simultáneamente estar inhibiendo (o activando como resultado indirecto de la inhibición de un represor) otros genes blancos desconocidos. En segundo lugar la expresión de los miRs es tejido específica e incluso en distintos tejidos un miR puede cumplir distintas funciones de acuerdo a los genes expresados por cada tejido. Teniendo en cuenta todo esto el desarrollo terapéutico de los miRs debe involucrar entonces la producción de conocimiento básico: comprender los mecanismos detrás de los efectos fisiológicos observados e identificar y validar funcionalmente los genes blanco de estos miRs. Asimismo este conocimiento básico debe contrastarse con estudios funcionales de silenciamiento o sobreexpresión en modelos murinos y en primates no humanos. La tolerancia a los efectos del tratamiento con antagonistas del miR122 observada en pacientes con hepatitis C es un paso importante en el desarrollo de estrategias farmacológicas a base de miRs.
1. Ambros V. The functions of animal microRNAs. Nature 431:350-55, 2004 [ Links ]
2. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 116:281-97, 2004 [ Links ]
3. Berezikov E, Cuppen E, Plasterk RH. Approaches to microRNA discovery. Nat. Genet. 38(Suppl.):S2-7, 2006 [ Links ]
4. Friedman RC, Farh KK, Burge CB, Bartel DP. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19:92-105, 2009 [ Links ]
5. Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB. Prediction of mammalian microRNA targets. Cell 115:787-798, 2003 [ Links ]
6. John B, Enright AJ, Aravin A, Tuschl T, Sander C, Marks DS. Human MicroRNA targets. PLoS Biol 2:e363, 2004 [ Links ]
7. Kiriakidou M, Nelson PT, Kouranov A, Fitziev P, Bouyioukos C, Mourelatos Z, Hatzigeorgiou A. A combined computational experimental approach predicts human microRNA targets. Genes Dev 18:1165-1178, 2004 [ Links ]
8. Bushati N, Cohen SM. microRNA functions. Annu. Rev. Cell Dev. Biol. 23:175-205, 2007 [ Links ]
9. Chang TC, Mendell JT. microRNAs in vertebrate physiology and human disease. Annu. Rev. Genomics Hum. Genet. 8:215-39, 2007 [ Links ]
10. Esquela-Kerscher A, Slack FJ. Oncomirs-microRNAs with a role in cancer. Nat. Rev. Cancer 6:259-69, 2006 [ Links ]
11. Krutzfeldt J, Stoffel M. MicroRNAs: a new class of regulatory genes affecting metabolism. Cell Metab. 4:9-12, 2006 [ Links ]
12. Lynn FC. Meta-regulation: microRNA regulation of glucose and lipid metabolism. Trends Endocrinol. Metab. 20:452-59, 2009 [ Links ]
13. Suarez Y, Sessa WC. MicroRNAs as novel regulators of angiogenesis. Circ. Res. 104:442-54, 2009 [ Links ]
14. Ouimet M, Marcel YL. Regulation of lipid droplet cholesterol effl ux from macrophage foam cells. Arterioscler. Thromb.Vasc. Biol. 32:575-581, 2012 [ Links ]
15. Krimbou L., Marcil M, Genest J. New insights into the biogenesis of human high-density lipoproteins. Curr. Opin. Lipidol. 17:258-267, 2006 [ Links ]
16. Von Eckardstein A, Nofer JR, Assmann G. High density lipoproteins and arteriosclerosis. Role of cholesterol efflux and reverse cholesterol transport. Arterioscler. Thromb. Vasc. Biol. 21:13-27, 2001 [ Links ]
17. Krieger M. Charting the fate of the "good cholesterol": identification and characterization of the high-density lipoprotein receptor SR-BI. Annu. Rev. Biochem. 68:523-558, 1999 [ Links ]
18. Wolkoff AW, Cohen DE. Bile acid regulation of hepatic physiology: I. Hepatocyte transport of bile acids. Am. J. Physiol. Gastrointest. Liver Physiol. 284:G175-G179, 2003 [ Links ]
19. BrownMS, Goldstein JL.The SREBPpathway: regulation of cholesterol metabolism by proteolysis of a membrane-bound transcription factor. Cell 89:331-40, 1997 [ Links ]
20. Zelcer N, Tontonoz P. Liver X receptors as integrators of metabolic and inflammatory signaling. J. Clin. Invest. 116:607-14, 2006 [ Links ]
21. Soh J, Iqbal J, Queiroz J, Fernandez-Hernando C, Hussain MM. MicroRNA-30c reduces hyperlipidemia and atherosclerosis in mice by decreasing lipid synthesis and lipoprotein secretion. Nat. Med. 19:892-900, 2013 [ Links ]
22. Elmén J, Lindow M, Silahtaroglu A, Bak M, Christensen M, Lind-Thomsen A, Hedtjärn M, Hansen JB, Hansen HF, Straarup EM, McCullagh K, Kearney P, Kauppinen S. Antagonism of microRNA-122 in mice by systemically administered LNA-antimiR leads to up-regulation of a large set of predicted target mRNAs in the liver. Nucleic Acids Res. 36:1153-1162, 2008 [ Links ]
23. Esau C, Davis S, Murray SF, Yu XX, Pandey SK, Pear M, Watts L, Booten SL, Graham M, McKay R, Subramaniam A, Propp S, Lollo BA, Freier S, Bennett CF, Bhanot S, Monia BP. miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting. Cell Metab. 3:87-98, 2006 [ Links ]
24. Elmén J, Lindow M, Schütz S, Lawrence M, Petri A, Obad S, Lindholm M, Hedtjärn M, Hansen HF, Berger U, Gullans S, Kearney P, Sarnow P, Straarup EM, Kauppinen S. LNA-mediated microRNA silencing in non-human primates. Nature 452:896-899, 2008 [ Links ]
25. Lanford RE, Hildebrandt-Eriksen ES, Petri A, Persson R, Lindow M, Munk ME, Kauppinen S, Ørum H. Therapeutic silencing of microRNA-122 in primates with chronic hepatitis C virus infection. Science 327:198-201 non-human primates. Nature 452:896-899, 2010 [ Links ]
26. Jopling CL, Schutz S, Sarnow P. Position-dependent function for a tandem microRNA miR-122-binding site located in the hepatitis C virus RNA genome. Cell Host Microbe 4:77-85, 2008 [ Links ]
27. Jopling CL, Yi M, Lancaster AM, Lemon SM, Sarnow P. Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA. Science 309:1577-1581, 2005 [ Links ]
28. Franciscus A. Hepatitis C treatments in current clinical development. HCV Advocate.http://www.hcvadvocate.org/hepatitis/hepC/HCVDrugs.html, 2010 [ Links ]
29. Chang J, Nicolas E, Marks D, Sander C, Lerro A, Buendia MA, Xu C, Mason WS, Moloshok T, Bort R, Zaret KS, Taylor JM. miR-122, a mammalian liver-specific microRNA, is processed from hcr mRNA and may downregulate the high affinity cationic amino acid transporter CAT-1. RNA Biol. 1:106-113, 2004 [ Links ]
30. Marquart TJ, Allen RM, Ory DS, Baldan A. miR-33 links SREBP-2 induction to repression of sterol transporters. Proc. Natl. Acad. Sci. USA 107:12228-12232, 2010 [ Links ]
31. Najafi-Shoushtari SH, Kristo F, Li Y, Shioda T, Cohen DE, Gerszten RE, Näär AM. MicroRNA-33 and the SREBP host genes cooperate to control cholesterol homeostasis. Science 328:1566-1569, 2010 [ Links ]
32. Rayner KJ, Suárez Y, Dávalos A, Parathath S, Fitzgerald ML, Tamehiro N, Fisher EA, Moore KJ, Fernández-Hernando C. MiR-33 contributes to the regulation of cholesterol homeostasis. Science 328:1570-1573, 2010 [ Links ]
33. Horie T, Ono K, Horiguchi M, Nishi H, Nakamura T, Nagao K, Kinoshita M, Kuwabara Y, Marusawa H, Iwanaga Y, Hasegawa K, Yokode M, Kimura T, Kita T. MicroRNA-33 encoded by an intron of sterol regulatory element-binding protein 2 (Srebp2) regulates HDL in vivo. Proc.Natl. Acad. Sci. USA 107:17321-17326, 2010 [ Links ]
34. Allen RM, Marquart TJ, Albert CJ , Suchy FJ, Wang DQ, Ananthanarayanan M , Ford DA, Baldan A. miR-33 controls the expression of biliary transporters, and mediates statin- and diet-induced hepatotoxicity. EMBO Mol. Med. 4:882-895, 2012 [ Links ]
35. Gerin I, Clerbaux LA, Haumont O, Lanthier N, Das AK, Burant CF, Leclercq IA, MacDougald OA, Bommer GT . Expression of miR-33 from an SREBP2 intron inhibits cholesterol export and fatty acid oxidation. J. Biol. Chem. 285:33652-33661, 2010 [ Links ]
36. Rayner KJ. , Sheedy FJ, Esau CC, Hussain FN, Temel RE, Parathath S, van Gils JM, A. J. Rayner AJ, A. N. Chang AN, Suarez Y. Antagonism of miR-33 in mice promotes reverse cholesterol transport and regression of atherosclerosis. J. Clin. Invest.121:2921-2931, 2011 [ Links ]
37. Horie T, Baba O, Kuwabara Y, Chujo Y, Watanabe S, Kinoshita M, Horiguchi M, Nakamura T, Chonabayashi K, Hishizawa M, Hasegawa K, Kume N, Yokode M, Kita T, Kimura T, Ono K. . MicroRNA-33 defi ciency reduces the progression of atherosclerotic plaque in ApoE( -/ - ) mice. J. Am.Heart Assoc. 1:e003376, 2012 [ Links ]
38. Wijesekara N, Zhang LH , Kang MH, Abraham T, Bhattacharjee A,Warnock GL, Verchere CB, Hayden MR. miR-33a modulates ABCA1 expression, cholesterol accumulation, and insulin secretion in pancreatic islets. Diabetes 61:653-658, 2012 [ Links ]
39. Soh J, Hussain MM. Supplementary site interactions are critical for the regulation of microsomal triglyceride transfer protein by microRNA-30c. Nutr. Metab. 4;10(1):56, 2013 [ Links ]
40. Zhu H, Fan GC. Extracellular/circulating microRNAs and their potential role in cardiovascular disease. Am. J. Cardiovasc. Dis. 1:138-149, 2011 [ Links ]
41. Mitchell PS, Parkin RK, Kroh EM, Fritz BR, Wyman SK, Pogosova-Agadjanyan EL, Peterson A, Noteboom J, O'Briant KC, Allen A, Lin DW, Urban N, Drescher CW, Knudsen BS, Stirewalt DL, Gentleman R, Vessella RL, Nelson PS, Martin DB, Tewari M. Circulating microRNAs as stable blood-based markers for cancer detection. Proc Natl Acad Sci USA 105:10513-10518, 2008 [ Links ]
42. Gilad S, Meiri E, Yogev Y, Benjamin S, Lebanony D, Yerushalmi N, Benjamin H, Kushnir M, Cholakh H, Melamed N, Bentwich Z, Hod M, Goren Y, Chajut A. Serum microRNAs are promising novel biomarkers. PLoS ONE 3:e3148, 2008 [ Links ]
43. Chen X, Ba Y, Ma L, Cai X, Yin Y, Wang K, Guo J, Zhang Y, Chen J, Guo X, Li Q, Li X, Wang W, Zhang Y, Wang J, Jiang X, Xiang Y, Xu C, Zheng P, Zhang J, Li R,Zhang H, Shang X, Gong T, Ning G, Wang J, Zen K, Zhang J, Zhang CY. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res. 18:997-1006, 2008 [ Links ]
44. Vickers KC, Palmisano BT, Shoucri BM, Shamburek RD, Remaley AT . MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nat. Cell Biol. 13: 423-433, 2011 [ Links ]
45. Lin Zhang, Dongxia Hou, Xi Chen, Donghai Li, Lingyun Zhu, Yujing Zhang, Jing Li, Zhen Bian, Xiangying Liang, Xing Cai, Yuan Yin, Cheng Wang, Tianfu Zhang, Dihan Zhu, Dianmu Zhang, Jie Xu, Qun Chen, Yi Ba, Jing Liu, Qiang Wang, Jianqun Chen, Jin Wang, Meng Wang, Qipeng Zhang, Junfeng Zhang, Ke Zen and Chen-Yu Zhang. Exogenous plant MIR168a specifically targets mammalian LDLRAP1: evidence of cross-kingdom regulation by microRNA Cell Research 22:107-126, 2012 [ Links ]