Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Ciencia y Tecnología
versión On-line ISSN 1851-7587
Rev. cienc. tecnol. no.14 Posadas jul./dic. 2010
GENÉTICA-BIOLOGÍA
Estandarización de un método para la extracción de DNA a partir de muestras clínicas parafinadas
Standarization of a method for paraffin-embedded clinic samples DNA extraction
María M. Tiscornia, Marisa A. Cubilla, María A. Lorenzati, Ariel E. Cariaga Martínez, Pedro D. Zapata
1. Laboratorio de Biotecnología Molecular (BIOTECMOL), Módulo de Bioquímica y Farmacia, Facultad de Ciencias Exactas Químicas y Naturales, UNaM, Posadas, Misiones, Argentina. Av. Mariano Moreno 1375 (3300), Posadas, Misiones, Argentina. (Bcmb@fceqyn.unam.edu.ar)
• María Mercedes Tiscornia1 Es Licenciada en Genética y alumna de Doctorado en Bioquímica en la Universidad Nacional de Tucumán. Actualmente es Auxiliar de Primera Regular de la Cátedra de Biología Molecular y Genética desde el año 2010. Es Becaria del CONICET Categoría I. Se desempeña como Investigador del Proyecto PICTO 37029 financiado por la ANPCyT, habiendo participado además del proyecto financiado por la ANPCyT (PICT 05-15058). Realiza tareas de extensión y servicio en el Laboratorio de Biotecnología Molecular. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• Marisa Angélica Cubilla1 Es Licenciada en Genética y alumna de Doctorado. Se desempeña como Auxiliar Alumno de la Cátedra de Biología Molecular y Genética desde el año 2006. Ha participado del Proyecto PICT 05-15058 financiado por la ANPCyT. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• Ariel Ernesto Cariaga Martinez1 Es Bioquímico y alumno de Doctorado de la Universidad de Alcalá de Henares, España. Se ha desempeñado como Auxiliar Alumno de la Cátedra de Biología Molecular y Genética hasta el año 2006. Participa del proyecto PICTO 37029 financiado por la ANPCyT, habiendo participado además del proyecto financiado por la ANPCyT (PICT 05-15058). Realiza tareas de extensión y servicio en el Laboratorio de Biotecnología Molecular. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• María Angélica Lorenzati1 Es Médica y Especialista en Anatomía Patológica. Actualmente es Jefe de Trabajos Prácticos Regular de la Cátedra de Fisiopatología de la Carrera de Bioquímica desde el año 1998. Se desempeña como Investigador del Proyecto PICTO 37029 financiado por la ANPCyT, habiendo participado además del proyecto financiado por la ANPCyT (PICT 05-15058). Realiza tareas de extensión y servicio en el Laboratorio de Biotecnología Molecular. Posee publicaciones en revistas indexadas y presentaciones a congreso.
• Pedro Darío Zapata1 Es Doctor por la Universidad de Alcalá de Henares. Se desempeña actualmente como Profesor Regular Adjunto en las Cátedras de Biología Celular y Molecular (Bioquímica), Genética Molecular (Lic. en Genética), Biología Celular (Lic. en Genética) y Biotecnología Molecular (Ingeniería Química, Bioquímica y Farmacia). Posee actualmente la Categoría III en el Sistema Nacional de Incentivos a los Docentes-Investigadores. En el área de Biomedicina ha sido beneficiario de subsidios de la ANPCyT: PICT 05-15058 y PICTO 37029. Dirige auxiliares, becarios de grado y posgrado. Posee publicaciones en revistas indexadas nacionales e internacionales y presentaciones a congresos nacionales e internacionales.
Resumen
La epidemiología molecular estudia variaciones genotípicas relacionándolas con diversos procesos biológicos, entre ellos los patológicos como el cáncer. Por ello el análisis de muestras clínicas parafinadas es de gran importancia para el diagnóstico, la prevención y la terapia. Se ha informado con anterioridad muchos problemas relacionados con la amplificación de DNA obtenido a partir tejido embebido en parafina (TEP).
En este trabajo se comparó la capacidad de 9 metodologías para obtener DNA a partir de TEP y amplificarlo por PCR para analizar fragmentos de menos de 250 pb correspondientes con polimorfismos presentes en enzimas pertenecientes a la vía del folato.
El método modificado de Salting-out con NaCl de 0,5 M, CTAB 2 % como detergente y Proteinasa K fue el más adecuado para obtener DNA amplificable considerando los amplicones elegidos. Todos los métodos mostraron una mejor amplificación con ciclos más prolongados y temperaturas de annealing menores comparado con DNA más íntegro.
Palabras clave: Muestras parafinadas; Extracción de DNA.
Abstract
Molecular Epidemiology includes genotypic studies of different biological processes, including pathological diseases such as cancer. Therefore, the paraffin-embedded clinical samples analysis is very important for diagnosis, prevention and therapy. Previously, many problems associated with amplification of DNA obtained from paraffin-embedded tissue samples were reported.
This paper compares the capability of nine methodologies for DNA extraction from paraffin-embedded clinical samples, amplifying it by PCR to analyze under-250-bp fragments corresponding to folate pathway enzymes polymorphisms. Modification of "salting-out" method with concentrations of 0.5 M NaCl and 2 % CTAB detergent and Proteinase K was the most suitable for amplifiable DNA obtention of selected amplicons. All methods showed better amplification with longer cycles and lower annealing temperatures as compared to less fragmented DNA samples.
Key words: Paraffin-embedded samples; DNA extraction.
Introducción
Actualmente, los estudios de epidemiología molecular, de gran potencial como herramienta de diagnóstico y pronóstico, utilizan métodos basados en la amplificación de DNA por reacción en cadena de la polimerasa (PCR). Sin embargo, el procedimiento de extracción de DNA a partir de muestras biológicas tiene un impacto significativo sobre la sensibilidad y reproducibilidad de esta prueba molecular. Por ello es de vital importancia desarrollar un método de extracción de DNA que permita obtener genoma amplificable con un óptimo rendimiento y bajo costo, aplicándo la metodología en modelos experimentales, para luego proceder a realizarlos sobre las muestras clínicas [1]. Los tejidos fijados en formalina y embebidos en parafina (FFPE) presentan una extraordinaria fuente de material biológico y morfológico conservado y archivado de enfermedades específicas, que permiten la correlación de los resultados moleculares, la terapia y la clínica [2]. Los problemas de las amplificaciones cuando se trabaja con DNA extraído a partir de FFPE y los intervalos de fijación se asociaron con una disminución de los rendimientos y de la incapacidad de la PCR para amplificar fragmentos de DNA de gran tamaño [3]. La cuantificación fiable de la expresión de genes en tejidos parafinados ha sido objeto de serias limitaciones hasta el momento, aunque estudios anteriores han demostrado que los ácidos nucleicos pueden ser extraídos desde este material. Algunos autores han demostrado que tanto el DNA como el RNA de tejidos fijados en parafina puede ser extraído a pesar de someterse a importante degradación enzimática, debido a las diferencias en el tiempo de fijación en formalina después de la extirpación quirúrgica o por influencias de la variable durante la transformación de los tejidos hasta completar la fijación y ser embebido para archivar. [4]
En este trabajo se realizaron estudios que permitan estandarizar las condiciones para el procesamiento y amplificación de muestras clínicas tumorales parafinadas, para posteriormente iniciar los estudios de asociación entre polimorfismos y procesos patológicos.
Materiales y métodos Muestras utilizadas
En este trabajo se utilizaron 24 cortes parafinados: 11 de próstata, 4 de mama, 2 de ganglios linfáticos, 1 de útero, 1 de testículo, 1 de esófago, 1 de colon, 1 de riñón, 1 de tumor epitelial indefinido y 1 de epitelio de labio. Como controles de amplificación se utilizaron 3 muestras de tejido fresco de próstata. Los cortes de tejidos embebidos en parafina y de tejido fresco fueron cedidos por el Servicio de Anotomopatología del Hospital Ramón Madariaga (Pdas-Mnes-Argentina) y por el Servicio de Anatomía Patológica del Sanatorio Boratti. El tiempo de fijación previo de las muestras fue de 72 hs para luego ser parafinadas y almacenadas durante un tiempo variable (promedio 2 meses). De cada muestra parafinada se utilizaron 3 o 4 cortes de 3 urn (aproximadamente 25 mg) por cada microtubo.
También se tomaron 2 muestras de sangre entera como controles. Las muestras de sangre fueron obtenidas por punción venosa del personal del Laboratorio de Biotecnología Molecular de la Facultad de Ciencias Exactas, Químicas y Naturales de la Universidad Nacional de Misiones (FCEQyN - UNaM).
Todas las muestras fueron clasificadas con un código para preservar la identidad del paciente y la información obtenida fue confidencial siguiendo las reglamentaciones éticas, legales y jurídicas establecidas en las normas bioéticas nacionales -Disposición ANMAT 5330/97- e internacionales -Código de Nüremberg, Declaración de Helsinski y sus modificaciones; así como también la Declaración Universal sobre Genoma Humano y Derechos Humanos aprobada por la Conferencia General de la UNESCO, del 11/11/1997.
Extracción de DNA de muestras no parafinadas
Extracción de DNA a partir de sangre: Se utilizó una modificación del protocolo de Miller y Dykes, 1988 [2]. Primeramente, se realizaron lavados sucesivos con buffer de lisis de glóbulos rojos (Tris-HCl 10 mM pH 8, Tritón X 100 1 %, sacarosa 11 %). Luego, se digirieron los leucocitos 1 h a 65°C con una solución de Tris-HCl 10 mM, NaCl 400 mM, EDTA 2 mM, Proteinasa K 0,1 mg/ ml (Fermentas, Life Sciences) y SDS 2 %. Las proteínas se eliminaron por precipitación salina con acetato de potasio 3 M y el DNA se precipitó con isopropanol y etanol 70 %. El DNA se conservó a -20 °C en agua destilada libre de DNasas hasta su utilización.
Extracción de DNA a partir de tejido fresco: El tejido fue digerido a 65 °C por 16 h con una solución de TEC/ SDS (Tris-HCl 10 mM pH 8, EDTA 10 mM, NaCl 100 mM, SDS 2 %) y proteinasa K 0,1 mg/ml. La purificación se realizó con cloroformo:alcohol isoamílico (24:1) y acetato de potasio 3 M. La precipitación se realizó con isopropanol y etanol 70 %. El DNA se conservó a -20 °C en agua destilada libre de DNasas hasta su utilización.
Extracción de DNA de cortes de tejido embebidos en parafina
Modificación de la técnica de Salting-Out: Previamente, los tejidos fueron desparafinados a través de inmersión en xileno a 55 °C, etanol absoluto, etanol 95 %, etanol 70 % y agua destilada hasta observar un tejido transparente. [5]
La extracción de DNA se realizó a través de la técnica de Salting-Out [2], sobre la que se realizaron algunas modificaciones consistentes en la combinación de diferentes detergentes para la digestión enzimática (Tabla 1). La purificación se realizó con cloroformo: isoamílico (24:1) y acetato de potasio 3 M. Finalmente, la precipitación se realizó con isopropanol, dejándose entre 24 y 48 horas a -20 °C. El pellet se centrifugó, se lavó con etanol 70 %, se resuspendió en agua libre de nucleasas y se conservó a -20 °C hasta su utilización.
Tabla 1. Tratamientos utilizados para la extracción de DNA a partir los tejidos embebidos en parafina utilizando modificaciones de la técnica de Salting-Out.
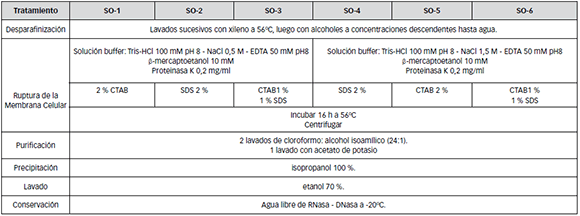
Además, se utilizó una modificación del tratamiento SO-1 intercambiando un lavado de cloroformo: alcohol isoamílico (24:1) por uno con fenol.
Técnica de extracción de DNA con el kit comercial Jet-Quick (Epicentre): Los tejidos se desparafinaron como se describió en la técnica anterior y luego se siguieron las indicaciones del fabricante. El DNA se resuspendió en agua libre de nucleasas y se conservó a -20 °C hasta su utilización.
Técnica de extracción de DNA utilizando resina Chelex-100: Los tejidos se desparafinaron en baño caliente a 90 °C con 5 % de Tween 20, luego se realizó una digestión con proteinasa K 0,125 mg/ml por 3 h a 55 °C. La purificación se realizó con Chelex 100 (BioRad) 5 % en Tris-HCl 10 mM pH 8, EDTA 1 mM a 99 °C. Posteriormente se centrifugó a 5.000 xg, se colocó a 0 °C, se transpasó a otro microtubo y se realizó una purificación adicional con cloroformo: alcohol isoamílico (24:1). El DNA se precipitó con isopropanol durante 24 h, se lavó con etanol 70 %, se resuspendió en agua libre de nucleasas y se conservó a -20 °C hasta su utilización. [6]
Evaluación de la calidad y cantidad del DNA obtenido
La calidad del DNA se evaluó por observación en geles de agarosa al 1 % comparándolo con DNA de referencia obtenido a partir de sangre entera y de tejido fresco. Todos los DNA obtenidos fueron cuantificados en espectrofotómetro a 260 nm (Shimadzu - UV 160 1 PC UV Visible).
Análisis de polimorfismos
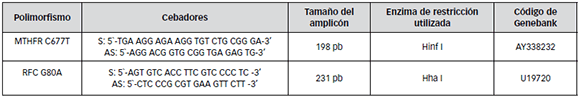
Para el análisis del polimorfismo C677T de la MTHFR y A80G del RFC se aplicó la técnica de PCR-RFLP (amplificación seguida de digestión con enzimas de restricción), realizándose una digestión de los productos de PCR con las enzimas de restricción HinfI y HhaI (Fermentas) respectivamente. [7, 8]
Los cebadores, previamente descriptos por la bibliografía y resumidos en la Tabla 2, fueron analizados utilizando las secuencias génicas disponibles en el NCBI (National Center for Biotechnology Information) y el programa Primer 3 (Howard Hughes Medical Institute and National Institutes of Health).
Tabla 2. Fragmentos amplificados para el análisis de polimorfismos.
Las amplificaciones se realizaron en un volumen de 20 ni conteniendo tampón PCR (200 mM (NH4)2SO4, 750 mM Tris-HCl pH 8,8, 0,1 % Tween 20), 200 uM de dNTPs, 1,5 mM de MgCl2, 10 pmol por reacción de cada cebador, 0,5 U de Taq polimerasa y 10 ng de DNA. El ciclado aplicado consistió en una etapa inicial de 3 min a 94 °C, seguidos de 40 ciclos de 94 °C por 20 s, 52 °C o 56 °C por 20 s dependiendo del par de cebadores y 72 °C por 20 s. Luego se modificaron las temperaturas de annealing y los segmentos se extendieron a 40 s.
Los fragmentos amplificados y digeridos fueron separados mediante electroforesis en geles de agarosa al 2 % y poliacrilamida al 10 %. Los geles de agarosa fueron corridos a 120 V por 20 min, se tiñeron con bromuro de etidio y se fotografiados con cámara digital Acer CS-5531. Los geles de poliacrilamida fueron corridos a 150 V durante 45 min, fijados 20 min en etanol 10 % - ácido acético 0,75 %, teñidos con nitrato de plata y escaneados con HP Deskjet F380.
Resultados y Discusión
La Figura 1 muestra los resultados obtenidos al extraer DNA con los diferentes métodos descriptos en materiales y métodos.
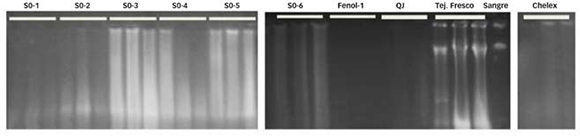
Figura 1. Calidad electroforética del DNA extraído. Se muestra el DNA extraído de muestras parafinadas con los diferentes tratamientos por triplicado. SO-1 al SO-6 coinciden con los tratamientos correspondientes al método de Salting-Out modificado (Tabla I). Las extracciones con el Kit Jet-Quick (Epicentre) se designan como QJ. El carril indicado como Fenol-1 corresponde a la modificación del tratamiento SO-1 en la que se agregó un lavado con fenol. El carril indicado como Chelex corresponde al tratamiento modificado de Coombs et al., 1999 [13]. Además, se observa el DNA obtenido a partir de tejido fresco y de sangre como controles de extracción. En cada carril se depositaron 2 de muestra.
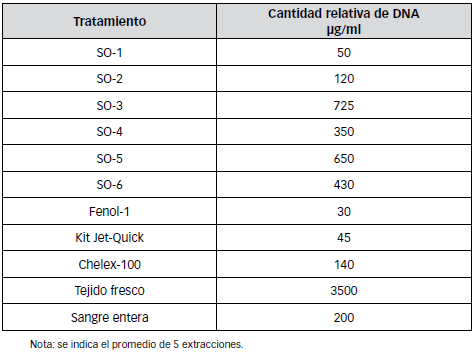
La Tabla 3 muestra los resultados de la cuantificación del DNA con cada método de extracción.
Tabla 3. Cuantificación del DNA extraído con cada método.
En la Tabla 4 se muestras resumen las amplificaciones logradas teniendo en cuenta los triplicados realizados para cada tratamiento evaluado.
Tabla 4. Amplificaciones logradas con cada tratamiento para los 2 polimorfismos seleccionados.
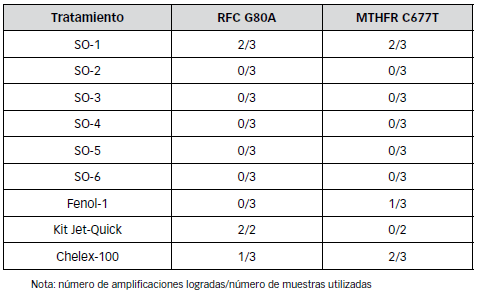
Todos los métodos de extracción muestran evidencias de degradación que influye notablemente en los resultados de la cuantificación, siendo recomendable en todos los casos la separación electroforética para la evaluación de la calidad. Los mejores valores de pureza y concentración se obtuvieron con el protocolo modificado de Salting-Out que corresponde al tratamiento SO-3, para la cual se calcula un rendimiento de 17,360 mg de DNA/ gr de tejido con una pureza de 1,72. Sin embargo, este tratamiento no permitió la amplificación de la muestras. Por otro lado, el protocolo SO-1 permitió la amplificación en el 66 % de las muestras a pesar de mostrar un bajo rendimiento en cantidad de DNA (Tabla 3). Esto puede deberse a que este tratamiento muestra el menor indicen de degradación al ser separado electroforéticamente (Figura 1).
Similar comportamiento se observa con el protocolo del Kit Jet-Quick y la resina Chelex-100 con los que se obtuvo un 50 % de amplificación.
El protocolo Fenol-1 también permitió la amplificación pero con un bajo rendimiento del 17 % de las muestras analizados.
En todos los casos el DNA extraído a partir de muestras parafinadas necesitó una menor temperatura de annealing (Tm) para ser amplificado y un aumento en los tiempos de cada segmento del ciclado.
Conclusiones
Los métodos de extracción más adecuado para amplificar DNA proveniente de muestras parafinadas fueron los que utilizan protocolo de Salting-out modificado por el agregado de NaCl 0,5 M; CTAB 2 % como detergente y Proteinasa K. Todos los métodos mostraron un mejor rendimiento con ciclos de amplificación más prolongados y temperaturas de annealing menores a las calculadas de manera teórica.
Referencias bibliográficas
1. Akalu A. y Reichardt J.K.V. Increasing PCR Sensitivity for Amplification from Paraffin-Embedded Tisúes. En: Chen, B.-Y. y Janes, H. W. (Ed.). New Jersey: Rutgers University. Methods in Molecular Biology. PCR Cloning Protocols, p. 67 - 74. 2005. [ Links ]
2. Miller S.A., Dykes D.D. y Polesky H.F. A simple salting out procedure for extracting DNA from human nucleated cells. 16(3): p.1215. 1988. [ Links ]
3. Specht K., Richter T., Müller U, Walch A., Werner M. y Hofler H. Quantitative Gene Expression Analysis in Microdissected Archival Formalin-Fixed and paraffin- Embedded Tumor Tissue. American Journal of Pathology, 158 (2): p. 419 - 429. 2001. [ Links ]
4. Gilbert M.T.P., Haselkorn T., Bunce M., Sanchez J.J., Lucas S.B., Jewells L.D., Van Marck E. y Worobey M. The Isolation of Nucleic Acids from Fixed, Paraffin-Embedded Tissues-Which Methods Are Useful When? PLOS ONE, 2(6): p. e537. 2007. [ Links ]
5. Nascimento E., Spinelli M., Rodrigues C. y Bozzini N. Protocolo da extragao de DNA de material parafinado para analise de microssatelites em leiomioma. Journal Bras Patol Med Lab, 39(3): p. 253-255. 2002. [ Links ]
6. Coombs N.J., Gough, A.C. y Primrose J.N. Optimisation of DNA and RNA extraction from archival formalin-fixed tissue. Nucleic Acids Research, 27: p. i-iii. 1999. [ Links ]
7. Singal R., Ferdinand L., Das P.M., Reis L.M. y Schlesselman J.J. Polymorphisms in the methylenetetrahydrofolate reductase gene and prostate cancer risk. International Journal of Oncology, 25: p. 1465-1471. 2004. [ Links ]
8. Relton C.L., Wilding C.S., Pearce M.S., Laffling A.J., Jonas P.A., Lynch S.A., Tawn E.J. y Burn J. Gene-gene interaction in folate-related genes and risk of neural tube defects in a UK population. J. Med. Genet., 41: p. 256-260. 2004. [ Links ]
Recibido: 30/05/10.
Aprobado: 21/09/10.